沙门氏菌(Salmonella)是一种来自肠杆菌科的革兰氏阴性杆状细菌,广泛栖息于家畜、家禽等温血动物的肠道中,可以通过粪便污染持续存在于食物和水等环境中,这些特点使其成为一种重要的人畜共患病原体[1]。目前已经鉴定出超过2 500 种沙门氏菌的血清型,其中1 500多种对人类致病[2]。据估计,70%~80%的食源性疾病由沙门氏菌污染造成,每年由沙门氏菌所导致的胃肠炎病例约为9 000万 例,死亡人数约为15万 人[3]。
动物源性食品被认为是沙门氏菌爆发的主要原因:一方面,肉制品中存在丰富的营养物质和微量元素,为沙门氏菌提供了良好的生长介质;另一方面,由于屠宰加工过程中的工序极易导致存在于动物消化道内的沙门氏菌通过粪便和破裂的消化道内容物污染胴体[4-5]。牛肉作为我国第3大消费肉类,仅次于猪肉和鸡肉,过去10 年牛肉消费量增加了50%[6]。牛肉在屠宰、加工、分销和销售过程中可能会受到食源性病原体的污染,尤其在屠宰加工过程中,动物和皮毛通常被认为是胴体污染的主要原因,然而,刀具、设备、环境及操作人员的处理也为沙门氏菌的交叉污染带来了可能性[7-9]。尽管已经实施了各种策略来控制牛肉生产各个阶段(即养殖、屠宰、分割和运输)的污染,但牛肉仍然是沙门氏菌爆发的重要来源[10]。Ren Xiang等[11]在2021年对我国15 个省份、779 份生牛肉的沙门氏菌流行情况进行调查,共有32 份样品检出沙门氏菌,流行率为4.11%。Xu Ziheng等[12]发现,2009—2016年我国南方零售牛肉沙门氏菌的流行率为11.79%(94/797)。可以看出,牛肉中沙门氏菌的流行率在不同的空间和时间中存在着一定的差异。同时,沙门氏菌近年来耐药性增加,在感染的过程中耐药性可以通过多重途径传播给人类,导致利用抗生素治疗细菌疾病收效甚微,严重威胁着人类的生命健康[13]。目前国内外针对动物源沙门氏菌的研究大多数集中在猪肉与禽类,关于牛源沙门氏菌的相关研究相对较少,而聚焦于肉牛屠宰过程中沙门氏菌流行特点的研究则更少[14-15]。因此,有必要对牛肉生产过程中沙门氏菌的流行率进行调研,明确屠宰加工过程中沙门氏菌流行特点,并借助分子分型技术对牛肉屠宰过程中沙门氏菌交叉污染进行分析,对于完善牛肉沙门氏菌生物信息数据库具有重要意义,为牛肉行业沙门氏菌的有效预防和控制措施提供数据支撑。
1 材料与方法
1.1 材料与试剂
鼠伤寒沙门氏菌(Salmonella Typhimurium)ATCC 14028由山东农业大学食品科学与工程学院动物源性食品研究室保存;其余47 株沙门氏菌菌株均来自于本次调研中从肉牛屠宰加工过程样品中分离纯化得到的菌株。
沙门氏菌显色培养基 法国CHROMager公司;亚硫酸铋琼脂、胰蛋白胨大豆肉汤(tryptone soy broth,TSB)、四硫磺酸钠煌绿增菌液(tatrathionate broth,TTB)、脑心浸出液肉汤(brain heart infusion broth,BHI)、半固体琼脂、营养琼脂、Mueller-Hinton肉汤(Mueller-Hinton broth,MHB)、Mueller-Hinton琼脂(Mueller-Hinton agar,MHA)、胰蛋白胨大豆琼脂、三糖铁琼脂、沙门氏菌干制生化鉴定试剂盒 北京陆桥技术股份有限公司;抗生素药敏纸片(头孢曲松、加替沙星、多黏菌素B、庆大霉素、链霉素、磺胺异噁唑、头孢噻吩、环丙沙星、头孢他啶、四环素、氟苯尼考、氨苄西林、红霉素、阿莫西林/克拉维酸) 杭州天和微生物试剂有限公司;细菌基因组DNA提取试剂盒、DNA Loading buffer、ddH2O、Taq Master Mix、Marker DM2000、Marker DM1000 康为世纪生物科技股份有限公司;Gold View I型核酸染料、5×Tris-硼酸-乙二胺四乙酸缓冲液 北京索莱宝科技有限公司;琼脂糖(Regular Agarose G-10) 法国Biowest公司;沙门氏菌属诊断血清(60 种) 宁波天润生物技术有限公司。
1.2 仪器与设备
BagMixer® 400均质器 法国Interscience公司;1300 SERIES A2生物安全柜 美国Thermo Scientific公司;5804R离心机 德国Eppendorf公司;G154DWS灭菌锅 致微(厦门)仪器有限公司;DHP系列生化培养箱 上海一恒科学仪器有限公司;EPS-301电泳仪英国Amersham公司;ChemiDoc MP凝胶成像仪 美国Bio-Rad公司。
1.3 方法
1.3.1 样品采集
2021年4—11月期间,于山东省的2 个省级重点龙头肉牛企业(A、B工厂分别位于滨州市阳信县与菏泽市曹县,均为危害分析及关键控制点认证企业)共采集样品435 份,A、B工厂的日屠宰量均为60~80 头。针对工厂屠宰流水线的工序,共设计9 个采样点,分别为:1)待宰圈,对肉牛屠宰前的待宰圈进行样品擦拭与采集;2)皮毛,对去皮前胴体的皮毛进行擦拭取样;3)去皮后胴体,对去除皮毛后的胴体样品进行擦拭取样;4)喷淋后胴体,经喷淋后的胴体立即进行擦拭取样;5)冷却后胴体,对冷却间悬挂冷却24 h后的胴体进行擦拭取样;6)分割肉样,在分割生产线上随机采集牛肉样品;7)粪便,收集靠近肛门段直肠中的粪便样品;8)刀具,随机选取分割生产线上的刀具进行擦拭采集;9)喷淋水,使用10 mL无菌离心管收集喷淋环节的水样。由于B工厂未设置喷淋环节,所以未采集喷淋后胴体和喷淋水样。
对采样点1~5、8,参照Breum等[16]描述的纱布擦拭法进行样品采集,即将20 cm×20 cm的无菌纱布用无菌缓冲蛋白胨水浸润后,随机擦拭牛二分体的胴体,每个二分体的擦拭面积约为0.25 m2,擦拭完成后,将纱布放入均质袋中。对于分割肉样品(采样点6),在肉牛分割过程中随机挑取25 g的分割肉样,每块肉样单独装入均质袋中带回。对于粪便样品(采样点7),按照Madden等[17]描述的方法,由工厂操作人员使用无菌刀具将直肠剪开,在靠近肛门处使用无菌离心管收集约10 g直肠粪便。对于喷淋水样(采样点9),使用无菌离心管进行盛放。所有样品采样后4 h内在4 ℃下运送至实验室进行下一步实验。屠宰过程中各采样环节的设置及采样数量如表1所示。
表1 采样环节的设置及采样数量
Table 1 Sampling points and amounts

注:-.未采集样品。表4同。
工厂采样点合计待宰圈皮毛去皮后胴体喷淋后胴体冷却后胴体分割肉样粪便刀具喷淋水A252525252525252525225 B303030-30303030-210合计555555255555555525435
1.3.2 样品的前增菌
样品运回实验室后,将放有擦拭、粪便样品的每个均质袋中加入100 mL TSB;喷淋水样无菌量取10 mL于均质袋中,加入90 mL TSB;分割肉样无菌称取25 g于均质袋中,加入225 mL TSB,以8 000 r/min均质2 min。参照Barkocy-Gallagher等[18]的方法,将样品置于25 ℃培养箱中培养2~3 h,然后转移至42 ℃培养6 h,结束培养后置于4 ℃过夜;吸取均质袋中的1 mL增菌液于10 mL TTB中,并置于(42±1)℃培养18~24 h。
1.3.3 沙门氏菌的选择性培养与分离纯化
使用直径3 mm的接种环蘸取1 环增菌液,分别划线接种于亚硫酸铋琼脂平板和沙门氏菌显色培养基平板,亚硫酸铋琼脂平板于(36±1)℃培养40~48 h,沙门氏菌显色培养基平板于(36±1)℃培养15~24 h,观察各个平板上生长的菌落特征。
培养结束后,挑取平板上的可疑菌落,再次划线接种于营养琼脂平板进行纯化,每份样品挑取2~3 株纯化后的单菌落接种于BHI,37 ℃摇床培养18~24 h;将其置于体积分数25%无菌甘油冻存管中,-80 ℃冰箱保存备用。
1.3.4 沙门氏菌的聚合酶链式反应(polymerase chain reaction,PCR)鉴定与生化鉴定
参考细菌基因组DNA提取试剂盒说明书对可疑菌株的DNA进行提取,以提取的DNA作为模板,沙门氏菌标准菌株鼠伤寒沙门氏菌ATCC 14028为阳性对照,ddH2O为阴性对照,以特异性基因invA为引物(正向序列:TTTACGGTCTATTTTGATTTG;反向序列:ATATGCTCCACAAGGTTAATG),参照Arnold等[19]的方法进行PCR。引物序列由生工生物工程(上海)股份有限公司合成,含有invA基因的菌株为沙门氏菌阳性。取扩增后的PCR产物进行琼脂糖凝胶电泳,电泳条件为120 V、180 mA、35 min,使用凝胶成像仪观察结果。
同时对可疑菌株进行沙门氏菌生化鉴定,根据GB 4789.4—2016《食品微生物学检验 沙门氏菌检验》的要求,通过沙门氏菌干制生化鉴定试剂盒对菌株进行进一步鉴定。将冻存的可疑菌株活化后划线接种至营养琼脂平板,用接种环仔细挑选单菌落,随后将其置于适量的蛋白胨水中,形成细菌浊度为0.5 MCF的均匀菌悬液。另外用接种环挑选单菌落对三糖铁琼脂进行接种,先于表面进行接种,然后进行底层穿刺。用无菌胶头滴管将菌悬液滴入生化鉴定管中进行鉴定,每管接种量约1~2 滴。根据具体指标要求将菌株在37 ℃培养18~24 h,必要时培养时间延长至48 h,根据产品附带的说明书判定结果。最后,将鉴定出的菌株保存在-80 ℃、含有25%甘油的冻存管中。
1.3.5 沙门氏菌的血清型鉴定
参照GB 4789.4—2016,将保存的菌株经BHI活化后划线于营养琼脂平板上,37 ℃培养18~24 h,通过沙门氏菌属诊断血清(60 种)进行血清型鉴定,选择单菌落进行玻片凝集试验,通过多价菌体抗原(O)和多价鞭毛抗原(H)对菌株进行鉴定。根据凝集现象的结果,参照产品附带的说明书附录和沙门氏菌血清型抗原表,确定菌株的血清型。
1.3.6 沙门氏菌的毒力基因检测
参照Fardsanei等[20]的方法采用多重PCR技术对分离出的菌株进行毒力基因检测,毒力基因引物序列由生工生物工程(上海)股份有限公司合成,将扩增后的PCR产物进行琼脂糖凝胶电泳,使用凝胶成像仪观察结果。毒力基因引物序列见表2。
表2 沙门氏菌毒力基因引物序列
Table 2 Primer sequences of Salmonella virulence genes

毒力基因引物序列(5’-3’)片段大小/bp参考文献invAF:TTTACGGTCTATTTTGATTTG44354[19]R:ATATGCTCCACAAGGTTAATG rckF:AACGGACGGAACACAGAGTC18959[21]R:TGTCCTGACGAAAGTGCATC sipCF:AGACAGCTTCGCAATCCGTT44661[20]R:ATTCATCCCTTCGCGCATCA sopEF:CGAGTAAAGACCCCGCATAC36258[21]R:GAGTCGGCATAGCACACTCA hilAF:CGTGAAGGGATTATCGCAGT29656[22]R:GTCCGGGAATACATCTGAGC pefAF:TTGCACTGGGTGGTTCTGG48556[23]R:TGTAACCCACTGCGAAAG sefAF:GCAGCGGTTACTATTGCAGC32155[24]R:TGTGACAGGGACATTTAGCG sopBF:CCTCAAGACTCAAGATG1 98756[25]R:TACGCAGGAGTAAATCGGTG spvCF:ACTCCTTGCACAACCAAATGCGGA57156[26]R:TGTCTCTGCATTTCGCCACCATCA ssaRF:GTTCGGATTTGCTTCGG1 62859[27]R:TCTCCAGTGACTAACCCTAACCAA ssrAF:CTTACGATTACGCCATTTACGG70658[28]R:ATTTGGTGGAGCTGGCGGGACT stnP1F:TTGTCTCGCTATCACTGGCAACC61759[29]R:ATTCGTAACCCGCTCTCGTCC sipAF:CCATTCGACTAACAGCAGCA44956[22]R:CGGTCGTACCGGCTTTATTA退火温度/℃
1.3.7 沙门氏菌的抗生素耐药性测试
根据美国临床实验室标准化协会(Clinical Laboratory Standards Institute,CLSI)指南,采用纸片扩散法对分离菌株进行抗生素药敏试验[30]。利用抑菌圈直径确定抗生素的耐药性。首先,将冻存菌株接种于MHB中,于37 ℃培养18 h,同时使用无菌生理盐水调整菌悬液浓度,直至达到0.5 MCF,然后用无菌棉签蘸取菌液均匀涂布于MHA平板,将抗生素药片均匀贴在琼脂平板表面,确保之间有足够的距离,以防止抑菌圈重叠,经室温放置15 min后于37 ℃下培养48 h。沙门氏菌标准菌株鼠伤寒沙门氏菌ATCC 14028用作质控菌株。最后,使用游标卡尺测量抑制圈直径。CLSI指南针对不同抗生素抑菌圈直径范围将其分为耐药(R)、中介(I)和敏感(S),选取10大类、14 种抗生素进行试验,包括头孢曲松(30 µg)、加替沙星(5 μg)、多黏菌素B(10 µg)、庆大霉素(10 μg)、链霉素(10 μg)、磺胺异噁唑(300 µg)、头孢噻吩(30 μg)、环丙沙星(5 μg)、头孢他啶(30 μg)、四环素(30 μg)、氟苯尼考(30 µg)、氨苄西林(10 μg)、红霉素(15 µg)和阿莫西林/克拉维酸(20 μg),判定标准如表3所示。
表3 沙门氏菌药敏纸片判定标准
Table 3 Interpretive criterion for antibiotic susceptibility determination of Salmonella
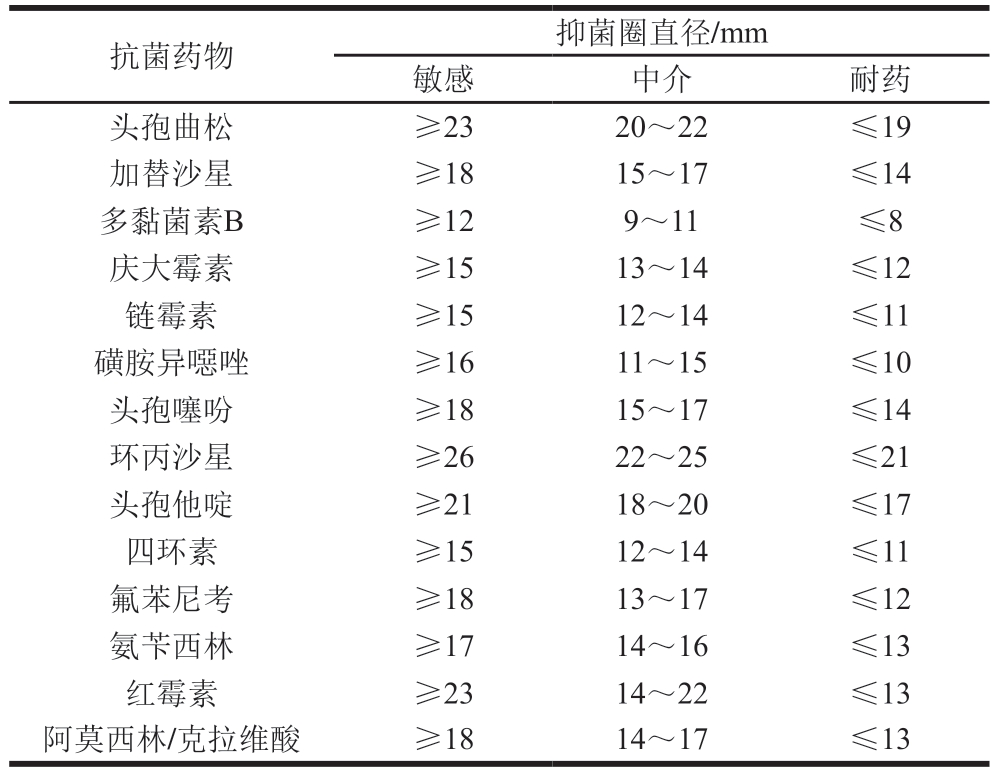
抗菌药物抑菌圈直径/mm敏感中介耐药头孢曲松≥2320~22≤19加替沙星≥1815~17≤14多黏菌素B≥129~11≤8庆大霉素≥1513~14≤12链霉素≥1512~14≤11磺胺异噁唑≥1611~15≤10头孢噻吩≥1815~17≤14环丙沙星≥2622~25≤21头孢他啶≥2118~20≤17四环素≥1512~14≤11氟苯尼考≥1813~17≤12氨苄西林≥1714~16≤13红霉素≥2314~22≤13阿莫西林/克拉维酸≥1814~17≤13
1.3.8 沙门氏菌的多位点序列分型(multilocus sequence typing,MLST)
首先对沙门氏菌的7 个管家基因(thrA、sucA、purE、hisD、hemD、dnaN、aroC)进行PCR扩增,将PCR产物送至生工生物工程(上海)股份有限公司进行双向测序,对等位基因序列进行拼接和校正后提交至PubMLST数据库(https://pubmlst.org/),与数据库中沙门氏菌相应管家基因的等位基因进行比对,每个管家基因都有一个特有的等位基因序号,7 个管家基因的等位基因序号组成该菌株的等位基因谱。将分离菌株的等位基因谱提交至数据库,确定分离菌株的序列型(sequence typing,ST)。将每个分离株的等位基因串联序列信息导入MEGA X软件,进行系统发育分析。
1.4 数据处理
通过阳性样本数与样本总数的比值计算流行率。通过SPSS软件对数据进行卡方检验分析,以确定取样环节流行率的显著差异,P<0.05时认为具有显著差异。
2 结果与分析
2.1 生鲜牛肉生产过程中沙门氏菌的流行率
在采集的435 份样品中,通过生化鉴定与PCR鉴定出18 份样品中显示沙门氏菌阳性,从18 份沙门氏菌阳性样品中共收集到47 个沙门氏菌分离株,总体流行率为4.1%(18/435)。其中A工厂并未检出沙门氏菌(0.0%;0/225),B工厂流行率为8.6%(18/210)(表4),这说明不同工厂沙门氏菌的污染情况有所差异。一方面可能是由于卫生环境的差异,在A工厂采集样品过程中,待宰圈气味良好,粪便带来的交叉污染概率较小;此外,与B工厂相比还设置了喷淋环节,对卫生环境的控制更为严格。B工厂中各环节样品均检出沙门氏菌,流行率最高的为皮毛(16.7%;5/30),其次为粪便(13.3%;4/30)和分割肉(10.0%;3/30)。就总体流行率而言,待宰圈、皮毛、去皮后胴体、冷却后胴体、分割肉、粪便、刀具流行率分别为3.6%、9.1%、3.6%、1.8%、5.5%、7.3%、1.8%,在喷淋后胴体和水样中未检出沙门氏菌,检出率最高的环节为皮毛(9.1%;5/55),其次为粪便(7.3%;4/55)。Dong Pengcheng等[31]在2010年对4 家牛肉加工厂沙门氏菌的流行情况进行调查,510 份样品共检出33 份沙门氏菌阳性样品,流行率为6.5%,同样,皮毛和粪便是流行率最高的环节。此外,有研究[32]对29 个国家牛源沙门氏菌进行荟萃分析,结果显示,沙门氏菌的总体流行率为9.0%(95%置信区间:7.0%~11.0%)。除上述外,在本研究中还可以观察到去皮前后胴体沙门氏菌流行率有明显的下降,此规律在许多研究中也同样存在,Zhang Haoqi等[33]对猪肉屠宰加工过程沙门氏菌进行调研,去皮前后的流行率从34.0%降低到10.0%。综上所述,牛的皮毛及皮毛上的污染物是造成沙门氏菌污染的主要来源[34]。
表4 肉牛屠宰工厂不同工序中沙门氏菌的流行情况
Table 4 Prevalence of Salmonella in different processes in beef slaughter plants
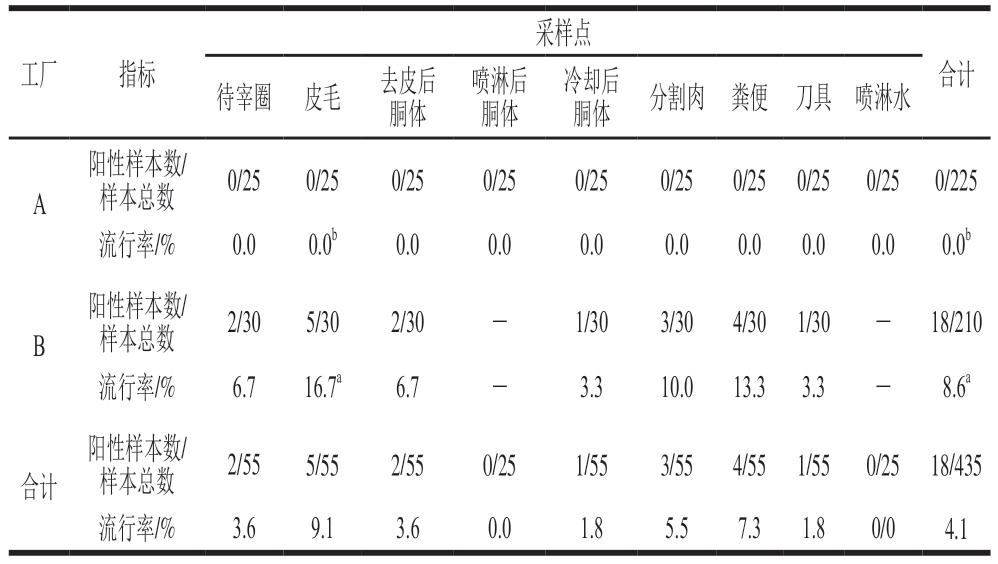
注:同列小写字母不同表示差异显著(P<0.05)。表5同。
采样点工厂指标合计待宰圈皮毛去皮后胴体喷淋后胴体冷却后胴体分割肉粪便刀具喷淋水A阳性样本数/样本总数0/250/250/250/250/250/250/250/250/250/225流行率/%0.00.0b0.00.00.00.00.00.00.00.0b B阳性样本数/样本总数2/305/302/30-1/303/304/301/30-18/210流行率/%6.716.7a6.7-3.310.013.33.3-8.6a合计阳性样本数/样本总数2/555/552/550/251/553/554/551/550/2518/435流行率/%3.69.13.60.01.85.57.31.80/04.1
B工厂中并未设置喷淋环节,但研究[35]表明,喷淋是降低胴体表面致病菌水平的有效手段,然而此过程中也有概率会出现喷淋液体飞溅导致交叉污染的情况。在分割环节,沙门氏菌的污染率大幅提升,可能是由于在胴体分割时,操作人员使用的刀具、传送带及桌面等均会成为重要的污染源[36]。食品加工表面和设备是微生物定植的主要场所,操作人员(如工作服)和使用的工具(如刀具)极易使胴体受到交叉污染,并且如果清洁不当,这些食品加工表面微生物存活的可能性会更高,同时对于肉制品来说,加工表面存在肉汁(渗出物)为微生物生长提供了有利的营养环境[37-38]。
2.2 生鲜牛肉生产过程中沙门氏菌的血清型分型
对工厂样品中分离的47 株沙门氏菌通过玻片凝集法进行血清型分型,如表5所示,共分为2 种血清型,其中25 株(53.2%;25/47)为鼠伤寒沙门氏菌,22 株(46.8%;22/47)为乙型副伤寒沙门氏菌(Salmonella Paratyphi B)。Kuus等[39]指出,在爱沙尼亚地区,人类感染沙门氏菌最普遍的血清型是肠炎沙门氏菌(Salmonella Enteritidis)、其次是鼠伤寒沙门氏菌、单相鼠伤寒沙门氏菌(monophasic Salmonella Typhimurium)和婴儿沙门氏菌(Salmonella Infantis),该研究对2016—2020年爱沙尼亚肉类生产链中沙门氏菌血清型进行分析,结果发现,在屠宰厂分离的血清型主要是德尔卑沙门氏菌(Salmonella Derby)、鼠伤寒沙门氏菌和单相鼠伤寒沙门氏菌,并未发现肠炎沙门氏菌。而德尔卑沙门氏菌是猪肉中最常检测到的沙门氏菌血清型[40]。在不同环节中,鼠伤寒沙门氏菌主要在皮毛(32.0%;8/25)、分割肉(56.0%;14/25)环节检出,乙型副伤寒沙门氏菌主要在粪便(54.5%;12/22)、刀具(31.8%;7/22)环节检出(表5)。喷淋后胴体、冷却后胴体、刀具、喷淋水4 个环节中未检出鼠伤寒沙门氏菌;而皮毛、喷淋后胴体、分割肉、喷淋水4 个环节中未检出乙型副伤寒沙门氏菌。Gutema等[32]进行的荟萃分析中表明,与牛相关的沙门氏菌血清型为蒙得维的亚沙门氏菌(Salmonella Montevideo)、鼠伤寒沙门氏菌、肯塔基沙门氏菌(Salmonella Kentucky),并且常见血清型在不同地区具有一定的差异,其中蒙得维的亚沙门氏菌是北美最常报告的血清型,非洲、亚洲和大洋洲最常见的血清型为鼠伤寒沙门氏菌,都柏林沙门氏菌是欧洲最常报告的血清型。在2011—2016年期间,Yang Xiaojuan等[41]调查了中国大部分省市的零售肉类,发现德尔卑沙门氏菌和鼠伤寒沙门氏菌为最普遍的血清型。综上所述,沙门氏菌血清型呈现多样性的特点,但在各国畜禽肉制品中还是以鼠伤寒沙门氏菌和肠炎沙门氏菌为主。
表5 肉牛屠宰工厂不同工序中沙门氏菌的血清型分布
Table 5 Distribution of Salmonella serotypes in different processes in beef slaughter plants
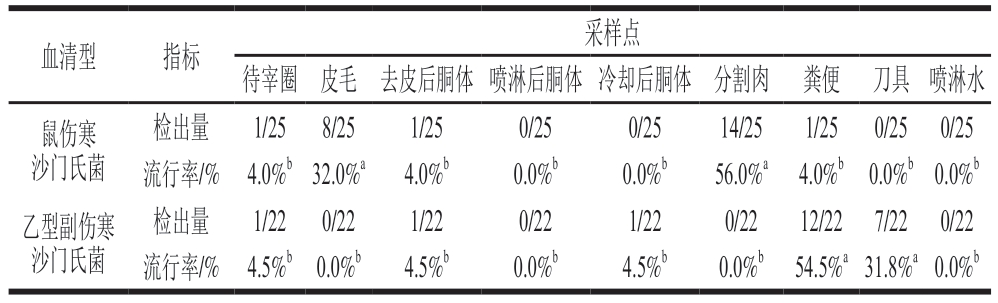
血清型指标采样点待宰圈皮毛去皮后胴体喷淋后胴体冷却后胴体分割肉粪便刀具喷淋水鼠伤寒沙门氏菌检出量1/258/251/250/250/2514/251/250/250/25流行率/%4.0%b32.0%a4.0%b0.0%b0.0%b56.0%a4.0%b0.0%b0.0%b乙型副伤寒沙门氏菌检出量1/220/221/220/221/220/2212/227/220/22流行率/%4.5%b0.0%b4.5%b0.0%b4.5%b0.0%b54.5%a31.8%a0.0%b
2.3 生鲜牛肉生产过程中沙门氏菌的毒力基因分布
对分离的47 株沙门氏菌进行13 种毒力基因检测,如图1所示,pefA、ssaR毒力基因未检出。invA、sipC、stnP1、sopB毒力基因检出率为100%,hilA、sipA检出率也较高,分别为95.74%(45/47)和93.62%(44/47)。沙门氏菌的致病潜力被认为与分离株中毒力基因的存在有关[42-43]。invA基因通常用作沙门氏菌鉴定的特异性标记,与其对上皮细胞的侵袭潜力密切相关。同样,sopB、ssrA、hilA、sipA基因在介导宿主细胞侵袭中发挥着至关重要的作用[33]。sipC基因不仅与沙门氏菌入侵相关,而且还是沙门氏菌感染炎症的关键调节因子,而ssaR基因也在全身感染中发挥着重要作用[44-45]。pefA基因与菌毛的功能相关[46]、stnP1作为肠毒素基因,可调节特定血清型沙门氏菌产生具有毒性的可溶性蛋白[47]。刘春红等[48]对鸡源山夫登堡沙门氏菌分离株进行毒力基因检测,与本研究结果类似,invA、hilA、sipC、ssrA、stnP1基因的检出率均为100%,未检出pefA基因,但sopB和ssaR基因的检出率与本研究有所差异,分别为50.0%和62.5%,说明不同地区、不同来源的沙门氏菌携带的毒力基因存在一定差异。
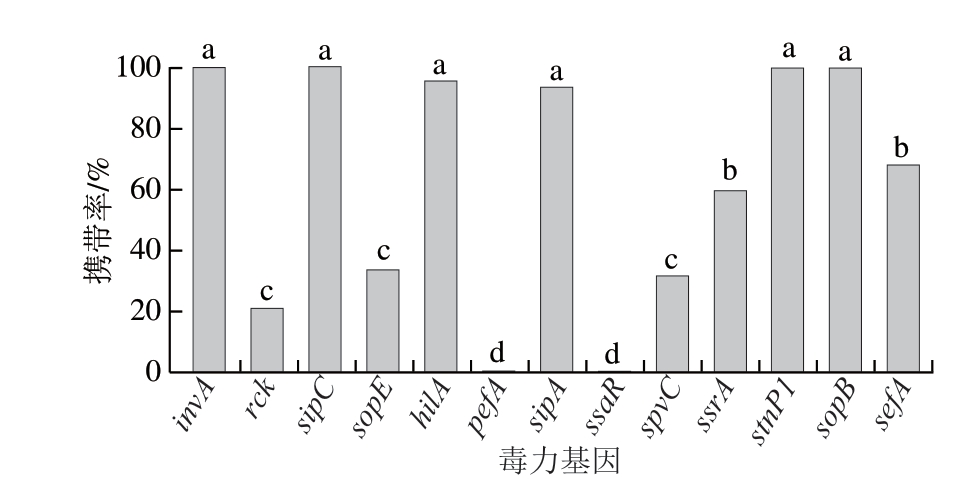
图1 肉牛屠宰工厂检出的沙门氏菌毒力基因携带率
Fig. 1 Prevalence of virulence genes of Salmonella in beef slaughter plants
小写字母不同表示差异显著(P<0.05)。
2.4 生鲜牛肉生产过程中沙门氏菌的抗生素耐药性
抗生素耐药性是沙门氏菌致病性不可忽视的指标,随着抗生素广泛且不合理的使用,使得沙门氏菌存在严重的耐药性,给临床治疗带来了一定的困难[49]。对3 种或更多抗生素表现出耐药性的菌株通常被称为多重耐药菌株[50]。如图2所示,红霉素的耐药率最强,为100%(47/47);其次为磺胺异噁唑(80.85%)和四环素(51.1%)。沙门氏菌对庆大霉素全部敏感(100%;47/47);对头孢他啶、头孢曲松、阿莫西林/克拉维酸、环丙沙星、加替沙星也表现出较高的敏感性,均在85%以上。与Zhang Lina等[51]关于广东地区零售肉的调查结果相似,该研究发现所有分离出的沙门氏菌菌株均对头孢他啶和阿莫西林/克拉维酸敏感。Tadesse[52]的荟萃分析结果显示,从埃塞俄比亚地区猪、牛、羊等动物中分离的沙门氏菌超过75%具有耐药性。近年来,由于抗生素的广泛和不合理使用以及耐药基因的水平转移极大加剧了抗生素耐药性负担的升级,导致沙门氏菌的耐药问题层出不穷,沙门氏菌的耐药性引起世界的广泛关注[53]。从纳入的14 种抗生素来看,肉牛屠宰过程中检出的沙门氏菌对绝大多数抗生素都产生了不同程度的耐药,应当引起重视。
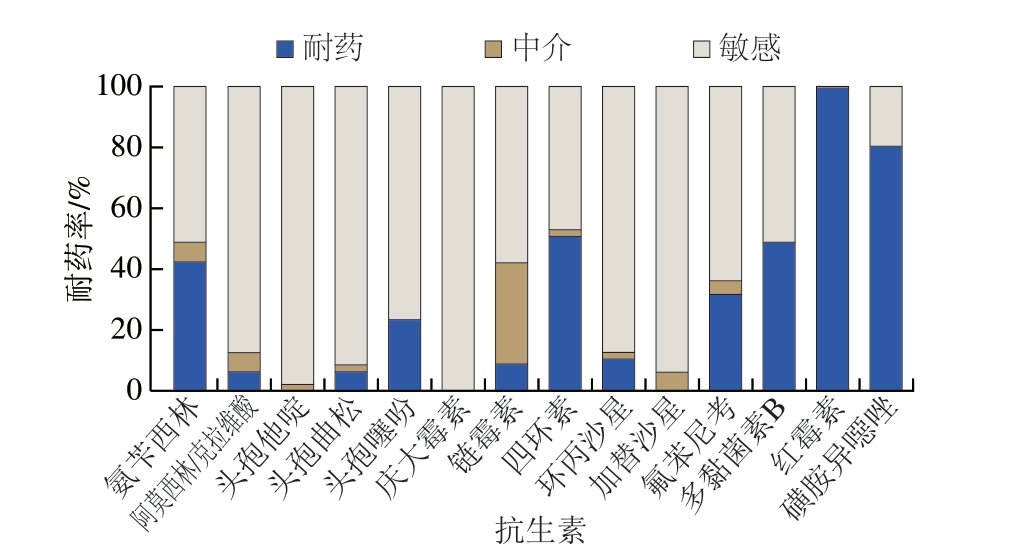
图2 肉牛屠宰工厂检出的沙门氏菌抗生素耐药率
Fig. 2 Antibiotic resistance rate of Salmonella in beef slaughter plants
如表6所示,47 株菌共产生17 种耐药谱,其中最常见的耐药谱为SIZ-FON-AM-EM(21.28%;10/47),其次为EM-SIZ(14.89%;7/47)、PB-SIZ-TET-EM(12.77%;6/47);有4 株菌对9 种抗生素耐药,耐药谱分别为PB-S-SIZ-CF-CIP-TET-FON-AM-EM(2.13%;1/47),CTR-PB-S-SIZ-CF-CIP-TET-AM-EM(6.38%;3/47)。从多重耐药性来看,本研究分离的菌株多重耐药情况较严重,多重耐药率为80.85%(38/47),在多重耐药菌株中,耐受4 种药物的比例最高,占多重耐药菌株的50.00%(19/38)。在2016年武汉市3 个猪屠宰场的沙门氏菌调查中,发现多重耐药率为75.7%(134/177)[54]。Xu Yaohui等[55]调查2014—2015年河南鸡场沙门氏菌的耐药情况,发现有69.64%的菌株产生了多重耐药性。抗生素的耐药性可以通过食物链等途径传递给人类,在2014—2017年广西腹泻患者沙门氏菌的调查中,发现46.6%的分离菌株具有多重耐药性[56]。因此,为了解决抗生素耐药性,需管理抗生素的使用,禁止其非治疗性应用,并定期评估细菌对常见抗生素的敏感性以及加强对多重耐药菌株的监测。
表6 肉牛屠宰工厂检出的沙门氏菌耐药谱
Table 6 Antibiotic resistance spectrum of Salmonella in beef slaughter plants
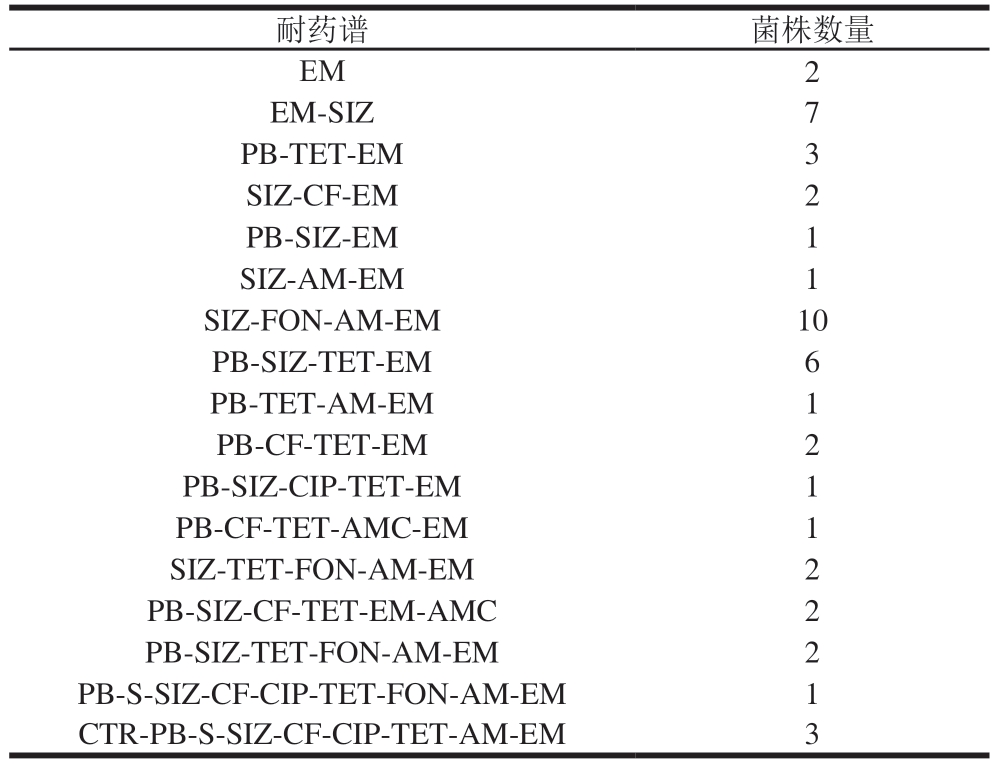
注:CTR.头孢曲松;GAT.加替沙星;PB.多黏菌素B;GM.庆大霉素;S.链霉素;SIZ.磺胺异噁唑;CF.头孢噻吩;CIP.环丙沙星;CAZ.头孢他啶;TET.四环素;FON.氟苯尼考;AM.氨苄西林;AMC.阿莫西林/克拉维酸;EM.红霉素。
耐药谱菌株数量EM2 EM-SIZ7 PB-TET-EM3 SIZ-CF-EM2 PB-SIZ-EM1 SIZ-AM-EM1 SIZ-FON-AM-EM10 PB-SIZ-TET-EM6 PB-TET-AM-EM1 PB-CF-TET-EM2 PB-SIZ-CIP-TET-EM1 PB-CF-TET-AMC-EM1 SIZ-TET-FON-AM-EM2 PB-SIZ-CF-TET-EM-AMC2 PB-SIZ-TET-FON-AM-EM2 PB-S-SIZ-CF-CIP-TET-FON-AM-EM1 CTR-PB-S-SIZ-CF-CIP-TET-AM-EM3
2.5 生鲜牛肉生产过程中沙门氏菌的溯源分析
如表7、图3所示,47 株沙门氏菌分为2 种ST,分别为ST19(53.2%;25/47)和ST42(46.8%;22/47)。待宰圈、皮毛、去皮后胴体、分割肉和粪便具有相同的基因型ST19,这说明待宰圈、皮毛和去皮后胴体中沙门氏菌的最初来源可能是粪便,造成污染的途径可能为粪便-待宰圈-皮毛-去皮后胴体-分割肉。通过比对,在本研究中ST19为鼠伤寒沙门氏菌,ST42为乙型副伤寒沙门氏菌。在肉类加工环境中,沙门氏菌多以ST19为主,并且其具有较高的致病力[57-59]。有研究[60]发现,很多ST与血清型呈现较强的相关性。而鼠伤寒沙门氏菌可以通过食物链和其他途径(与动物的接触)进行传播,对人类健康构成严重威胁[61]。Zhou Zihao等[62]对生猪屠宰场的调查中发现,ST40、ST34均从胴体、环境、肠道内容物和设备中检出,该研究推测屠宰场内发生了4 种污染源之间的交叉污染。ST42为乙型副伤寒沙门氏菌,主要从待宰圈、粪便和刀具中检出,未发现明确的污染来源,推测乙型副伤寒沙门氏菌的宿主以粪便和设备为主,因此,屠宰企业需在处理区域内执行卫生标准控制,并通过操作卫生培训增强员工能力。总体而言,MLST的结果表明,牛肉屠宰过程中屠宰链污染与沙门氏菌的传播高度相关,大多数ST都指向待宰圈和粪便的污染物来源,并且在生产链中极易发生交叉污染和连锁污染。应加强待宰圈的卫生措施,作为污染控制的主要点。
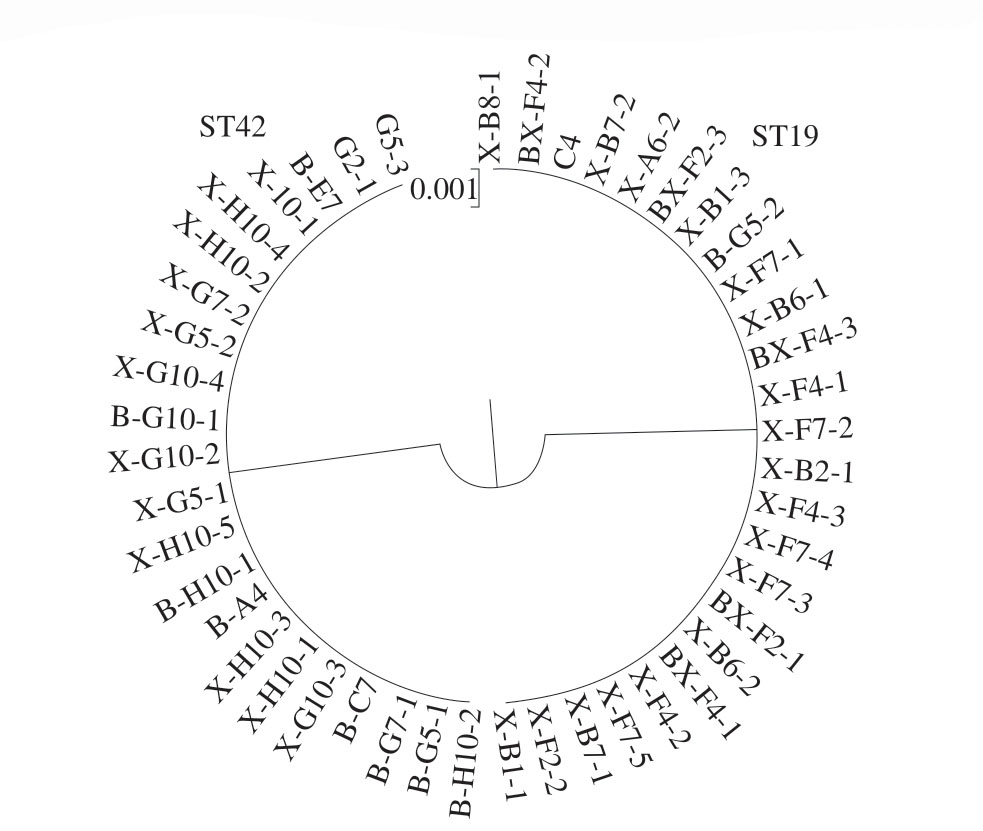
图3 肉牛屠宰工厂检出的沙门氏菌系统发育树
Fig. 3 Phylogenetic tree of Salmonella in beef slaughter plants
表7 肉牛屠宰工厂检出的沙门氏菌MLST结果
Table 7 MLST results of Salmonella isolates from beef slaughter plants
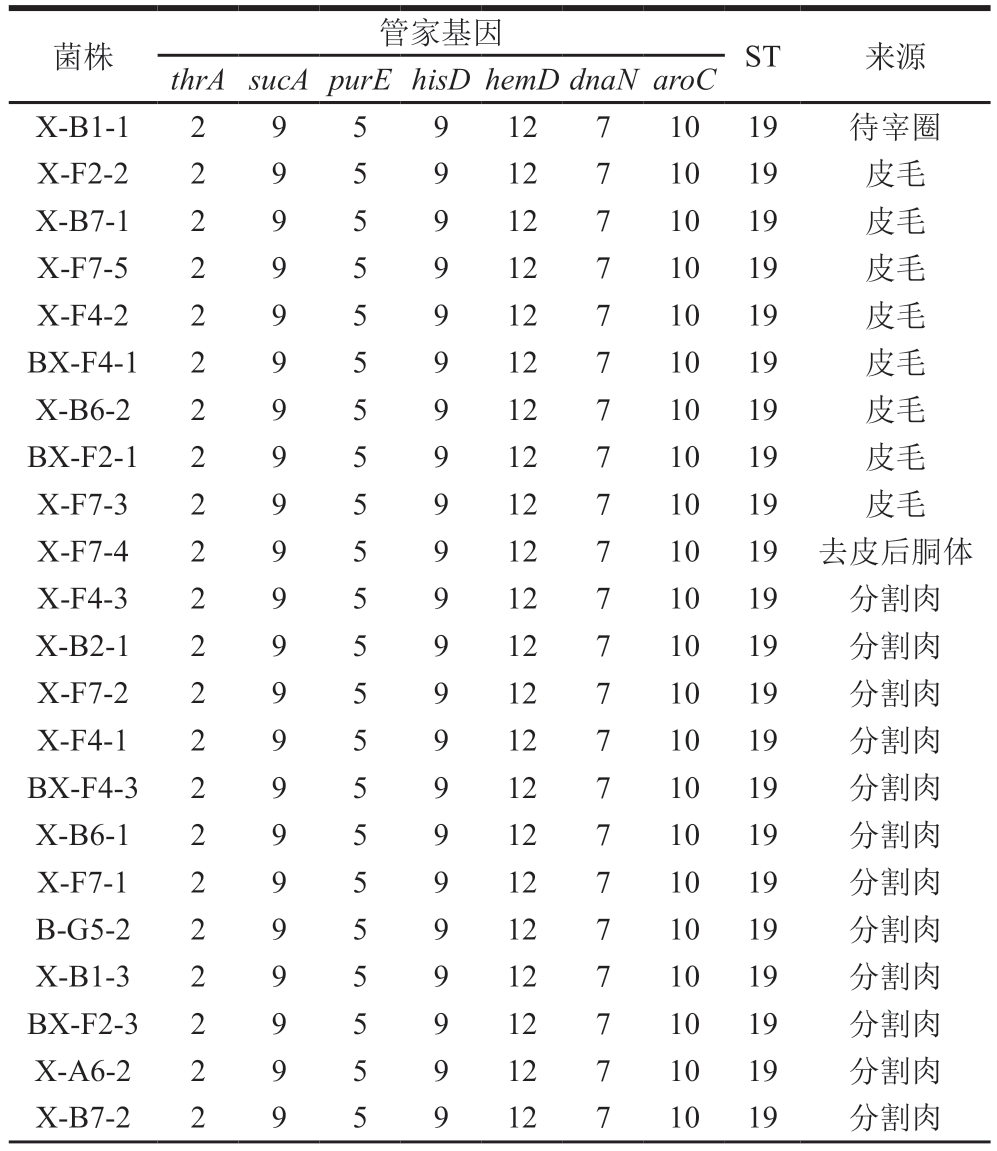
菌株管家基因ST来源thrAsucApurEhisDhemDdnaNaroC X-B1-129591271019待宰圈X-F2-229591271019皮毛X-B7-129591271019皮毛X-F7-529591271019皮毛X-F4-229591271019皮毛BX-F4-129591271019皮毛X-B6-229591271019皮毛BX-F2-129591271019皮毛X-F7-329591271019皮毛X-F7-429591271019去皮后胴体X-F4-329591271019分割肉X-B2-129591271019分割肉X-F7-229591271019分割肉X-F4-129591271019分割肉BX-F4-329591271019分割肉X-B6-129591271019分割肉X-F7-129591271019分割肉B-G5-229591271019分割肉X-B1-329591271019分割肉BX-F2-329591271019分割肉X-A6-229591271019分割肉X-B7-229591271019分割肉
续表7
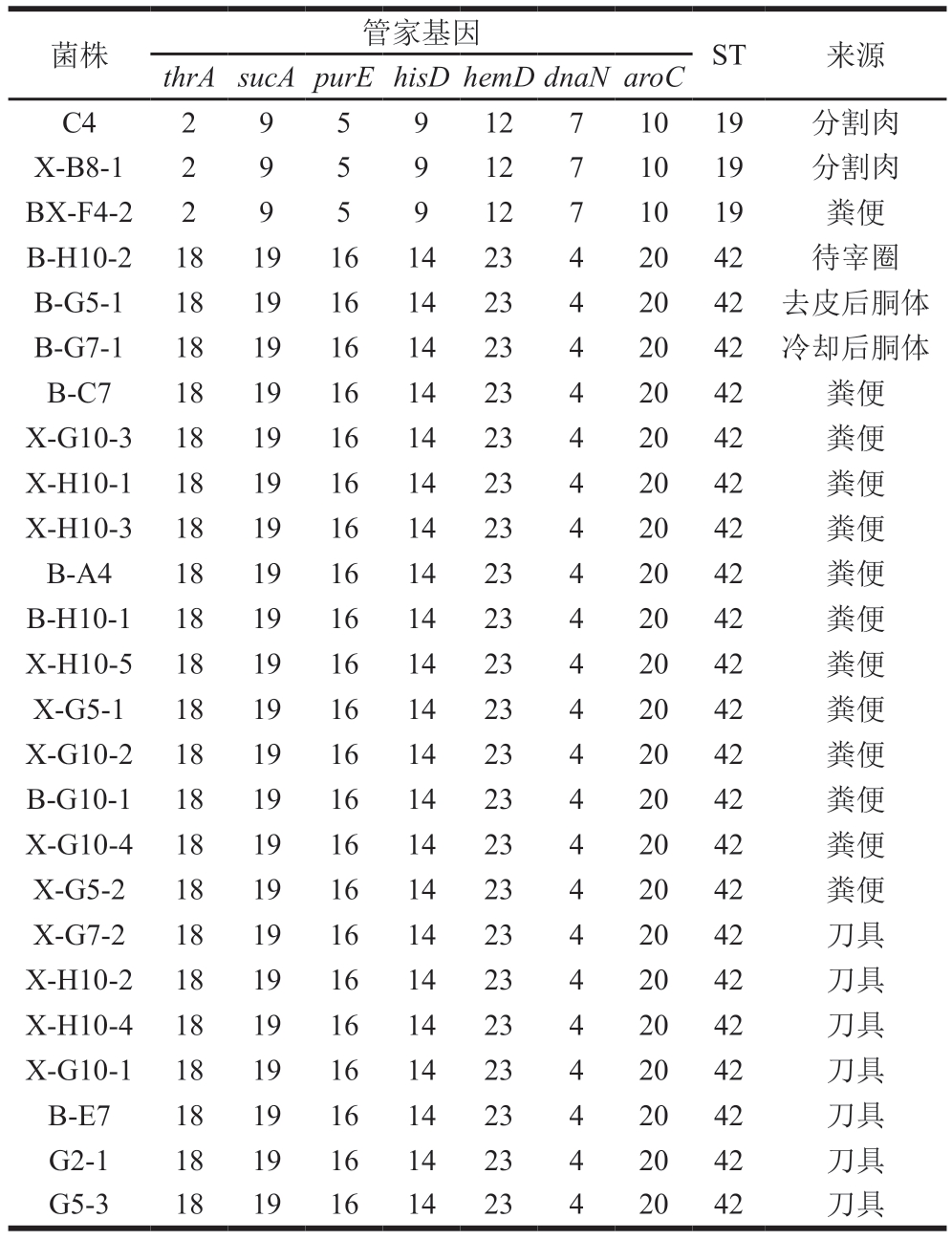
菌株管家基因ST来源thrAsucApurEhisDhemDdnaNaroC C429591271019分割肉X-B8-129591271019分割肉BX-F4-229591271019粪便B-H10-2181916142342042待宰圈B-G5-1181916142342042去皮后胴体B-G7-1181916142342042冷却后胴体B-C7181916142342042粪便X-G10-3181916142342042粪便X-H10-1181916142342042粪便X-H10-3181916142342042粪便B-A4181916142342042粪便B-H10-1181916142342042粪便X-H10-5181916142342042粪便X-G5-1181916142342042粪便X-G10-2181916142342042粪便B-G10-1181916142342042粪便X-G10-4181916142342042粪便X-G5-2181916142342042粪便X-G7-2181916142342042刀具X-H10-2181916142342042刀具X-H10-4181916142342042刀具X-G10-1181916142342042刀具B-E7181916142342042刀具G2-1181916142342042刀具G5-3181916142342042刀具
3 结论
本研究对2 家肉牛屠宰厂进行沙门氏菌流行率调查,共采集435 份样品,流行率为4.1%;不同工厂检出率差异显著,并确认了皮毛和粪便为高风险来源。分离出的沙门氏菌不仅携带高毒力基因,而且普遍呈现出较强的抗生素耐药性。进一步的溯源分析明确了污染途径为粪便-待宰圈-皮毛-去皮后胴体-分割肉,揭示了沙门氏菌在屠宰过程中存在明显的交叉污染风险。生产企业需加强操作规范和卫生措施,以降低污染风险。研究结果不仅为肉类企业制定靶向性卫生干预策略提供了数据支撑,也为监管部门完善沙门氏菌耐药性监测体系与食品安全标准提供了理论参考,对降低食源性疾病负担具有重要现实意义。
[1] 高旭, 刘扬, 罗欣, 等. 酸适应过程中添加牛至精油和氯化钙对沙门氏菌诱导耐酸及其并发的多重抗逆性的影响[J]. 食品科学, 2024,45(20): 136-144. DOI:10.7506/spkx1002-6630-20231124-195
[2] 刘素可, 张彪, 路娟娥, 等. 沙门氏菌在食品中的生存策略及其防控的研究进展[J]. 食品科学, 2022, 43(13): 218-226. DOI:10.7506/spkx1002-6630-20210329-359.
[3] SUN T, LIU Y T, QIN X J, et al. The prevalence and epidemiology of Salmonella in retail raw poultry meat in China: a systematic review and meta-analysis[J]. Foods, 2021, 10(11): 2757. DOI:10.3390/foods10112757.
[4] ADZITEY F, TEYE G A, AMOAKO D G. Prevalence, phylogenomic insights, and phenotypic characterization of Salmonella enterica isolated from meats in the Tamale metropolis of Ghana[J]. Food Science & Nutrition, 2020, 8(7): 3647-3655. DOI:10.1002/fsn3.1647.
[5] 吴海晶, 程逸宇, 沈威, 等. 南京地区猪肉源中沙门氏菌分子分型及耐药性分析[J]. 现代食品科技, 2023, 39(10): 307-314.DOI:10.13982/j.mfst.1673-9078.2023.10.1362.
[6] LIU Y X Y, YANG X Y, LIU Y G, et al. Chinese consumer perception and purchasing behavior of beef: mainly in North and East China[J]. Meat Science, 2025, 220: 109696. DOI:10.1016/j.meatsci.2024.109696.
[7] GEBREMEDHIN E Z, SOBOKA G T, BORANA B M, et al.Prevalence, risk factors, and antibiogram of nontyphoidal Salmonella from beef in Ambo and Holeta Towns, Oromia Region,Ethiopia[J]. International Journal of Microbiology, 2021, 2021: 1-13.DOI:10.1155/2021/6626373.
[8] NAZARI MOGHADAM M, RAHIMI E, SHAKERIAN A, et al.Prevalence of Salmonella Typhimurium and Salmonella Enteritidis isolated from poultry meat: virulence and antimicrobial-resistant genes[J]. BMC Microbiology, 2023, 23(1): 168. DOI:10.1186/s12866-023-02908-8.
[9] 杨克慧, 董鹏程, 刘昀阁, 等. pmrA影响酸胁迫鼠伤寒沙门氏菌生物膜的形成能力[J]. 中国食品学报, 2023, 23(3): 147-156.DOI:10.16429/j.1009-7848.2023.03.016.
[10] CANNING M, BIRHANE M G, DEWEY-MATTIA D, et al.Salmonella outbreaks linked to beef, United States, 2012—2019[J].Journal of Food Protection, 2023, 86(5): 100071. DOI:10.1016/j.jfp.2023.100071.
[11] REN X, YANG D J, YANG Z S, et al. Prevalence and antimicrobial susceptibility of foodborne pathogens from raw livestock meat in China, 2021[J]. Microorganisms, 2024, 12(11): 2157. DOI:10.3390/microorganisms12112157.
[12] XU Z H, WANG M, ZHOU C Y, et al. Prevalence and antimicrobial resistance of retail-meat-borne Salmonella in southern China during the years 2009-2016: the diversity of contamination and the resistance evolution of multidrug-resistant isolates[J]. International Journal of Food Microbiology, 2020, 333: 108790. DOI:10.1016/j.ijfoodmicro.2020.108790.
[13] 靳浩展, 李亮, 魏琦麟, 等. 屠宰场沙门氏菌耐药性分析和毒力基因检测[J]. 中国畜牧兽医, 2022, 49(10): 3943-3952. DOI:10.16431/j.cnki.1671-7236.2022.10.027.
[14] 于淼, 耿英芝, 张铭琰, 等. 辽宁省市售禽肉中沙门氏菌的污染状况及其MLST分型与耐药性分析[J]. 微生物学通报, 2024, 51(7): 2486-2493. DOI:10.13344/j.microbiol.china.230957.
[15] 蔡秋慧, 龚莹婷, 李港回, 等. 一株猪源德尔卑沙门氏菌的分离鉴定、生物学特性以及全基因组序列分析[J]. 食品与发酵工业, 2024,50(11): 18-26. DOI:10.13995/j.cnki.11-1802/ts.037968.
[16] BREUM S Ø, BOEL J. Prevalence of Escherichia coli O157 and verocytotoxin producing E. coli (VTEC) on Danish beef carcasses[J].International Journal of Food Microbiology, 2010, 141(1): 90-96.DOI:10.1016/j.ijfoodmicro.2010.03.009.
[17] MADDEN R H, MURRAY K A, GILMOUR A. Carriage of four bacterial pathogens by beef cattle in Northern Ireland at time of slaughter[J]. Letters in Applied Microbiology, 2007, 44(2): 115-119.DOI:10.1111/j.1472-765X.2006.02064.x.
[18] BARKOCY-GALLAGHER G A, BERRY E D, RIVERABETANCOURT M, et al. Development of methods for the recovery of Escherichia coli O157:H7 and Salmonella from beef carcass sponge samples and bovine fecal and hide samples[J]. Journal of Food Protection, 2002, 65(10): 1527-1534. DOI:10.4315/0362-028X-65.10.1527.
[19] ARNOLD T, SCHOLZ H C, MARG H, et al. Impact of invA-PCR and culture detection methods on occurrence and survival of Salmonella in the flesh, internal organs and lymphoid tissues of experimentally infected pigs[J]. Journal of Veterinary Medicine, Series B, 2004,51(10): 459-463. DOI:10.1111/j.1439-0450.2004.00808.x.
[20] FARDSANEI F, SOLTAN DALLAL M M, DOURAGHI M, et al.Antimicrobial resistance, virulence genes and genetic relatedness of Salmonella enterica serotype Enteritidis isolates recovered from human gastroenteritis in Tehran, Iran[J]. Journal of Global Antimicrobial Resistance, 2018, 12: 220-226. DOI:10.1016/j.jgar.2017.10.005.
[21] CAPUANO F, MANCUSI A, CAPPARELLI R, et al. Characterization of drug resistance and virulotypes of Salmonella strains isolated from food and humans[J]. Foodborne Pathogens and Disease, 2013, 10(11):963-968. DOI:10.1089/fpd.2013.1511.
[22] WANG Y P, LI L, SHEN J Z, et al. Quinolone-resistance in Salmonella is associated with decreased mRNA expression of virulence genes invA and avrA, growth and intracellular invasion and survival[J].Veterinary Microbiology, 2009, 133(4): 328-334. DOI:10.1016/j.vetmic.2008.07.012.
[23] HEITHOFF D M, SHIMP W R, LAU P W, et al. Human Salmonella clinical isolates distinct from those of animal origin[J]. Applied and Environmental Microbiology, 2008, 74(6): 1757-1766. DOI:10.1128/AEM.02740-07.
[24] MIRZAIE S, HASSANZADEH M, ASHRAFI I. Identification and characterization of Salmonella isolates from captured house sparrows[J]. Turkish Journal of Veterinary and Animal Sciences, 2010,34(2): 181-186. DOI:10.3906/vet-0810-43.
[25] RAFFATELLU M, WILSON R P, CHESSA D, et al. SipA, SopA,SopB, SopD, and SopE2 contribute to Salmonella enterica serotype Typhimurium invasion of epithelial cells[J]. Infection and Immunity,2005, 73(1): 146-154. DOI:10.1128/IAI.73.1.146-154.2005.
[26] CHIU C H, SU L H, CHU C H, et al. Detection of multidrug-resistant Salmonella enterica serovar Typhimurium phage types DT102, DT104,and U302 by multiplex PCR[J]. Journal of Clinical Microbiology,2006, 44(7): 2354-2358. DOI:10.1128/jcm.00171-06.
[27] HU Q, COBURN B, DENG W, et al. Salmonella enterica serovar senftenberg human clinical isolates lacking SPI-1[J]. Journal of Clinical Microbiology, 2008, 46(4): 1330-1336. DOI:10.1128/jcm.01255-07.
[28] KUTSUKAKE K, NAKASHIMA H, TOMINAGA A, et al. Two DNA invertases contribute to flagellar phase variation in Salmonella enterica serovar Typhimurium strain LT2[J]. Journal of Bacteriology, 2006,188(3): 950-957. DOI:10.1128/JB.188.3.950-957.2006.
[29] PRAGER R, FRUTH A, TSCHÄPE H. Salmonella enterotoxin(stn) gene is prevalent among strains of Salmonella enterica, but not among Salmonella bongori and other Enterobacteriaceae[J].FEMS Immunology and Medical Microbiology, 1995, 12(1): 47-50.DOI:10.1016/0928-8244(95)00048-C.
[30] ANSARIAN BAREZI A, SHAKERIAN A, RAHIMI E, et al.Examining the extent of contamination, antibiotic resistance, and genetic diversity of Clostridioides (Clostridium) difficile strains in meat and feces of some native birds of Iran[J]. BioMed Research International, 2023, 2023(1): 3524091. DOI:10.1155/2023/3524091.
[31] DONG P C, ZHU L X, MAO Y W, et al. Prevalence and profile of Salmonella from samples along the production line in Chinese beef processing plants[J]. Food Control, 2014, 38: 54-60. DOI:10.1016/j.foodcont.2013.09.066.
[32] GUTEMA F D, AGGA G E, ABDI R D, et al. Prevalence and serotype diversity of Salmonella in apparently healthy cattle: systematic review and meta-analysis of published studies, 2000-2017[J]. Frontiers in Veterinary Science, 2019, 6: 102. DOI:10.3389/fvets.2019.00102.
[33] ZHANG H Q, ZHOU G H, YANG C S, et al. The prevalence,distribution, and diversity of Salmonella isolated from pork slaughtering processors and retail outlets in the Shandong Province of China[J]. Meat Science, 2025, 221: 109734. DOI:10.1016/j.meatsci.2024.109734.
[34] OLIULLA H, MIZAN M F R, ASHRAFUDOULLA M D, et al. The challenges and prospects of using cold plasma to prevent bacterial contamination and biofilm formation in the meat industry[J]. Meat Science, 2024, 217: 109596. DOI:10.1016/j.meatsci.2024.109596.
[35] KANNAN G, MAHAPATRA A K, DEGALA H L. Preharvest management and postharvest intervention strategies to reduce Escherichia coli contamination in goat meat: a review[J]. Animals,2021, 11(10): 2943. DOI:10.3390/ani11102943.
[36] WANG H, HE A N, YANG X Q. Dynamics of microflora on conveyor belts in a beef fabrication facility during sanitation[J]. Food Control,2018, 85: 42-47. DOI:10.1016/j.foodcont.2017.09.017.
[37] ALVAREZ-ORDÓÑEZ A, COUGHLAN L M, BRIANDET R,et al. Biofilms in food processing environments: challenges and opportunities[J]. Annual Review of Food Science and Technology,2019, 10(1): 173-195. DOI:10.1146/annurev-food-032818-121805.
[38] YANG X Q, WANG H, HRYCAUK S, et al. Microbial dynamics in mixed-culture biofilms of Salmonella Typhimurium and Escherichia coli O157:H7 and bacteria surviving sanitation of conveyor belts of meat processing plants[J]. Microorganisms, 2023, 11(2): 421.DOI:10.3390/microorganisms11020421.
[39] KUUS K, KRAMARENKO T, SÕGEL J, et al. Prevalence and serotype diversity of Salmonella enterica in the Estonian meat production chain in 2016-2020[J]. Pathogens, 2021, 10(12): 1622.DOI:10.3390/pathogens10121622.
[40] TIAN Y Q, GU D, WANG F, et al. Prevalence and characteristics of Salmonella spp. from a pig farm in Shanghai, China[J]. Foodborne Pathogens and Disease, 2021, 18(7): 477-488. DOI:10.1089/fpd.2021.0018.
[41] YANG X J, WU Q P, ZHANG J M, et al. Prevalence, bacterial load,and antimicrobial resistance of Salmonella serovars isolated from retail meat and meat products in China[J]. Frontiers in Microbiology, 2019,10: 2121. DOI:10.3389/fmicb.2019.02121.
[42] 干鸿翔, 许安宁, 周璐, 等. 食源和人源鼠伤寒沙门氏菌单相变种的基因组特征和毒力因子对比分析[J]. 食品科学, 2025, 46(6): 116-123. DOI:10.7506/spkx1002-6630-20240821-158.
[43] 刘理慧, 储锦华, 隋雨欣, 等. 沙门氏菌中主要毒力因子的研究进展[J].生物技术通报, 2022, 38(9): 72-83. DOI:10.13560/j.cnki.biotech.bull.1985.2022-0687.
[44] HALLSTROM K N, MCCORMICK B A. The type three secreted effector SipC regulates the trafficking of PERP during Salmonella infection[J]. Gut Microbes, 2016, 7(2): 136-145. DOI:10.1080/194909 76.2015.1128626.
[45] XU Y L, YU Z T, WU S P, et al. Pathogenicity of multidrug-resistant Salmonella Typhimurium isolated from ducks[J]. Microorganisms,2024, 12(7): 1359. DOI:10.3390/microorganisms12071359.
[46] HURTADO-ESCOBAR G A, GRÉPINET O, RAYMOND P, et al.H-NS is the major repressor of Salmonella Typhimurium Pef fimbriae expression[J]. Virulence, 2019, 10(1): 849-867. DOI:10.1080/21505594.2019.1682752.
[47] LIN H H, CHEN H L, JANAPATLA R P, et al. Hyperexpression of type III secretion system of Salmonella typhi linked to a higher cytotoxic effect to monocyte-derived macrophages by activating inflammasome[J]. Microbial Pathogenesis, 2020, 146: 104222.DOI:10.1016/j.micpath.2020.104222.
[48] 刘春红, 吕志航, 张玉倩, 等. 鸡源山夫登堡沙门菌分离鉴定及耐药性和毒力基因检测[J]. 中国畜牧兽医2024, 51(4): 1773-1783.DOI:10.16431/j.cnki.1671-7236.2024.04.044.
[49] 洪意, 谢雅妮, 吴瑜凡, 等. 质粒介导的氨苄西林耐药肠炎沙门氏菌生长特性及抗性研究[J]. 食品与发酵工业, 2023, 49(23): 70-75.DOI:10.13995/j.cnki.11-1802/ts.035114.
[50] VIANA C, SERENO M J, PEGORARO K, et al. Distribution,diversity, virulence genotypes and antibiotic resistance for Salmonella isolated from a Brazilian pork production chain[J]. International Journal of Food Microbiology, 2019, 310: 108310. DOI:10.1016/j.ijfoodmicro.2019.108310.
[51] ZHANG L N, FU Y, XIONG Z Y, et al. Highly prevalent multidrugresistant Salmonella from chicken and pork meat at retail markets in Guangdong, China[J]. Frontiers in Microbiology, 2018, 9: 2104.DOI:10.3389/fmicb.2018.02104.
[52] TADESSE G. A meta-analysis of the proportion of animal Salmonella isolates resistant to drugs used against human salmonellosis in Ethiopia[J]. BMC Infectious Diseases, 2015, 15(1): 84. DOI:10.1186/s12879-015-0835-x.
[53] VAN BOECKEL T P, PIRES J, SILVESTER R, et al. Global trends in antimicrobial resistance in animals in low- and middle-income countries[J]. Science, 2019, 365: eaaw1944. DOI:10.1126/science.aaw1944.
[54] JIU Y G, MENG X R, HONG X, et al. Prevalence and characterization of Salmonella in three typical commercial pig abattoirs in Wuhan,China[J]. Foodborne Pathogens and Disease, 2020, 17(10): 620-627.DOI:10.1089/fpd.2019.2737.
[55] XU Y H, ZHOU X, JIANG Z H, et al. Epidemiological investigation and antimicrobial resistance profiles of Salmonella isolated from breeder chicken hatcheries in Henan, China[J]. Frontiers in Cellular and Infection Microbiology, 2020, 10: 497. DOI:10.3389/fcimb.2020.00497.
[56] ZENG X Y, LÜ S L, QU C, et al. Serotypes, antibiotic resistance, and molecular characterization of non-typhoidal Salmonella isolated from diarrheic patients in Guangxi Zhuang Autonomous Region, China,2014-2017[J]. Food Control, 2021, 120: 107478. DOI:10.1016/j.foodcont.2020.107478.
[57] 刘靓, 李兵兵, 李洁, 等. 全基因组测序分析生畜肉中沙门氏菌血清型、耐药性与毒力因子[J]. 肉类研究, 2024, 38(4): 23-29.DOI:10.7506/rlyj1001-8123-20240421-089.
[58] 余树坤, 刘浪, 谭雅心, 等. 1 起ST19型鼠伤寒沙门氏菌引起的食物中毒调查、溯源分析与探讨[J]. 中国人兽共患病学报, 2024, 40(1):82-89. DOI:10.3969/ji.ssn.1002-2694.2024.00.012
[59] 赵翠, 张庆, 郭树源, 等. 山东省动物源沙门氏菌MLST和血清分型与分布研究[J]. 中国人兽共患病学报, 2017, 33(9): 793-799.DOI:10.3969/j.issn.1002-2694.2017.09.008
[60] 曲梅, 田祎, 黄瑛, 等. 2018—2021年北京市沙门菌血清型及喹诺酮类耐药表型和基因型分析[J]. 现代预防医学, 2022, 49(13): 2413-2419. DOI:10.20043/j.cnki.MPM.202201164.
[61] 肖雪, 张红蕊, 张鹏, 等. 鼠伤寒沙门氏菌群体感应干扰策略研究进展[J]. 食品与发酵工业, 2025, 51(2): 382-389. DOI:10.13995/j.cnki.11-1802/ts.039291.
[62] ZHOU Z H, LI J W, ZHENG H J, et al. Diversity of Salmonella isolates and their distribution in a pig slaughterhouse in Huaian,China[J]. Food Control, 2017, 78: 238-246. DOI:10.1016/j.foodcont.2017.02.064.