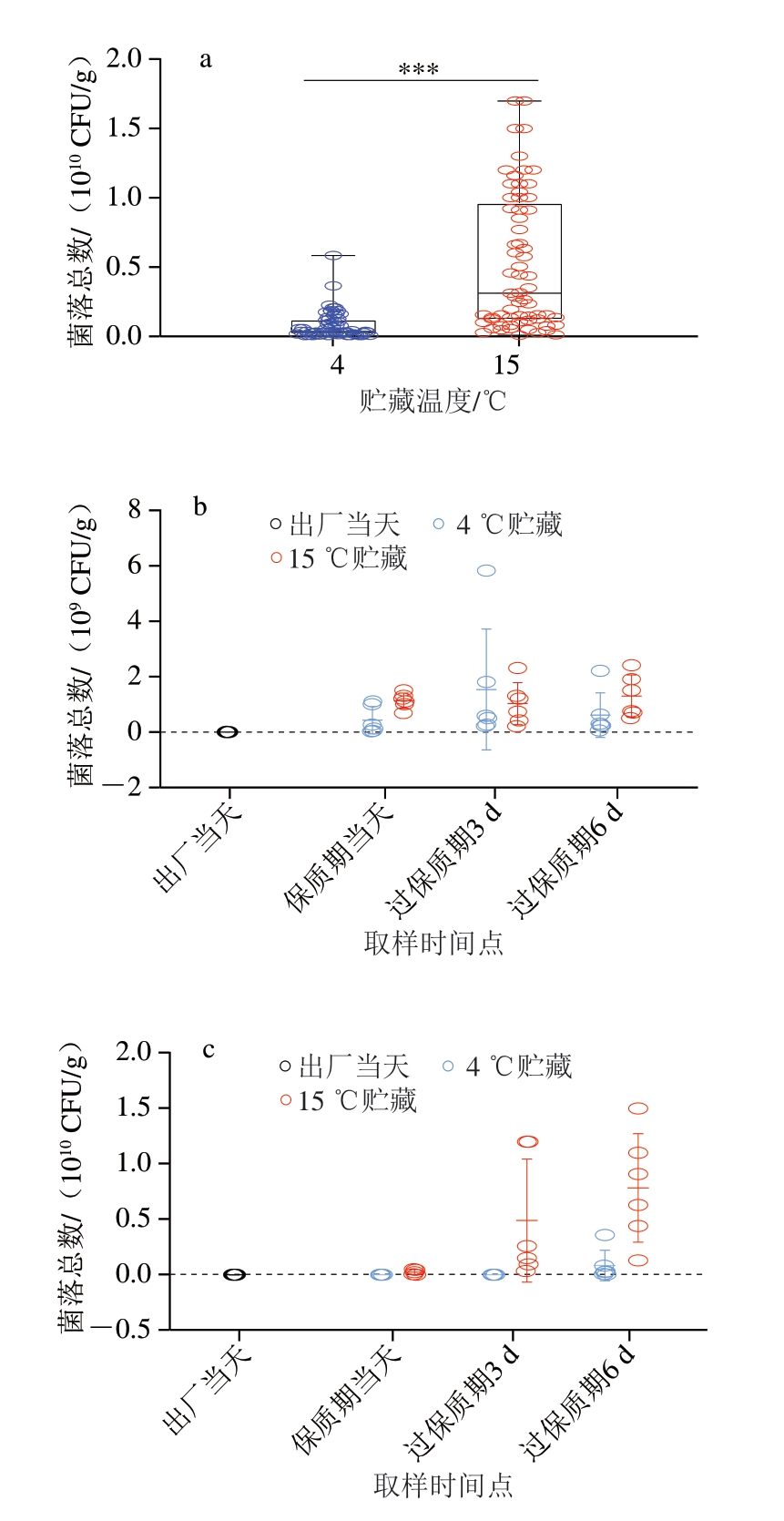
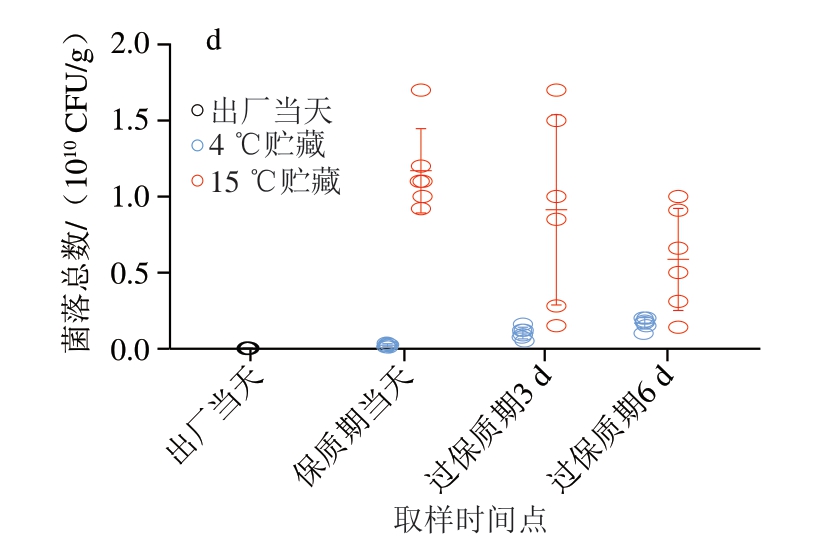
图1 不同贮藏温度下的调理牛排菌落总数
Fig.1 Total number of colonies of processed beef steak at different storage temperatures
a.4 ℃和15 ℃下所有取样点的菌落总数;b~d.分别为F1、F2、F3在各取样点的菌落总数。***.组间差异极显著(P<0.001)。
Dynamic Changes and Interactions of Bacterial Communities in Processed Beef Steak at Different Storage Temperatures
史娜, 邢超, 宋丽萍, 等.不同贮藏温度下调理牛排菌群动态变化和相互作用[J].肉类研究, 2025, 39(4): 30-39.DOI:10.7506/rlyj1001-8123-20241016-264.http://www.rlyj.net.cn
SHI Na, XING Chao, SONG Liping, et al.Dynamic changes and interactions of bacterial communities in processed beef steak at different storage temperatures[J].Meat Research, 2025, 39(4): 30-39.(in Chinese with English abstract)DOI:10.7506/rlyj1001-8123-20241016-264.http://www.rlyj.net.cn
2022年,中国牛肉市场消费量为9 86.1万 t,位居全球第二。在牛排、生冷鲜牛肉、牛肉卷、牛肉丸等众多牛肉产品中,国民对牛排的消费热情持续升高。据《2022年牛排消费趋势洞察》报告显示,2021年线上牛排消费增速持续上涨,同比增长7 倍。牛肉中的水分和营养物质能够为微生物提供生长条件,如假单胞菌属(Pseudomonas)和不动杆菌属(Acinetobacter)等,这些微生物可导致牛肉腐败[1-4]。因此,腐败微生物的生长和代谢活动引起的牛肉腐败成为肉类工业的关注热点。
贮藏温度是影响鲜肉货架期的主要因素[5]。目前,冷藏与冷冻是最常用的2 种肉类贮藏方式,其对应温度范围分别为0~10、-18~-20 ℃[6]。尽管大多数国家已经规定了食品最高贮藏温度限值,但据报道,由于配送系统、运输距离和时间的不稳定性,运输过程中的温度可能达到10 ℃甚至更高。这种不受控制的温度条件可能会显著缩短肉类货架期[7-8]。而且,一些乡村市场的肉类仍然在室温下露天保存,这无疑会增加肉类变质风险[9]。因此,有必要更深入地了解不同贮藏温度下肉品中的微生物组成动态变化及其相互作用。
高通量测序技术和生物信息学分析技术为食品中可培养和不可培养细菌的研究提供了高效的分析工具,使全面描述菌群的动态变化、分析微生物代谢活动与肉类腐败之间的相关性成为可能[10-11]。目前,已有关于采用高通量测序技术表征肉类腐败中微生物组成的报道[12],然而肉类菌群组成在不同贮藏温度下的差异较大。目前,大多数研究集中在不同冷藏或过冷温度(包括4、-1、-2、-3 ℃)或不同包装方式(如气调包装)对菌群的影响[6,13-14],比较脱冷贮藏下肉类菌群组成和种间相互作用的研究较为少见。
本研究将新鲜调理牛排分别进行冷藏(4 ℃)和脱冷贮藏(15 ℃),评估不同贮藏温度与时间对牛排菌群组成的影响,旨在深入了解贮藏温度对牛排菌群及菌种之间潜在相互作用的影响,确定牛排的关键致腐微生物。
在北京大型商超采买3 个品牌的调理牛排(F1、F2、F3),共63 个样品(每种品牌各21 个),样品出厂日期均为当天,保质期2 d。
BagMixer 400稀释用电子秤、DiluFow基本型均质器法国Interscience公司;HPX-25085-III恒温恒湿箱 上海新苗医疗器械制造有限公司。
1.3.1 样品制备
因从保质期内牛排中鉴定的高丰度细菌无法与腐败建立有效联系,因此本研究以不同温度下长时间贮藏至过保质期的调理牛排为样品,分别在冷藏(4 ℃)、脱冷(15 ℃)条件下贮藏,在保质期结束当天、过保质期3 d及过保质期6 d取样,进行菌落总数测定和16S rRNA基因高通量测序。
1.3.2 菌落总数测定
参考GB 4789.2—2022《食品安全国家标准 食品微生物学检验 菌落总数测定》测定菌落总数。
1.3.3 16S rRNA基因高通量测序
1.3.3.1 PacBio文库制备和测序
提取样品总D N A 后,根据全长引物序列合成带有Barcode的特异性引物,进行聚合酶链式反应(polymerase chain reaction,PCR)扩增并对其产物进行纯化、定量和均一化,形成的测序文库(SMRT Bell)质检合格后,采用PacBio Sequel II进行测序。
1.3.3.2 数据预处理
将PacBio下机数据导出为CCS文件后,使用Lima v1.7.0软件,通过Barcode对CCS进行识别,得到Raw-CCS序列数据。使用Cutadapt 1.9.1软件对引物序列进行识别与去除,并进行长度过滤,得到不包含引物序列的Clean-CCS序列。使用UCHME v4.2软件鉴定并去除嵌合体序列,得到Effective-CCS序列。
1.3.3.3 α-多样性、主坐标分析(principal coordinate analysis,PCoA)及属水平网络分析
α-多样性、PCoA及属水平分布分析在BMKCloud中完成。其中,α-多样性分类水平选择属,组间差异检验方法选择Student’s t-test;PCoA分类水平选择属;属水平分布分析选择相对丰度前10。属水平丰度相关性网络图基于R包igraph实现[15],物种数量选择前30,相关性类型选择Spearman,相关系数阈值选择|0.6|,显著性水平选择0.05。采用Gephi v.0.9.2对构建网络图的图密度和PageRank值进行计算,图密度反映菌属之间相互作用程度,图密度越大,表示菌属之间相互作用越频繁[16]。
数据表示为平均值±标准差,多组间数据比较使用SPSS 24.0中方差分析的邓肯多重范围检验。使用R v.4.0.2软件的Hmisc包对属水平相对丰度的Pearson相关性进行分析。
由图1a可知,4 ℃贮藏下,菌落总数为7.4×108 CFU/g,显著低于15 ℃贮藏下菌落总数(5.2×109 CFU/g),脱冷贮藏下调理牛排的菌落总数极显著升高(P<0.001),加速了其腐败变质进程。


图1 不同贮藏温度下的调理牛排菌落总数
Fig.1 Total number of colonies of processed beef steak at different storage temperatures
a.4 ℃和15 ℃下所有取样点的菌落总数;b~d.分别为F1、F2、F3在各取样点的菌落总数。***.组间差异极显著(P<0.001)。
由图1 b ~d 可知,F 1 在出厂当天菌落总数为4.3×106 CFU/g。4 ℃和15 ℃贮藏下,在保质期当天,F1菌落总数分别为4.3×108 CFU/g和1.1×109 CFU/g;在过保质期3 d,F1菌落总数分别为1.5×109 CFU/g和1.0×109 CFU/g;在过保质期6 d,F1菌落总数分别为6.1×108 CFU/g和1.3×109 CFU/g。F2在出厂当天菌落总数为1.0×106 CFU/g。4 ℃和15 ℃贮藏下,在保质期当天,F2菌落总数分别为5.0×106 CFU/g和2.5×108 CFU/g;在过保质期3 d,F2菌落总数分别为7.8×106 CFU/g和4.9×109 CFU/g;在过保质期6 d,F2菌落总数分别为8.5×108 CFU/g和7.9×109 CFU/g。F3在出厂当天菌落总数为2.3×107 CFU/g。4 ℃和15 ℃贮藏下,在保质期当天,F3菌落总数分别为2.3×108 CFU/g和1.2×1010 CFU/g;在过保质期3 d,F3菌落总数分别为1.0×109 CFU/g和9.1×109 CFU/g;在过保质期6 d,F3菌落总数分别为1.7×109 CFU/g和5.9×109 CFU/g。目前,我国尚未针对生肉制定统一的菌落总数限量指标。欧盟、美国及日本规定每克牛肉需氧平板计数约在5.0×106 CFU/g以下,视为合格。由上述结果可知,本研究所取调理牛排F1和F2菌落总数在出厂当天均小于该限值,F3菌落总数则大于该限值。在保质期当天,仅4 ℃贮藏的F2菌落总数符合该限值标准,F1和F3菌落总数均大于该限值。15 ℃贮藏下的牛排细菌生长更快,可加速牛排腐败变质。
α-多样性反映牛排菌群物种丰富度和多样性[11]。Shannon指数用以描述菌群多样性。Chao1指数用以描述菌群物种丰富度,其值越高,菌群物种丰富度越高。由表1、2可知,不同贮藏温度和时间下,F1、F2、F3菌群多样性均未发生显著改变,这表明初始菌群中的部分细菌参与了牛排的腐败变质过程,多种适应性强的细菌在牛排腐败过程依然保持生长与繁殖。值得注意的是,F2菌群丰富度在一定程度上受到贮藏时间和温度的影响,但与对照组相比差异不显著。
表1 不同贮藏温度和时间下调理牛排菌群的Shannon指数(n =3)
Table 1 Shannon index of bacterial community in processed beef steak at different storage temperatures and times (n = 3)
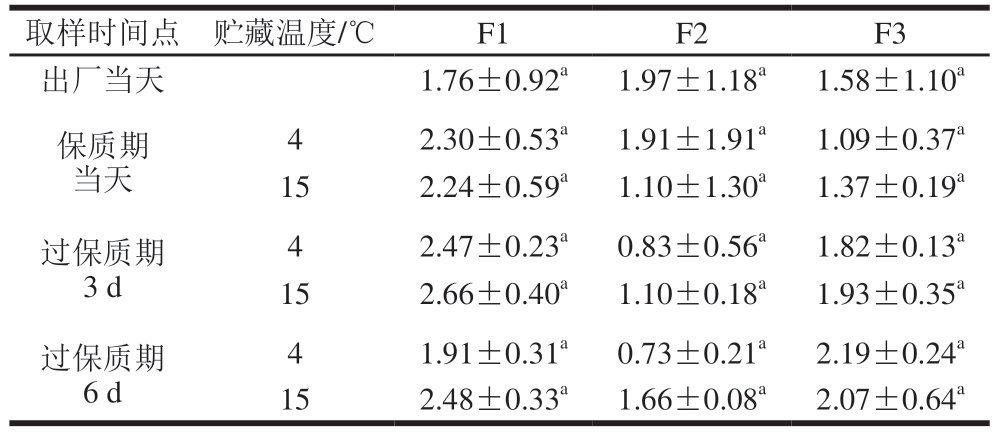
注:同列小写字母不同表示组间差异显著(P<0.05)。表2同。
取样时间点 贮藏温度/℃F1F2F3出厂当天1.76±0.92a1.97±1.18a1.58±1.10a保质期当天42.30±0.53a1.91±1.91a1.09±0.37a 152.24±0.59a1.10±1.30a1.37±0.19a过保质期3 d过保质期6 d 42.47±0.23a0.83±0.56a1.82±0.13a 152.66±0.40a1.10±0.18a1.93±0.35a 41.91±0.31a0.73±0.21a2.19±0.24a 152.48±0.33a1.66±0.08a2.07±0.64a
表2 不同贮藏温度和时间下调理牛排菌群的Chao1指数(n =3)
Table 2 Chao1 index of bacterial community in processed beef steak at different storage temperatures and times (n = 3)
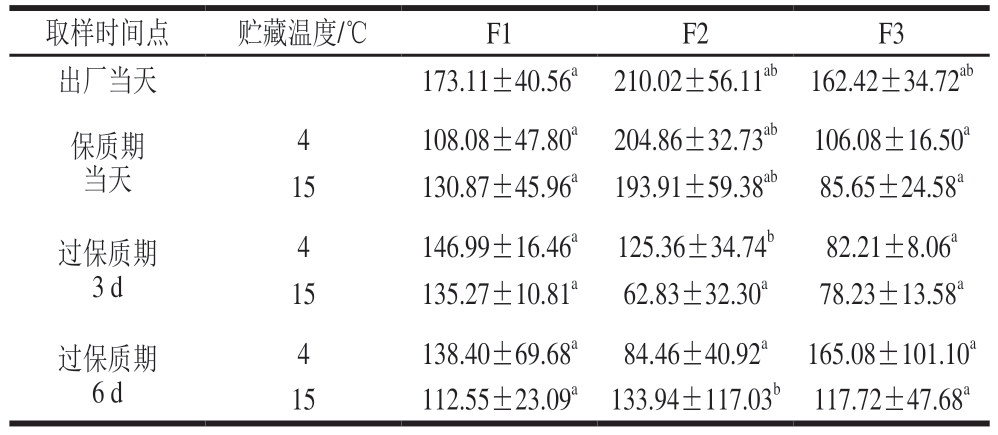
取样时间点贮藏温度/℃F1F2F3出厂当天173.11±40.56a 210.02±56.11ab 162.42±34.72ab保质期当天151过保质期41 3 d151过保质期41 6 d151 4108.08±47.80a 204.86±32.73ab 106.08±16.50a 30.87±45.96a 193.91±59.38ab85.65±24.58a 46.99±16.46a 125.36±34.74b82.21±8.06a 35.27±10.81a62.83±32.30a78.23±13.58a 38.40±69.68a84.46±40.92a 165.08±101.10a 12.55±23.09a 133.94±117.03b 117.72±47.68a
本研究选择相对丰度排名前10的菌属(占总菌属丰度的90%以上,为优势菌属)进行相对丰度分析。由图2a可知,在出厂当天,F1中相对丰度排名前3的优势菌属分别为嗜冷杆菌属(Psychrobacter)(52.4%)、肉杆菌属(Carnobacterium)(15.3%)、索丝菌属(Brochothrix)(13.5%);F2中相对丰度排名前3的优势菌属分别为弯曲乳杆菌属(Latilactobacillus)(34.8%)、肉杆菌属(29.0%)、索丝菌属(10.0%);F3中相对丰度排名前3的优势菌属分别为肉杆菌属(46.2%)、弯曲乳杆菌属(20.1%)、索丝菌属(12.6%)。其中,嗜冷杆菌属、弯曲乳杆菌属及肉杆菌属分别为3 种牛排出厂时相对丰度最高的优势菌属。肉杆菌属和索丝菌属在3 种牛排中均为优势菌属。Liang Ce等[6]对不同贮藏温度下新鲜羊肉菌群变化的研究显示,冷藏(4 ℃)和过冷(-1.5 ℃)贮藏下索丝菌属为优势腐败菌属,嗜冷杆菌属则为超冷(-4 ℃)和超冷冻(-9 ℃)贮藏下的优势腐败菌属。Fang Jinyu等[17]研究表明,Brochothrix thermosphata BT27为4 ℃好氧贮藏条件下牛肉的优势腐败菌。由此可见,本研究中出厂当天牛排已存在报道的腐败菌属。
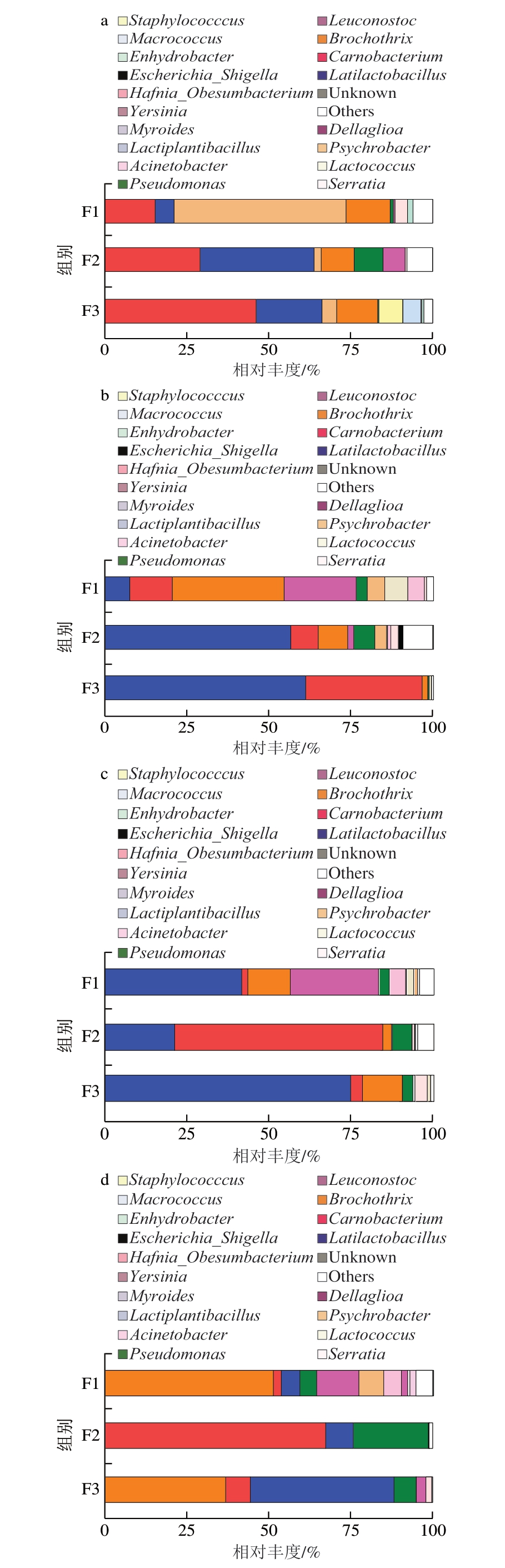
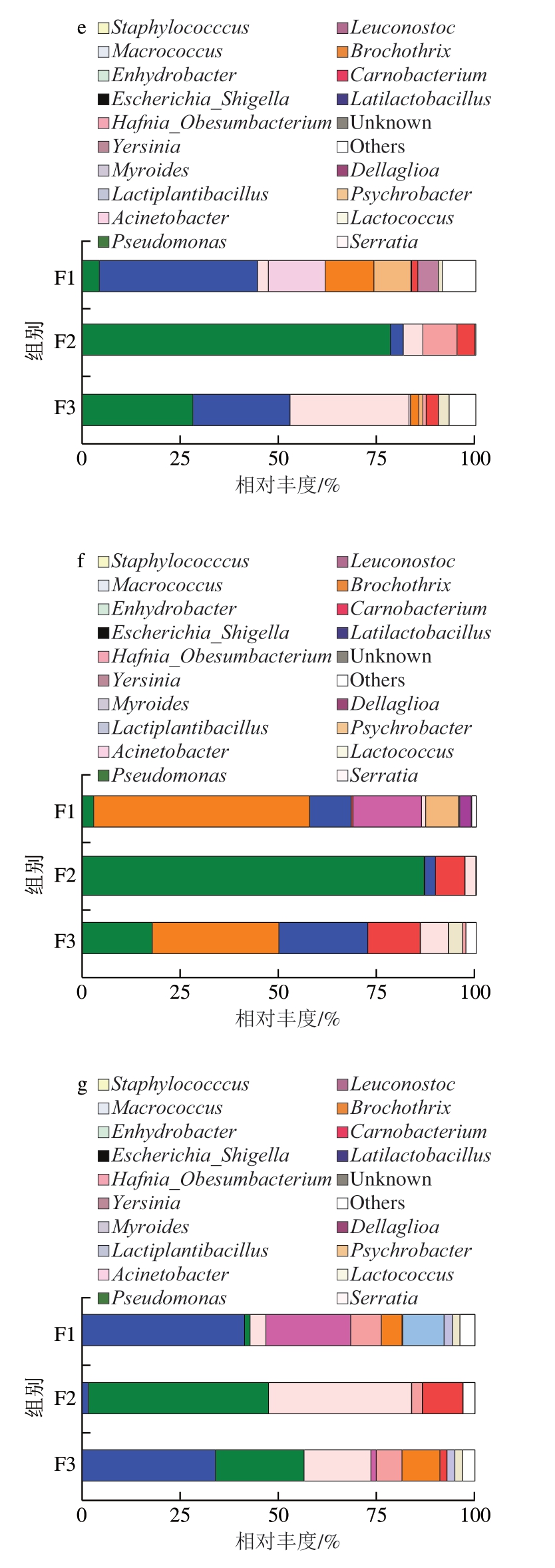
图2 不同贮藏温度和时间下调理牛排优势菌属分布
Fig.2 Distribution of dominant bacterial genera in processed beef steak at different temperatures and times
a.出厂当天;b、c.分别为4 ℃和15 ℃贮藏下保质期当天;d、e.分别为4 ℃和15 ℃贮藏下过保质期3 d;f、g.分别为4 ℃和15 ℃下过保质期6 d。图3、4、6、7同。
由图2b、c可知,在保质期当天,与4 ℃贮藏相比,脱冷贮藏并不影响F1、F2与F3的菌群组成,但不同贮藏温度下牛排优势菌属存在差异。具体地,4 ℃贮藏F1中相对丰度最高的优势菌属为索丝菌属(34.1%),F2、F3中相对丰度最高的优势菌属均为弯曲乳杆菌属(F2:56.5%,F3:61.1%);15 ℃贮藏下F1、F3中相对丰度最高的优势菌属均为弯曲乳杆菌属(F1:41.8%,F3:74.9%),F2中相对丰度最高的优势菌属为肉杆菌属(63.2%)。
由图2d、e可知,在过保质期3 d时,4 ℃和15 ℃贮藏下牛排均已彻底腐败。4 ℃贮藏F1、F2、F3中相对丰度最高的优势菌属分别为索丝菌属(51.5%)、肉杆菌属(67.0%)、弯曲乳杆菌属(43.8%),这与出厂当天、保质期当天牛排的优势菌属类似。15 ℃贮藏F1、F2、F3中相对丰度最高的优势菌属分别为弯曲乳杆菌属(40.2%)、假单胞菌属(78.3%)、沙雷氏菌属(Serratia)(30.2%),表明过保质期后15 ℃贮藏牛排的优势腐败菌发生改变。
由图2f、g可知,过保质期6 d时,随着腐败程度的增加,4 ℃贮藏F1、F3中相对丰度最高的优势菌属均为索丝菌属(F1:54.8%,F3:32.1%),F2中相对丰度最高的优势菌属为假单胞菌属(86.8%);15 ℃贮藏F1、F3中相对丰度最高的则为弯曲乳杆菌属(F1:41.3%,F3:33.9%),F2中相对丰度最高的优势菌属仍为假单胞菌属(45.9%)。
对比可知,4 ℃贮藏时,F1中的索丝菌属丰度逐渐升高,而15 ℃贮藏时,F1中的弯曲乳杆菌属则始终为相对丰度最高的优势菌属;F2中相对丰度最高的优势菌属的出现受冷藏影响呈现滞后性,即假单胞菌属在过保质期6 d时成为相对丰度最高的优势菌属,而在15 ℃贮藏时这一现象过保质期3 d便已出现。F3中优势菌属在2 种贮藏条件下呈现类似的动态演替,即在保质期当天出现相对丰度最高的优势菌属,随着贮藏时间的延长,更多细菌出现相对丰度增高的现象。肉类涉及分切、运输、销售等诸多环节,可能导致样品中不同细菌丰度存在差异[18-20]。然而,本研究所抽取的F1和F3中的菌群结构在4 ℃和15 ℃贮藏下比较稳定,优势菌属的相对丰度变化明显,未引入新的优势菌属。这可能是由于从生产到销售过程中F1和F3处于较为稳定的环境,而F2在贮藏过程中,优势菌属发生改变,可能是由细菌种间的相互作用引起的生态位竞争所导致。
由图3可知,在出厂当天,3 个品牌牛排样品的置信区间相交,表明此时各样品菌群组成相似。随着贮藏时间的延长,4 ℃和15 ℃下,F1、F2、F3牛排样品各自聚类,表明3 个品牌的牛排菌群组成出现差异。
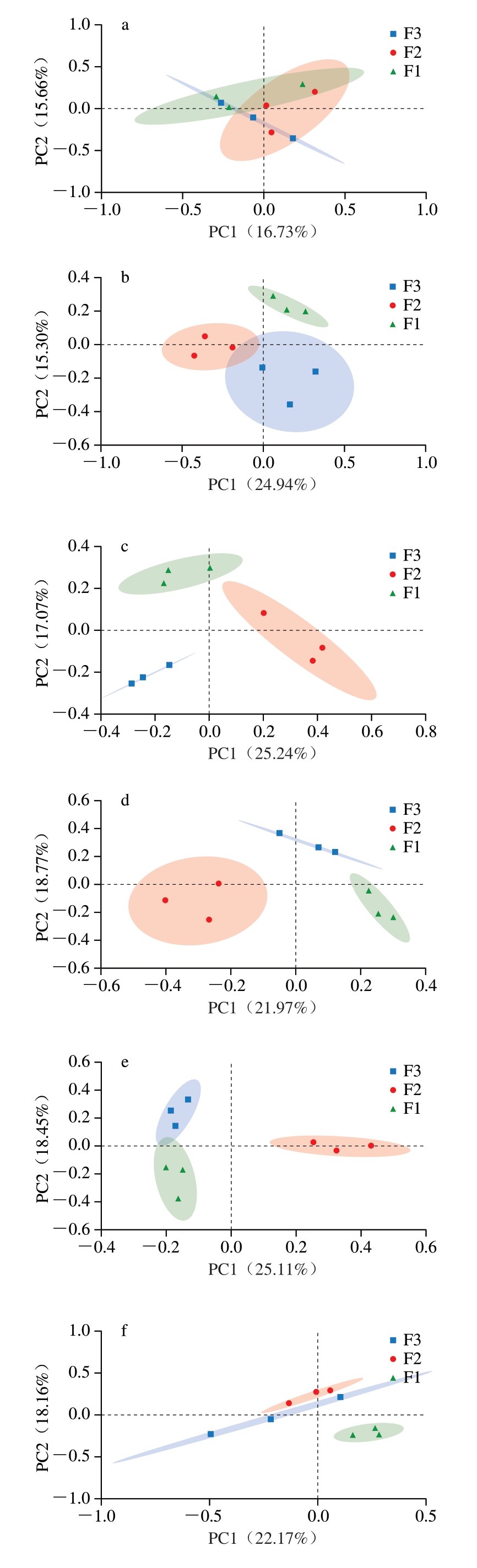
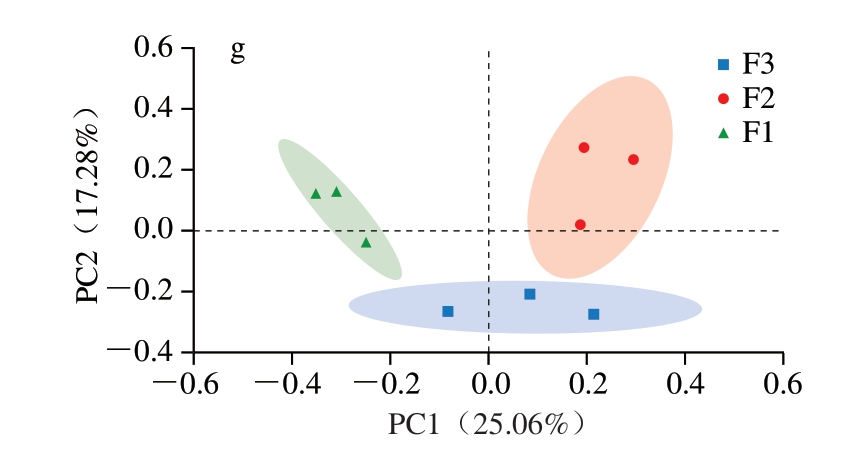
图3 3 个品牌调理牛排菌群的PCoA图
Fig.3 PCoA plots of bacterial communities in three brands of commercial processed beef steak
为探究腐败过程中低丰度菌属与高丰度细菌属之间的相关性,对F1、F2、F3菌属相对丰度进行相关性分析,以相对丰度前30的菌属构建丰度相关性网络,见图4。
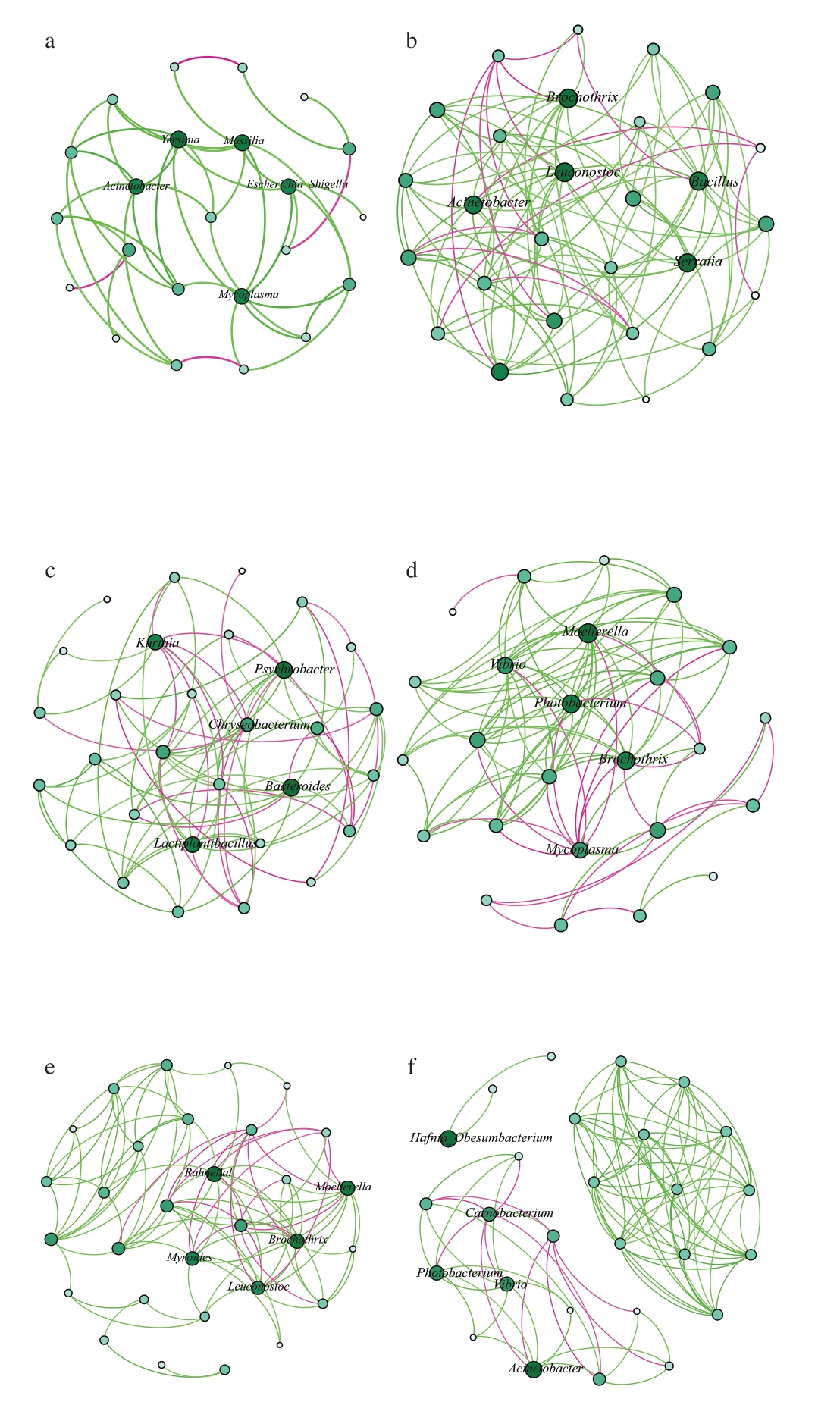
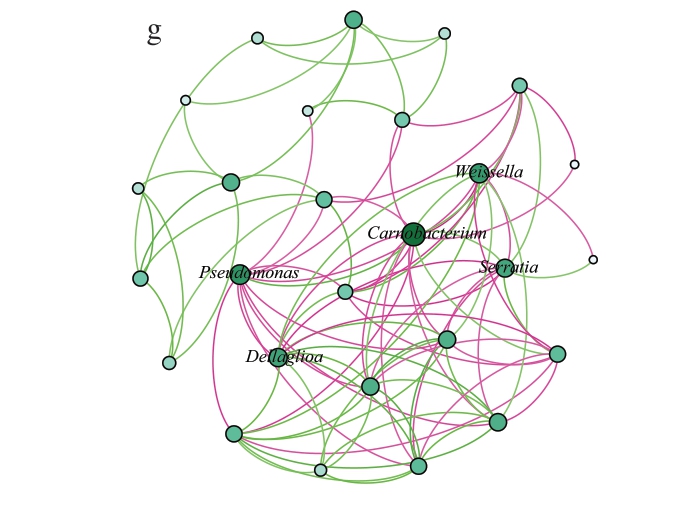
图4 不同贮藏温度和时间下调理牛排菌属丰度相关性网络图
Fig.4 Abundance correlation network diagrams of bacteria genus in processed beef steak stored at different temperatures for different times
由图5可知,保质期当天、过保质期3 d与过保质期6 d牛排在4 ℃和15 ℃下的菌属丰度相关性网络图的图密度均高于出厂当天,表明随着贮藏时间的延长,细菌之间丰度变化更为复杂。此外,4 ℃贮藏下,相关性网络图的图密度明显大于15 ℃,这表明4 ℃贮藏牛排中细菌之间的相互作用更加频繁与复杂。
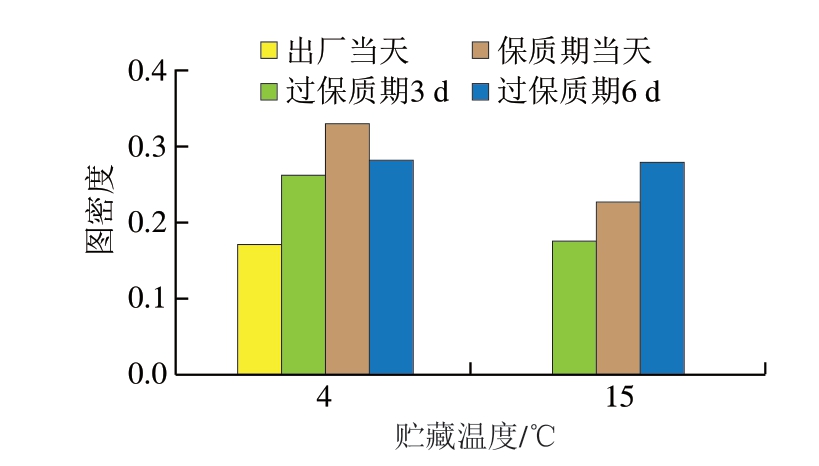
图5 丰度相关性网络图的图密度
Fig.5 Graph density for abundance correlation network diagrams
Page Rank 值能够反映节点在网络图中的重要性[21-23]。由图6可知,PageRank值排名前5的菌属中,不动杆菌属和索丝菌属出现频率最高,共在5 个网络图中出现。不动杆菌属在4 ℃下的2 个网络图中出现。虽然不动杆菌属并不是优势菌属,但相关性网络分析结果提示其重要性,强调非优势菌属在牛排腐败进程中也具有关键作用。已有研究[24-27]表明,不动杆菌属能够造成冷藏期间南美白对虾、金枪鱼、冰鲜鸡肉及水煮小龙虾等多种食品基质腐败。
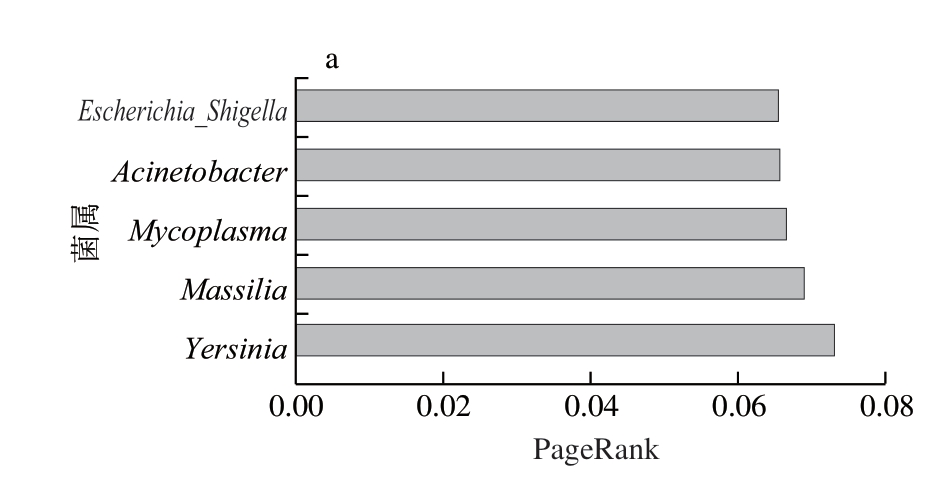
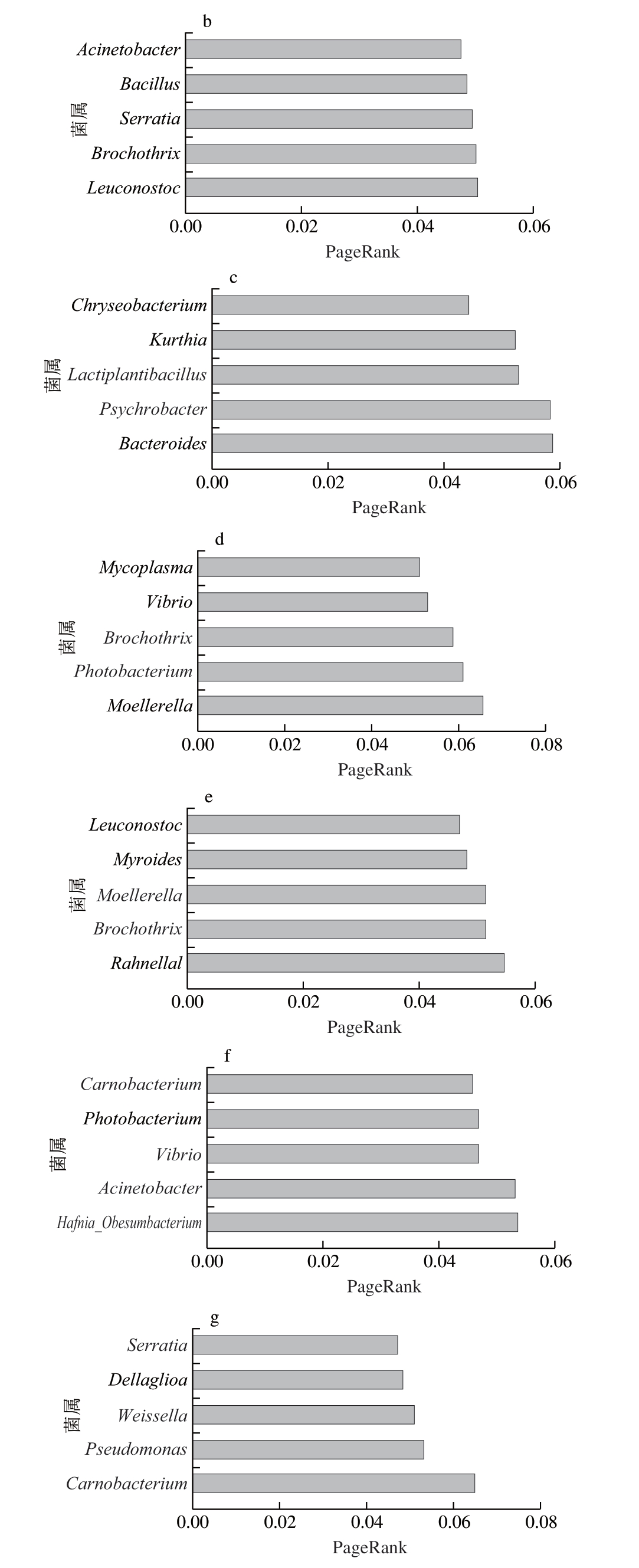
图6 丰度相关性网络图中菌属的PageRank值
Fig.6 PageRank values of bacterial genera in abundance correlation network diagrams
微生物群落并非由众多微生物简单拼凑而成,而是不同物种间通过资源竞争、代谢互补、信号交流和基因水平转移等相互作用形成的高度复杂的生态系统。微生物群落在地球化学元素驯化、农业生产和人类健康等领域承担着重要功能[28-29]。在食品领域,微生物平衡体系与食品腐败密切相关。Andreevskaya等[30]探究3 种嗜冷乳酸菌(Leuconostoc gelidum、Lactococcus piscium和Lactobacillus oligofermentans)在肉类腐败中的相互作用发现,3 种嗜冷乳酸菌的种间相互作用强烈影响碳水化合物分解代谢/运输、发酵、能量产生和翻译相关基因表达。Joishy等[31]通过构建生凝乳(由生乳制备的凝乳)、煮沸乳与凝乳的核心细菌共生网络发现,凝乳中Lactobacillus丰度具有优势,且与Enterococcus、Streptococcus及Leuconostoc丰度呈负相关,推测与其内部存在生态位竞争有关。对与相关性网络图中关键节点菌属相对丰度呈现正/负相关的菌属进行统计,由图7可知,优势腐败菌属相对丰度和关键节点菌属相对丰度之间存在相关性。例如,出厂当天牛排菌群相关性网络中,不动杆菌属与沙雷氏菌属相对丰度相关性为0.72,保质期当天(4 ℃)的不动杆菌属与假单胞菌属、索丝菌属相对丰度相关性分别为0.75、0.78,沙雷氏菌属和索丝菌属相v对丰度相关性为0.75。4 ℃贮藏下,过保质期3 d时不动杆菌属与索丝菌属相对丰度相关性为0.83,过保质期6 d时不动杆菌属与假单胞菌属、索丝菌属的相关性分别为-0.87、0.80;15 ℃贮藏下,过保质期3 d时索丝菌属与假单胞菌属相对丰度相关性为-0.83。这表明致腐细菌在牛排腐败过程中可能存在相互作用。值得注意的是,4 ℃贮藏下,保质期当天不动杆菌和假单胞菌相对丰度呈现正相关性,而在过保质期后,不动杆菌和假单胞菌相对丰度呈现负相关,提示致腐菌之间可能也存在相互竞争。
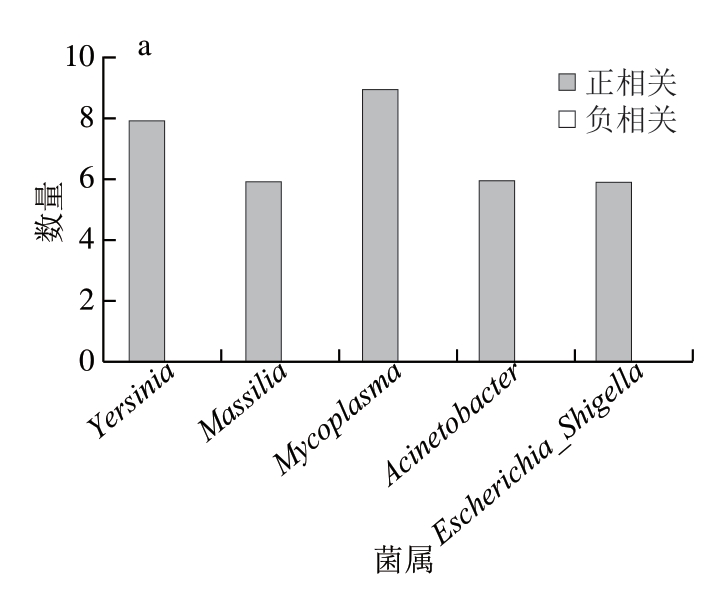
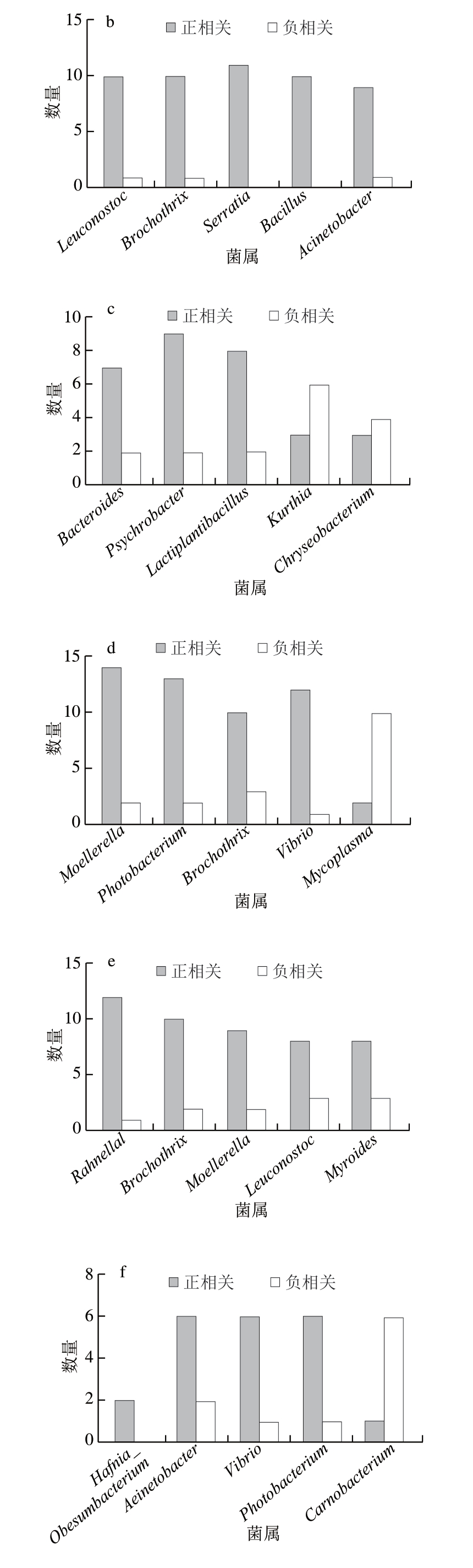
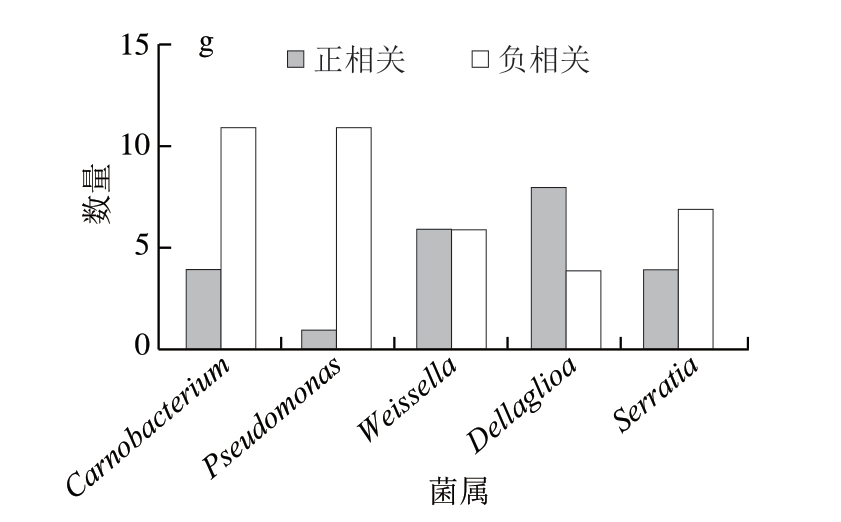
图7 PageRank>0.4节点上相对丰度具有相关性的菌属数量
Fig.7 Number of bacterial genera that correlated in abundance with nodes with a PageRank value of > 0.4
索丝菌属、假单胞菌属、沙雷氏菌属、肉杆菌属及弯曲乳杆菌属是牛排在不同贮藏温度与时间下的优势菌属。其中,肉杆菌属、弯曲乳杆菌属及假单胞菌属为4、15 ℃下的优势菌属。而索丝菌属仅在4 ℃下成为优势菌属、沙雷氏菌属仅在15 ℃下成为优势菌属,表明不同贮藏温度下牛排腐败菌群组成存在差异。不动杆菌属与沙雷氏菌属、假单胞菌属、索丝菌属,沙雷氏菌属与索丝菌属,索丝菌属与假单胞菌属相对丰度在特定贮藏温度和时间下存在相关性,表明牛排腐败菌之间存在潜在相互作用,这一结果提供了肉类腐败菌种间相互作用的高通量测序证据。本研究可为牛排质量控制中腐败菌评估体系的完善提供理论基础。
[1] ERCOLINI D, RUSSO F, NASI A, et al.Mesophilic and psychrotrophic bacteria from meat and their spoilage potential in vitro and in beef[J].Applied and Environmental Microbiology, 2009, 75(7):1990-2001.DOI:10.1128/AEM.02762-08.
[2] ZHU Y L, WANG W, LI M, et al.Microbial diversity of meat products under spoilage and its controlling approaches[J].Frontiers in Nutrition,2022, 9: 1078201.DOI:10.3389/fnut.2022.1078201.
[3] 方金玉.冷鲜牛肉中假单胞菌和热死环丝菌致腐机制及其光动力杀菌控制研究[D].杭州: 浙江工商大学, 2022.DOI:10.27462/d.cnki.ghzhc.2022.000876.
[4] 邓森荣, 向蓉, 魏琦麟, 等.冷鲜肉微生物种类及微生物预测模型的研究进展[J].广东农业科学, 2021, 48(3): 133-141.DOI:10.16768/j.issn.1004-874X.2021.03.016.
[5] NYCHAS G J, SKANDAMIS P N, TASSOU C C, et al.Meat spoilage during distribution[J].Meat Science, 2008, 78(1/2): 77-89.DOI:10.1016/j.meatsci.2007.06.020.
[6] LIANG C, ZHANG D Q, ZHENG X C, et al.Effects of different storage temperatures on the physicochemical properties and bacterial community structure of fresh lamb meat[J].Food Science of Animal Resources, 2021, 41(3): 509-526.DOI:10.5851/kosfa.2021.e15.
[7] DOULGERAKI A I, ERCOLINI D, VILLANI F, et al.Spoilage microbiota associated to the storage of raw meat in different conditions[J].International Journal of Food Microbiology, 2012,157(2): 130-141.DOI:10.1016/j.ijfoodmicro.2012.05.020.
[8] GIARRATANA F, NALBONE L, ZIINO G, et al.Temperature fluctuations along food supply chain: a dynamic and stochastic predictive approach to establish the best temperature value in challenge tests for Listeria monocytogenes[J].Italian Journal of Food Safety,2022, 11(1): 9981.DOI:10.4081/ijfs.2022.9981.
[9] MIAO Z M, LI S, QIN K, et al.Prevalence and antimicrobial resistance of Salmonella isolates recovered from retail pork in major village markets in Tai’an region, China[J].Journal of Food Protection,2017, 80(10): 1635-1640.DOI:10.4315/0362-028X.JFP-17-019.
[10] JOHNSON J S, SPAKOWICZ D J, HONG B Y, et al.Evaluation of 16S rRNA gene sequencing for species and strain-level microbiome analysis[J].Nature Communications, 2019, 10(1): 5029.DOI:10.1038/s41467-019-13036-1.
[11] ZHANG T Y, LI H Z, MA S L, et al.The newest oxford nanopore r10.4.1 full-length 16S rRNA sequencing enables the accurate resolution of species-level microbial community profiling[J].Appiled and Environmental Microbiology, 2023, 89(10): e0060523.DOI:10.1128/aem.00605-23.
[12] YANG X Y, ZHU L X, ZHANG Y M, et al.Microbial community dynamics analysis by high-throughput sequencing in chilled beef longissimus steaks packaged under modified atmospheres[J].Meat Science, 2018, 141: 94-102.DOI:10.1016/j.meatsci.2018.03.010.
[13] YANG J, YANG X Y, LIANG R R, et al.The response of bacterial communities to carbon dioxide in high-oxygen modified atmosphere packaged beef steaks during chilled storage[J].Food Research International, 2022, 151: 110872.DOI:10.1016/j.foodres.2021.110872.
[14] TOOMIK E, ROOD L, BOWMAN J P, et al.Microbial spoilage mechanisms of vacuum-packed lamb meat: a review[J].International Journal of Food Mcirobiology, 2023, 387: 110056.DOI:10.1016/j.ijfoodmicro.2022.110056.
[15] LIU D H, ZHANG R T, S CHEN, et al.Analysis of gastric microbiome reveals three distinctive microbial communities associated with the occurrence of gastric cancer[J].BMC Microbiology, 2022, 22: 184.DOI:10.1186/s12866-022-02594-y.
[16] JACOMY M, VENTURINI T, HEYMANN S, et al.ForceAtlas2,a continuous graph layout algorithm for handy network visualization designed for the Gephi software[J].PLoS ONE, 2014, 9: e98679.DOI:10.1371/journal.pone.0098679.
[17] FANG J Y, FENG L F, LU H X, et al.Metabolomics reveals spoilage characteristics and interaction of Pseudomonas lundensis and Brochothrix thermosphacta in refrigerated beef[J].Food Research International, 2022, 156: 111139.DOI:10.1371/journal.pone.0098679.
[18] VAN BA H, SEO H W, SEONG P N, et al.The fates of microbial populations on pig carcasses during slaughtering process, on retail cuts after slaughter, and intervention efficiency of lactic acid spraying[J].International Journal of Food Microbiology, 2019, 294: 10-17.DOI:10.1016/j.ijfoodmicro.2019.01.015.
[19] PARK J, BAE D, KIM S A.Microbial trace investigation throughout the entire chicken supply chain based on metagenomic high-throughput sequencing[J].Food Research International, 2023, 169: 112775.DOI:10.1016/j.foodres.2023.112775.
[20] CAMPOS CALERO G, CABALLERO GOMEZ N, et al. In silico mapping of microbial communities and stress responses in a porcine slaughterhouse and pork products through its production chain, and the efficacy of HLE disinfectant[J].Food Research International, 2020,136: 109486.DOI:10.1016/j.foodres.2020.109486.
[21] TANG H, SHI R X, HE T S, et al.Tensorflow solver for quantum PageRank in large-scale networks[J].Science Bulletin, 2021, 66(2):120-126.DOI:10.1016/j.scib.2020.09.009.
[22] GONG Y H.Hedgerank: heterogeneity-aware, energy-efficient partitioning of personalized PageRank at the edge[J].Micromachines,2023, 14(9): 1714.DOI:10.3390/mi14091714.
[23] MA K, LI J W, ZHAO M Y, et al.PPRTGI: a personalized PageRank graph neural network for TF-target gene interaction detection[J].IEEE/ACM Transactions on Computational Biology and Bioinformatics,2024, 21(3): 480-491.DOI:10.1109/TCBB.2024.3374430.
[24] XIA J Y, JIANG N, ZHANG B, et al.Bacterial changes in boiled crayfish between different storage periods and characterizations of the specific spoilage bacteria[J].Foods, 2023, 12(16): 3006.DOI:10.3390/foods12163006.
[25] ZHU S Q, WU H H, ZHANG C L, et al.Spoilage of refrigerated Litopenaeus vannamei: eavesdropping on Acinetobacter acylhomoserine lactones promotes the spoilage potential of Shewanella baltica[J].Journal of Food Science and Technology, 2018, 55(5):1903-1912.DOI:10.1007/s13197-018-3108-z.
[26] WANG X Y, XIE J.Growth kinetics and spoilage potential of coculturing Acinetobacter johnsonii and Pseudomonas fluorescens from bigeye tuna (Thunnus obesus) during refrigerated storage[J].Current Microbiology, 2020, 77(8): 1637-1646.DOI:10.1007/s00284-020-01978-5.
[27] ZHANG T, DING H, CHEN L, et al.Characterization of chilled chicken spoilage using an integrated microbiome and metabolomics analysis[J].Food Research International, 2021, 144: 110328.DOI:10.1016/j.foodres.2021.110328.
[28] FALKOWSKI P G, FENCHEL T, DELONG E F.The microbial engines that drive earth’s biogeochemical cycles[J].Science, 2008,320: 1034-1039.DOI:10.1126/science.1153213.
[29] CHO I, BLASER M J.The human microbiome: at the interface of health and disease[J].Nature Reviews Genetics, 2012, 13: 260-270.DOI:10.1038/nrg3182.
[30] ANDREEVSKAYA M, JAASKELAINEN E, et al.Food spoilageassociated Leuconostoc, Lactococcus and Lactobacillus species display different survival strategies in response to competition[J].Applied and Environmental Microbiology, 2018, 84: e00554-18.DOI:10.1128/AEM.00554-18.
[31] JOISHY T K, DEHINGIA M, KHAN M R.Bacterial diversity and metabolite profiles of curd prepared by natural fermentation of raw milk and back sloping of boiled milk[J].World Journal of Microbiology & Biotechnology, 2019, 35: 102.DOI:10.1007/s11274-019-2677-y.