表1 5 种蛋白酶最适水解条件
Table 1 Optimal hydrolysis conditions for five proteases

条件木瓜蛋白酶 碱性蛋白酶 中性蛋白酶 风味蛋白酶 胰蛋白酶温度/℃5050505037 pH710778
Isolation, Purification, Structural Identification and Action Mechanism of Antioxidant Peptides Derived from Porcine Bones
陈露露, 雷阳, 郝阳雪, 等.猪骨源抗氧化肽分离纯化、结构鉴定及抗氧化作用机制[J].肉类研究, 2025, 39(3): 8-17.DOI:10.7506/rlyj1001-8123-20240809-203.http://www.rlyj.net.cn
CHEN Lulu, LEI Yang, HAO Yangxue, et al.Isolation, purification, structural identification and action mechanism of antioxidant peptides derived from porcine bones[J].Meat Research, 2025, 39(3): 8-17.(in Chinese with English abstract)DOI:10.7506/rlyj1001-8123-20240809-203.http://www.rlyj.net.cn
猪肉在我国肉食消费结构中占主导地位,据国家统计局官网报道,2023年我国猪肉产量达到5 794万 t,伴随猪骨产量也高达600多万 t。但目前畜禽骨资源的整体利用率低于总产量的10%,大部分猪骨未能得到有效利用而被遗弃,不仅导致资源浪费[1],更因富含营养物质的猪骨易于腐败,从而导致严重的环境污染问题[2]。猪骨富含多种氨基酸,主要包括Gly、Pro、Ala与Asp等主要氨基酸[3]。此外,猪骨经加工后具有一定的抗氧化活性[4-5],可以延缓皮肤老化、预防动脉粥样硬化等,能够显著提高其附加值。
研究表明,机体衰老与其产生的活性氧密切相关。抗氧化剂可清除体内过量的自由基,在减轻机体氧化损伤方面起着重要作用[6]。近年来,食源性生物活性肽凭借其结构简单、安全、易吸收及无免疫反应等特点,在抗氧化剂的开发应用中备受青睐。因此,从自然资源中筛选抗氧化肽成为研究热点[7]。Bashir等[8]从日本鲭鱼肌肉蛋白中制备的抗氧化肽具有较好的1,1-二苯基-2-三硝基苯肼(1,1-diphenyl-2-picrylhydrazyl,DPPH)自由基清除活性,可作为抗氧化剂来源。白玉等[9]通过复合酶解法从牛骨中制备抗氧化肽,并对酶解工艺进行优化。Liu Qian等[10]采用碱性蛋白酶水解猪血浆蛋白,结果显示,酶解后的猪血浆蛋白抗氧化能力明显提高。
分子对接技术可通过模拟小分子多肽与受体蛋白间的结合模式和结合自由能筛选最佳配体[11]。李红梅等[12]采用分子对接技术研究抗氧化三肽SHW与Kelch样ECH相关蛋白1(Kelch-like ECH-associated protein 1,Keap1)的相互作用机制,发现SHW通过与Keap1蛋白活性口袋形成氢键和疏水相互作用影响Keap1-核转录因子E2相关因子2(nuclear factor erythroid 2 related factor 2,Nrf2)信号通路。Igbokwe等[13]利用分子对接技术从薏仁中筛选具有抗氧化活性的多肽,发现8 个肽段与Keap1-Nrf2具有良好的结合作用,其中FFDR肽段具有增强Keap1-Nrf2信号通路抗氧化基因表达能力。因此,通过分子对接技术深入研究抗氧化肽与Keap1的结合能力,有望从分子水平阐明多肽抗氧化活性的潜在作用机制。
本研究以猪骨粉为原料进行酶法制备抗氧化肽,以2,2’-联氮-双(3-乙基苯并噻唑啉-6-磺酸)(2,2’-azino-bis(3-ethylbenzothiazoline-6-sulfonic acid),ABTS)阳离子自由基与DPPH自由基清除率及总还原能力为筛选指标,采用超滤和凝胶色谱技术分离纯化,筛选抗氧化能力较强的组分,通过液相色谱-串联质谱(liquid chromatographytandem mass spectrometry,LC-MS/MS)鉴定多肽的氨基酸序列,并采用分子对接技术检测其与Keap1的结合能力,分析抗氧化作用机制,为进一步解决猪骨资源浪费及猪骨抗氧化肽的制备提供参考。
猪骨粉 西安本丰生物科技有限公司;碱性蛋白酶(200 U/mg)、风味蛋白酶(150 U/mg)、木瓜蛋白酶(>200 U/mg)、中性蛋白酶(100 U/mg)、胰蛋白酶(130 U/mg) 上海麦克林生化科技有限公司;ABTS、DPPH 美国Sigma公司。
SHA-CA水浴恒温振荡器 常州普天仪器制造有限公司;SN-MS-6D多联磁力搅拌器 上海尚普仪器设备有限公司;G1-21M低温高速离心机 上海安亭电子仪器有限公司;SCIENTZ-18N真空冷冻干燥机 宁波新芝生物科技股份有限公司;Easy-nLC1200 LC仪、电喷雾-组合型离子阱Orbitrap MS仪 美国Thermo Fisher Scientific公司。
1.3.1 猪骨抗氧化肽制备
参照张周莉[14]的方法并稍作修改。猪骨粉∶水=1∶1(m/m),调节至各蛋白酶最适pH值和温度,加入蛋白酶(5 000 U/g)进行酶解,酶解完成后调节pH值至7,100 ℃灭酶10 min,冷却,4 ℃、10 000 r/min离心10 min,取上清液冷冻干燥,即得猪骨抗氧化肽粗肽粉。
1.3.2 蛋白酶筛选
分别在木瓜蛋白酶、碱性蛋白酶、中性蛋白酶、风味蛋白酶和胰蛋白酶最适温度及pH值(表1)条件下进行酶解,固定底物质量浓度4 g/100 mL、酶解时间4 h、酶添加量5 000 U/g,以水解度(degree of hydrolysis,DH)、ABTS阳离子自由基清除率及多肽得率为指标筛选酶解效果最佳的蛋白酶。
表1 5 种蛋白酶最适水解条件
Table 1 Optimal hydrolysis conditions for five proteases

条件木瓜蛋白酶 碱性蛋白酶 中性蛋白酶 风味蛋白酶 胰蛋白酶温度/℃5050505037 pH710778
1.3.3 DH测定
参照GB 5009.5—2016《食品安全国家标准 食品中蛋白质的测定》测定总氮含量。氨基酸态氮含量测定参考Rao等[15]的方法并稍作修改。取5 mL猪骨酶解液与25 mL蒸馏水混匀,调节pH值至8.2,加入10 mL 37%(m/m)中性甲醛,采用0.05 mol/L NaOH标准溶液调节pH值至9.2,记录消耗的NaOH标准溶液体积(V1)。同时以蒸馏水为空白对照,记录消耗的NaOH标准溶液体积(V2)。按照式(1)计算氨基酸态氮含量,然后按照式(2)计算DH:

式中:ρ为NaOH标准溶液浓度/(mol/L);5为酶解液体积/mL。
1.3.4 DPPH自由基清除率测定
参照尹国友等[16]的方法,用95%(V/V)乙醇溶液配制0.20 mmol/L DPPH溶液,将100 μL DPPH溶液和100 μL样品溶液(1 mg/mL)混匀后,在室温条件下避光反应30 min,在517 nm处测定吸光度(A1);对照组采用100 μL 95%(V/V)乙醇溶液代替DPPH溶液,测定吸光度(A2);空白组采用100 μL蒸馏水代替样品溶液,测定吸光度(A0)。按照式(3)计算DPPH自由基清除率:
1.3.5 ABTS阳离子自由基清除率测定
参考Carrillo等[17]的方法并稍作修改。将7 mmol/L ABTS试剂与等体积的2.45 mmol/L过硫酸钾溶液混合为ABTS母液,4 ℃避光条件下反应24 h,备用。使用前采用去离子水稀释ABTS母液至734 nm处吸光度达0.70±0.02,即得ABTS工作液。将20 μL样品溶液和180 μL ABTS母液混匀,室温下避光反应5 min后在734 nm处测定吸光度(A1),等体积蒸馏水代替样品溶液为空白组,测定吸光度(A0)。按照式(4)计算ABTS阳离子自由基清除率:
1.3.6 总还原能力测定
参考Xiao Fan等[18]的方法并加以修改。将1 mL样品溶液(1 mg/mL)与等体积0.2 mol/L磷酸盐缓冲液和10 mg/mL铁氰化钾溶液混匀,50 ℃水浴20 min,加入1 mL 10 g/100 mL三氯乙酸溶液,8 000 r/min离心10 min,取上清液2 mL,加入1 mL去离子水和0.5 mL 0.1 g/100 mL氯化铁溶液,在700 nm处测定吸光度,吸光度越大表明其总还原能力越强。
1.3.7 多肽得率测定
参考沈晓勇等[19]的方法,取1 mL酶解液与等体积15 g/100 mL三氯乙酸溶液混匀,静置10 min后8 000 r/min离心5 min,采用双缩脲法测定上清液中的蛋白含量。按照式(5)计算多肽得率:
1.3.8 单因素试验
根据预实验及文献报道,考察酶解温度(40、45、50、55、60 ℃)、酶解pH(6.00、6.50、7.00、7.50、8.00)、加酶量(2 000、4 000、6 000、8 000、10 000 U/g)、酶解时间(2、3、4、5、6 h)对酶解液ABTS阳离子自由基清除率、DH的影响。
1.3.9 响应面分析
在单因素试验的基础上,以ABTS阳离子自由基清除率为指标,利用Design-Expert v8.0.6.1软件,根据Box-Behnken设计原理对酶解工艺进行优化,得到制备抗氧化肽的最佳酶解条件,试验因素水平见表2。
表2 Box-Behnken试验中变量水平
Table 2 Levels of variables used for Box-Behnken design
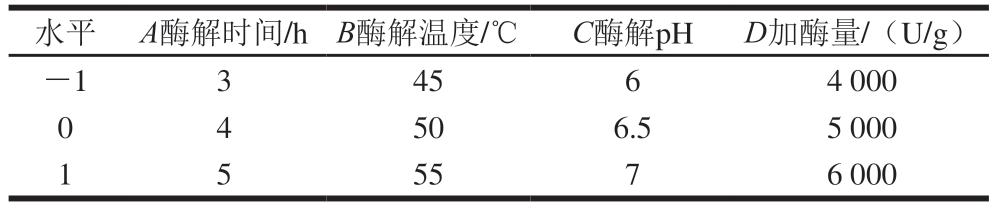
水平 A酶解时间/h B酶解温度/℃C酶解pHD加酶量/(U/g)-134564 000 0 4 506.55 000 1 5 5576 000
1.3.10 超滤分离
采用分子质量为10 kDa和3 kDa的超滤管对猪骨酶解液进行超滤,得到分子质量范围为>10 kDa、3~10 kDa、<3 kDa 3 个组分,将收集到的各组分粗肽液进行冷冻干燥备用。
1.3.11 凝胶色谱分离
参考Feng Yanxia等[20]的方法稍作修改。采用Sephadex G-25柱进行凝胶层析。将超滤后抗氧化活性最强的组分溶解于超纯水,经0.22 μm水系滤膜过滤,加入2 mL样品后用去离子水洗脱,每5 min收集1 次,在280 nm处测定吸光度,室温下采用自动收集器收集滤液,并根据色谱峰混合后冷冻干燥。
1.3.12 LC-MS/MS鉴定
委托北京百泰派克生物科技有限公司对上述所得抗氧化活性最好的组分进行肽段鉴定。质谱测试原始文件采用PEAKS Studio 10.6软件进行分析,得到肽段序列结果。
LC条件:洗脱流速600 nL/min;洗脱缓冲液由A相(0.1%(V/V)甲醇溶液)和B相(0.2%(V/V)甲醇乙腈溶液)组成;洗脱条件:0~2 min,8% B;2~45 min,8%~28% B;45~55 min,28%~40% B;55~56 min,40%~95% B;56~66 min,95% B。
MS条件:一级质谱条件:分辨率70 000,最大注射时间100 ms,自动增益控制目标3×106;二级质谱条件:分辨率17 500,最大注射时间50 ms,自动增益控制目标1×105。
1.3.13 抗氧化肽筛选
利用PeptideRanker对LC-MS/MS鉴定的肽段进行生物活性评分,评分越高,表明肽段的抗氧化活性越强。使用PepDraw工具预测肽段的净电荷和疏水性,Innovagen工具预测肽段的相对分子质量,AnOxPePred 1.0工具(https://services.healthtech.dtu.dk/services/AnOxPePred-1.0/)预测活性肽的抗氧化活性[21]。
1.3.14 分子对接
通过检索PDB(Protein Data Bank)数据库(https://www.rcsb.org/)获取Keap1(PDB ID:4L7B)的蛋白晶体结构,使用PyMOL 2.3.0去除蛋白结晶水、氢原子及原有受体所携带的配体,利用Chem Draw 7.0构建多肽3D结构并进行能量最小化,将小分子导入到AutoDock工具进行加全氢、计算电荷等前处理,采用AutoDock Vina 1.1.2进行对接,然后通过Discovery Studio 2019和LigPlot v2.2.8将对接结果进行可视化分析。
各组实验均进行3 次平行,数据表示为平均值±标准差,通过SPSS 17.0软件进行数据处理,P<0.05表示差异显著,采用Design-Expert v8.0.6.1软件进行响应面优化分析,并采用Origin 2019b软件制图。
酶可以将蛋白质水解成小分子物质,DH是衡量酶解效果的重要指标之一[22]。不同蛋白酶作用于同一底物时,因酶切位点差异可导致酶解后多肽结构各有特点,因此不同蛋白酶酶解所得多肽的抗氧化活性存在差异[23]。由图1可知,5 种蛋白酶作用于底物时,DH均随酶解时间延长而增大,酶解结束时,各组DH存在较大差异,风味蛋白酶组DH最高,达到(32.93±0.26)%。同样,风味蛋白酶酶解产物的多肽得率和ABTS阳离子自由基清除率显著高于其他组(P<0.05),分别为(51.39±0.72)%和(80.04±0.39)%,其次为碱性蛋白酶和中性蛋白酶,胰蛋白酶酶解效果最差。Wang Jiao等[24]利用风味蛋白酶对金枪鱼蛋白进行酶解,其DPPH自由基清除率显著高于其他组,与本研究结果较为一致。因此,选择风味蛋白酶进行后续研究。
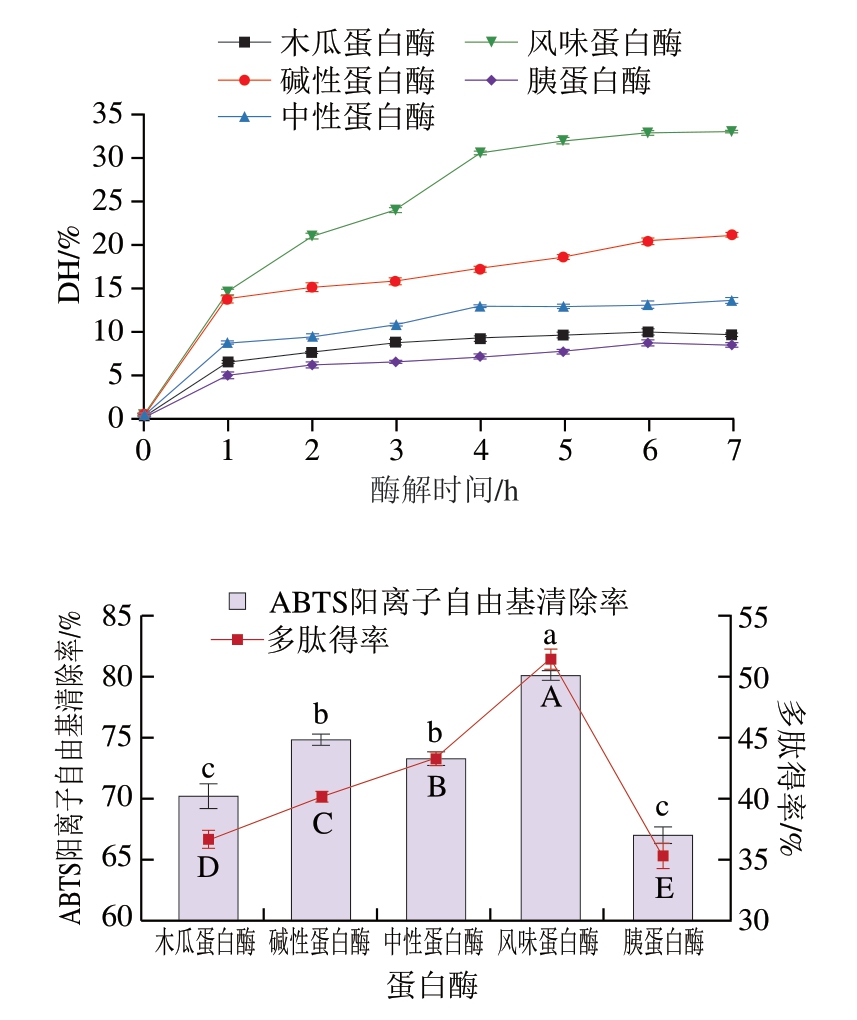
图1 5 种蛋白酶对DH、多肽得率及ABTS阳离子自由基清除率的影响
Fig.1 Effect of five proteases on degree of hydrolysis, peptide yield and ABTS anionic radical scavenging capacity
字母不同表示组间差异显著(P<0.05)。图2同。
如图2所示,随着酶解温度的升高,DH和ABTS阳离子自由基清除率呈先增高后下降的变化趋势,这是由于温度对蛋白酶活性产生影响[25]。酶解温度为50 ℃时,ABTS阳离子自由基清除率达到最大值81.33%。因此选择酶解温度为50 ℃。
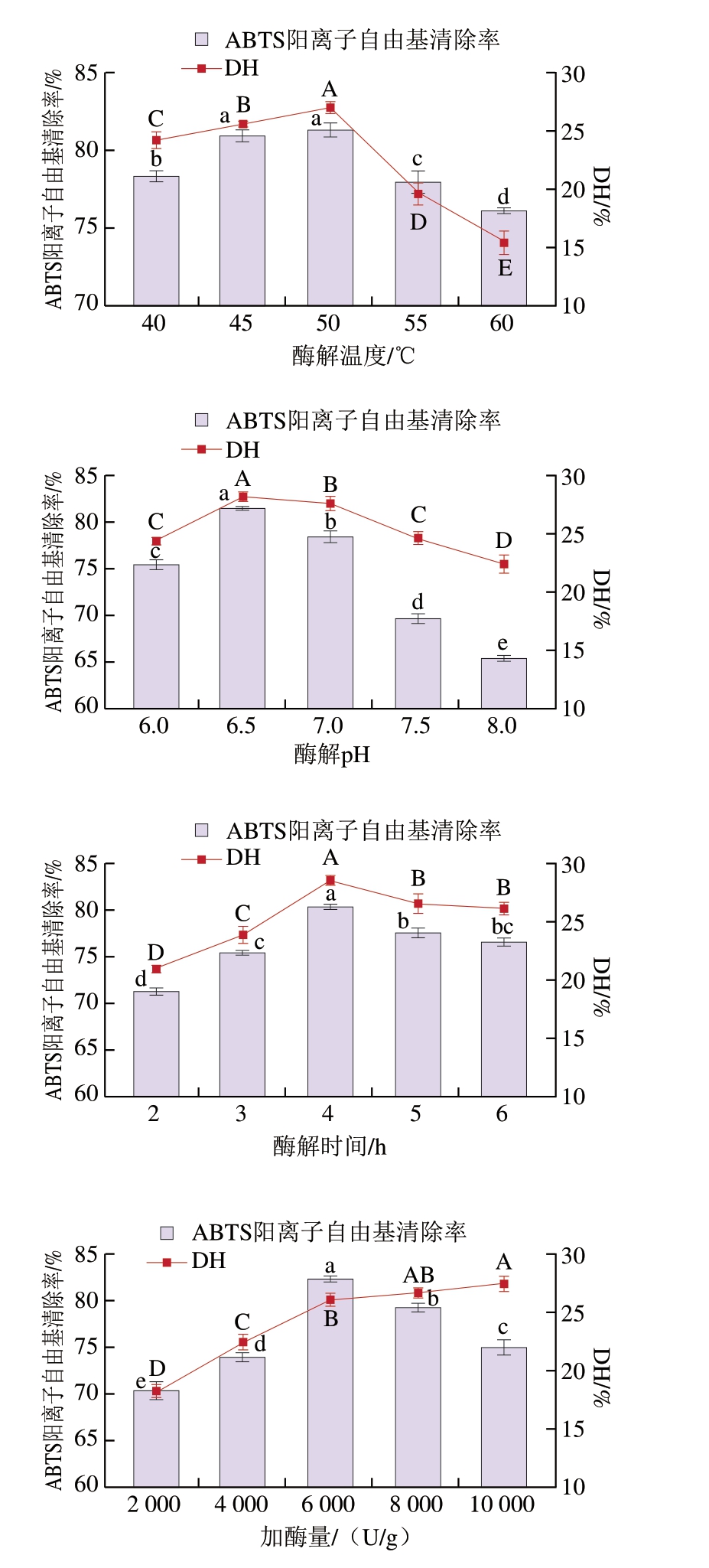
图2 不同酶解条件对酶解产物ABTS阳离子自由基清除率和DH的影响
Fig.2 Effect of different enzymatic conditions on hydrolysis degree and ABTS anionic radical scavenging capacity of hydrolysate
随着pH值的增加,DH和ABTS阳离子自由基清除率也呈先升后降的趋势,当pH 6.5时,DH和ABTS阳离子自由基清除率分别达到最大值,这可能是由于风味蛋白酶在弱酸环境下活性更高,而pH值升高会导致蛋白变性,进而降低酶解效率[26]。因此选择pH值为6.5。
酶解时间为4 h时,ABTS阳离子自由基清除率达到最大值。随着酶解反应的进行,底物中可切割结合位点减少,延长酶解时间并不能显著提高DH及ABTS阳离子自由基清除率[27]。因此选择酶解时间为4 h。
随着加酶量的增大,ABTS阳离子自由基清除率呈先升后降趋势,当加酶量达到6 000 U/g后,DH增幅变缓,可能是因为结合位点趋于饱和,继续增加酶量并不能大幅提高DH[28]。因此选择加酶量为6 000 U/g。
在单因素试验的基础上,选择酶解时间(A)、酶解温度(B)、酶解pH值(C)及加酶量(D)4 个因素作为自变量,根据Box-Behnken试验设计原则建立4因素3水平试验。试验设计与结果见表3。
表3 Box-Behnken试验设计及结果
Table 3 Box-Behnken design with experimental results
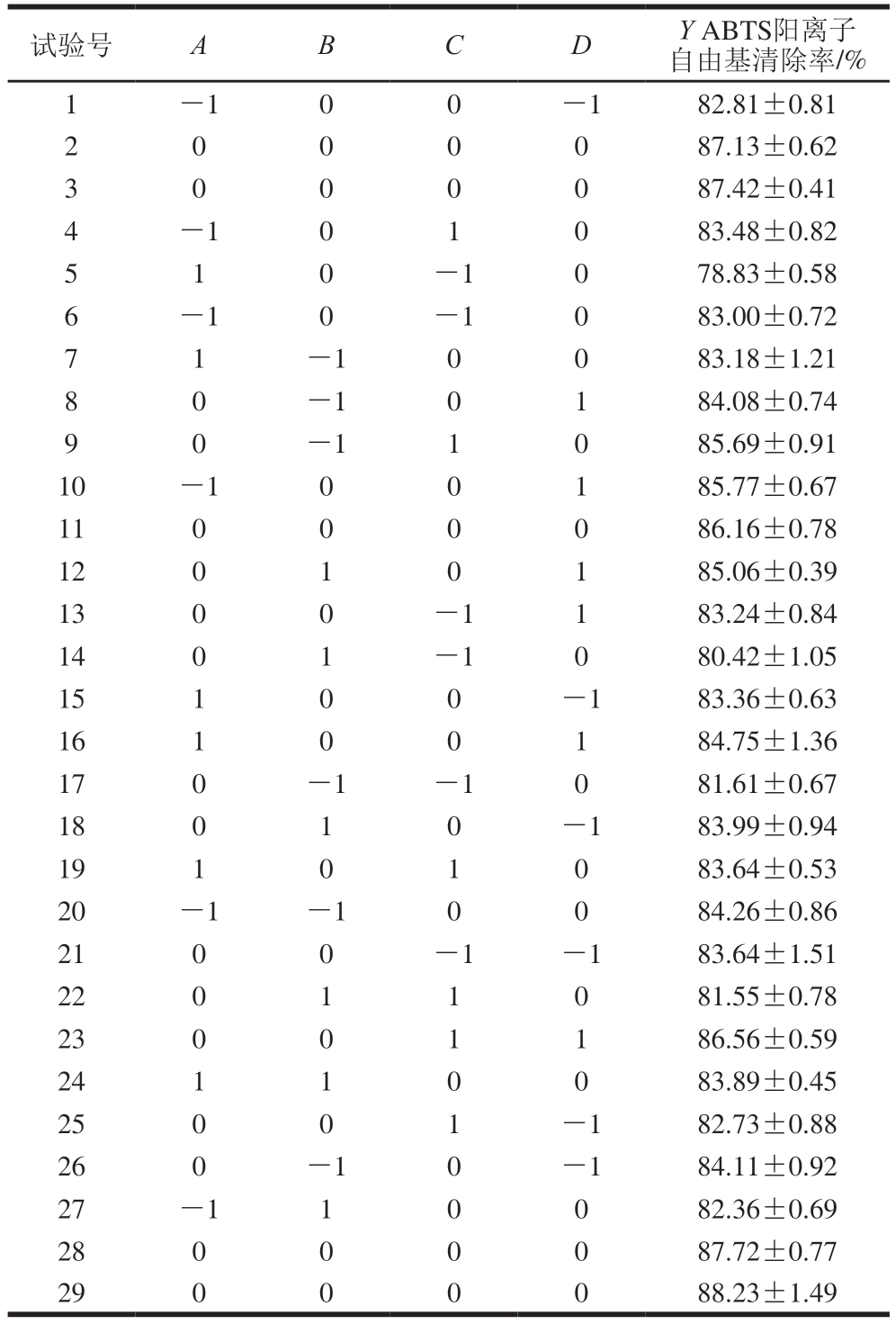
试验号ABCDY ABTS阳离子自由基清除率/%1-100-182.81±0.81 2000087.13±0.62 3000087.42±0.41 4-101083.48±0.82 510-1078.83±0.58 6-10-1083.00±0.72 71-10083.18±1.21 80-10184.08±0.74 90-11085.69±0.91 10-100185.77±0.67 11000086.16±0.78 12010185.06±0.39 1300-1183.24±0.84 1401-1080.42±1.05 15100-183.36±0.63 16100184.75±1.36 170-1-1081.61±0.67 18010-183.99±0.94 19101083.64±0.53 20-1-10084.26±0.86 2100-1-183.64±1.51 22011081.55±0.78 23001186.56±0.59 24110083.89±0.45 25001-182.73±0.88 260-10-184.11±0.92 27-110082.36±0.69 28000087.72±0.77 29000088.23±1.49
运用Design Expert v8.0.6软件,通过拟合进行二次多元回归分析,得到回归方程为:Y=87.35-0.34A-0.47B+1.10C+0.71D+0.65AB+1.08AC-0.39AD-0.74BC+0.28BD+0.99CD-2.19A2-2.08B2-2.77C2-0.81D2。响应面分析结果如表4所示,二次多元回归方程失拟项不显著(P=0.283 2>0.05),回归模型显著(P<0.000 1),说明模型与实际结果拟合程度较好。回归相关系数R2=0.893 4,表明模型拟合度较好。各因素对ABTS阳离子自由基清除率的影响顺序为:酶解pH值>加酶量>酶解温度>酶解时间。
表4 ABTS阳离子自由基清除率回归模型方差分析
Table 4 Analysis of variance of regression model for ABTS anionic radical scavenging capacity
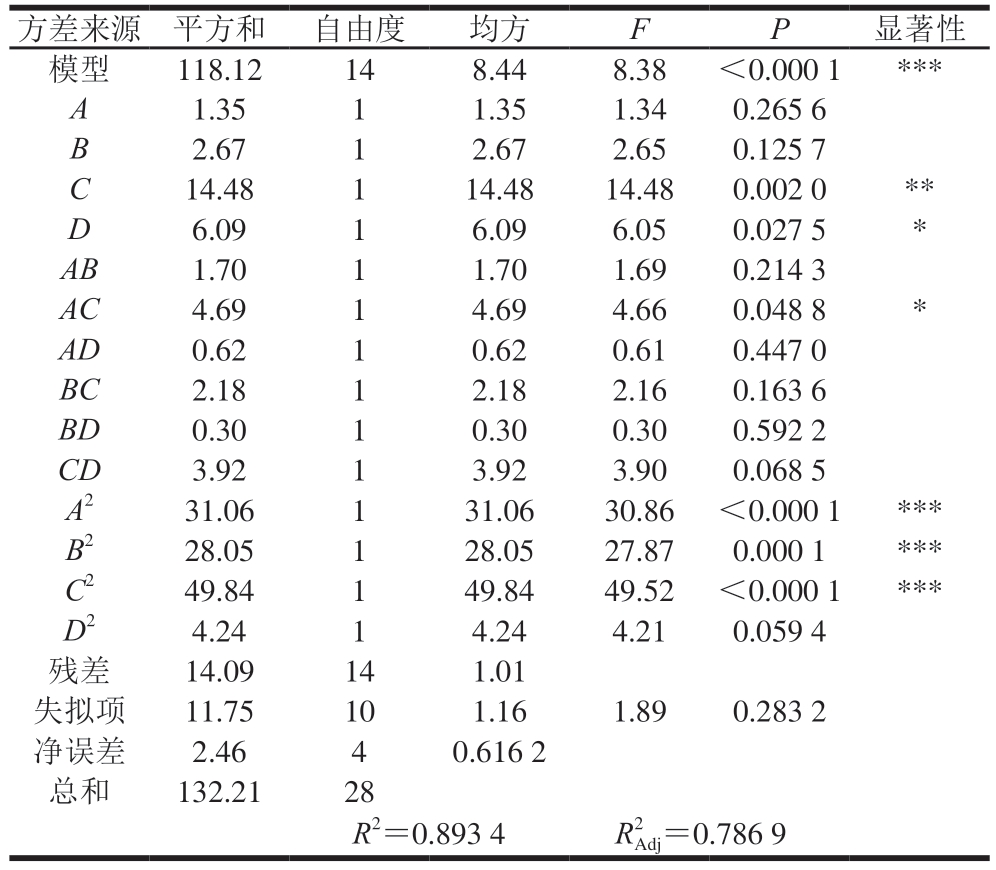
注:*.显著(P<0.05);**.极显著(P<0.01);***.高度显著(P<0.001)。
方差来源 平方和 自由度均方FP显著性模型118.12148.448.38<0.000 1***A1.3511.351.340.265 6 B2.6712.672.650.125 7 C14.48114.4814.480.002 0**D6.0916.096.050.027 5*AB1.7011.701.690.214 3 AC4.6914.694.660.048 8*AD0.6210.620.610.447 0 BC2.1812.182.160.163 6 BD0.3010.300.300.592 2 CD3.9213.923.900.068 5 A231.06131.0630.86 <0.000 1***B228.05128.0527.870.000 1***C249.84149.8449.52 <0.000 1***D24.2414.244.210.059 4残差14.09141.01失拟项11.75101.161.890.283 2净误差2.4640.616 2总和132.2128 R2=0.893 4 R2 Adj=0.786 9
等高线趋于椭圆表示两因素交互作用显著,等高线趋于圆形则表示交互作用不显著[29],响应曲面陡峭程度反映酶解条件对ABTS阳离子自由基清除率的影响。由图3可知,酶解时间和酶解pH值的响应面陡峭程度最大,表明其交互作用最显著。求解回归方程可得最佳酶解工艺条件为:酶解温度49.86 ℃、酶解时间3.92 h 、pH 6.82、加酶量6 000.65 U/g,此时ABTS阳离子自由基清除率预测值为87.79%。根据实际操作将酶解工艺条件调整为:酶解温度50 ℃、酶解时间4 h、pH 6.80、加酶量6 000 U/g,此条件下,ABTS阳离子自由基清除率为(88.03±0.62)%,与预测值较为接近。
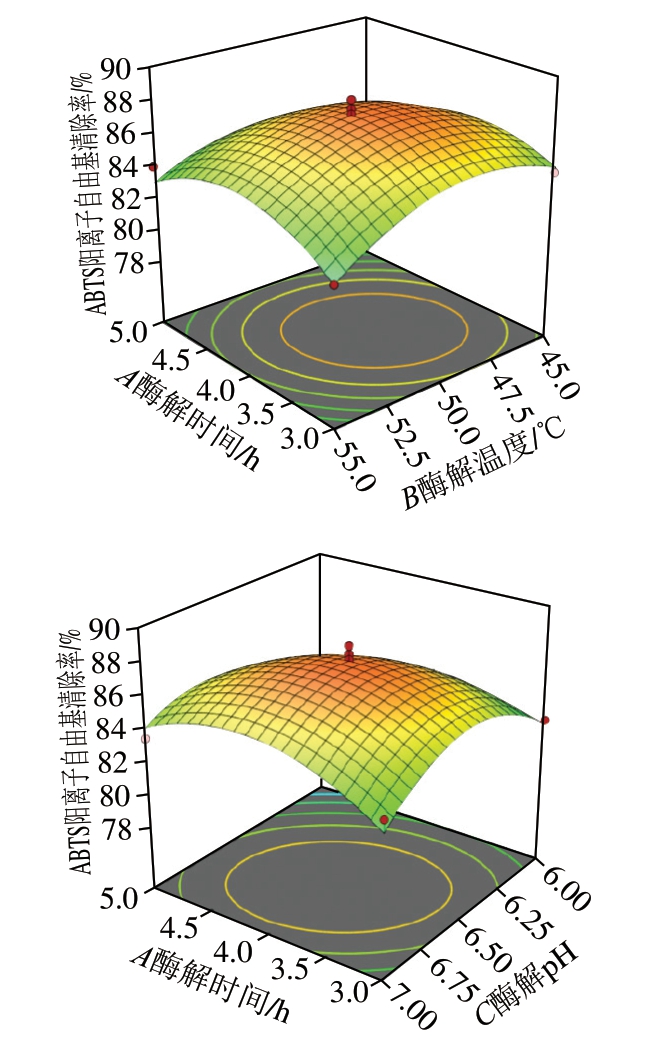
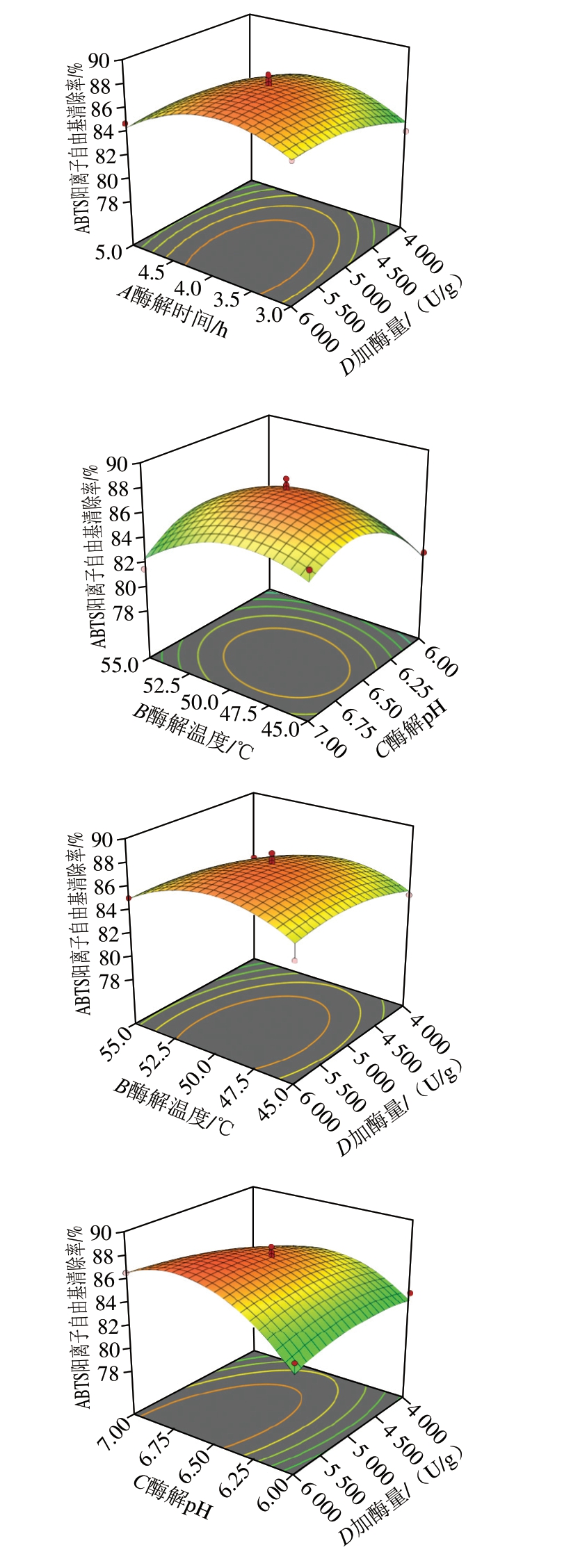
图3 各因素交互作用对ABTS阳离子自由基清除率的响应面图
Fig.3 Response surface plots showing interactive effects of variables on ABTS anionic radical scavenging capacity
酶解液经超滤分离得到3 个组分,分别为分子质量>10 kDa、3~10 kDa及<3 kDa。如表5所示,分子质量<3 kDa组分抗氧化活性最强,其ABTS阳离子自由基清除率、DPPH自由基清除率和总还原能力分别为(92.83±0.53)%、(74.10±0.81)%和0.09±0.03。分子质量较小的多肽抗氧化活性更高可能是由于小分子肽因体积小更易被人体吸收,或更易与自由基发生反应,进而促进自由基转化为更稳定的物质[30]。Phongthai等[31]将米糠蛋白水解物分别通过3、5、10 kDa超滤膜,也发现分子质量<3 kDa组分具有最好的DPPH自由基和ABTS阳离子自由基清除活性;Wali等[32]从蝎蛋白中提取抗氧化肽,也发现超滤所得中分子质量<3 kDa组分具有较高的抗氧化活性;蔡金秀等[33]采用超滤法分离马面鱼皮胶原蛋白抗氧化肽,得到的3 个组分抗氧化活性由大到小分别为<5 kDa、5~10 kDa、>10 kDa组分,以上结果均与本研究结果相一致。因此,选择<3 kDa猪骨酶解物组分进行下一步的葡聚糖凝胶分离纯化。
表5 不同超滤组分的抗氧化活性
Table 5 Antioxidant activity of different ultrafiltration fractions
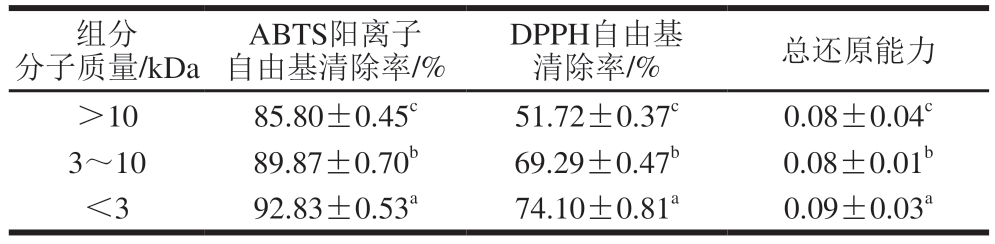
注:同列小写字母不同表示组间差异显著(P<0.05)。表6同。
组分分子质量/kDa ABTS阳离子自由基清除率/%DPPH自由基清除率/%总还原能力>1085.80±0.45c51.72±0.37c0.08±0.04c 3~1089.87±0.70b69.29±0.47b0.08±0.01b<392.83±0.53a74.10±0.81a0.09±0.03a
凝胶色谱根据被分离物质的分子质量实现分离纯化,小分子物质能够进入凝胶微孔中,而大分子物质直接通过填料的间隙而被洗脱下来[34]。将分子质量<3 kDa猪骨抗氧化粗肽进行葡聚糖凝胶分离,其在Sephadex G-25柱上的洗脱曲线如图4所示。经凝胶分离后,收集到3 个组分,分别记为F-1、F-2、F-3,峰尖分别在46、88、120 min出现,其中F-1组分出峰时间早,其分子质量最大,而F-3组分出峰时间最晚,分子质量最小。
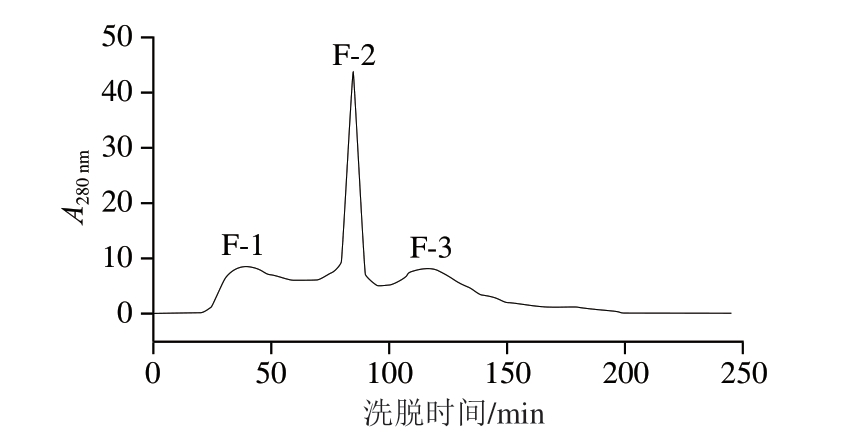
图4 超滤<3 kDa组分的Sephadex G-25凝胶色谱图
Fig.4 Sephadex G-25 chromatogram of ultrafiltration fraction < 3 kDa
如表6所示,F-2组分抗氧化活性最高,其ABTS阳离子自由基清除率为(94.57±1.08)%、DPPH自由基清除率为(79.08±0.61)%、总还原能力为1.24±0.06;其次是F-3组分。由此可见,多肽的抗氧化活性与其分子质量有关,但分子质量越小的多肽其抗氧化活性不一定越强,这与Wu Huichun等[35]的研究结果一致,鲭鱼蛋白酶解液中分子质量1 400 Da多肽的抗氧化活性高于分子质量900 Da和200 Da多肽。Xing Lujuan等[36]通过葡聚糖凝胶色谱分离宣威火腿多肽得到的4 个组分中,第3个组分的羟自由基清除率为35.06%,显著高于其他组分。在葡聚糖凝胶柱中保留时间最长的组分其抗氧化活性较弱可能是由于洗脱液中存在大量游离氨基酸和无抗氧化活性的小肽[37],这也可能是F-3组分抗氧化活性较差的原因。综上,选择F-2组分对其进行进一步结构鉴定与活性预测。
表6 Sephadex G-25凝胶分离组分的抗氧化活性
Table 6 Antioxidant activity of Sephadex G-25 gel separation fractions
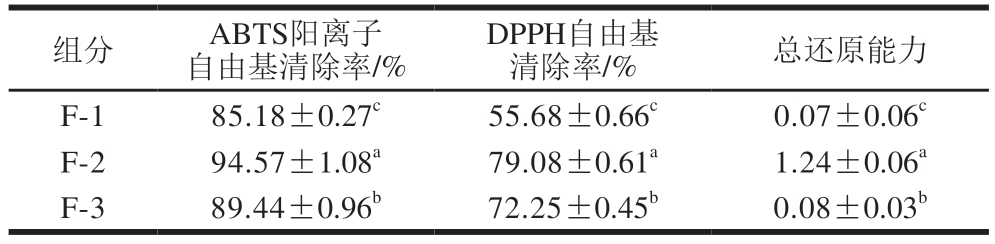
组分ABTS阳离子自由基清除率/%DPPH自由基清除率/%总还原能力F-185.18±0.27c55.68±0.66c0.07±0.06c F-294.57±1.08a79.08±0.61a1.24±0.06a F-389.44±0.96b72.25±0.45b0.08±0.03b
采用LC-MS/MS从F-2组分中共鉴定出232 条多肽序列,肽段分子质量在565~2 234 Da之间。根据PeptideRanker肽段评分结果筛选得到3 条肽段,分别为Gly-Pro-Val-Gly-Arg-Arg-Gly-Pro、Gly-Arg-Pro-Gly-Pro-Val-Gly-Pro、Gln-Pro-Gly-Pro-Arg-Gly-Pro。多肽的抗氧化活性与其分子质量、氨基酸组成及疏水性密切相关[38]。如表7所示,3 条多肽分子质量均较低,由7~8 个氨基酸组成,与Ma等[39]研究结果相一致。
表7 3 条抗氧化肽氨基酸序列及活性预测
Table 7 Amino acid sequence and activity prediction of three antioxidant peptides
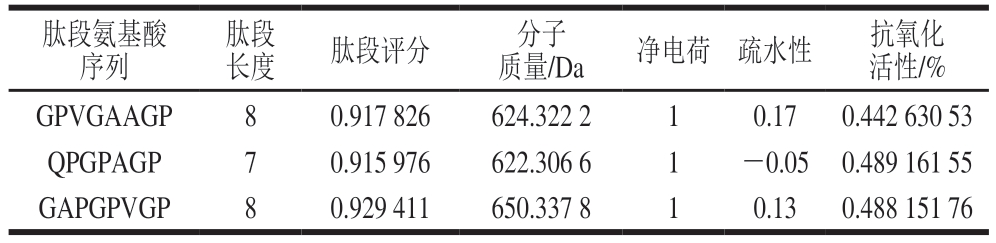
质量/Da净电荷 疏水性抗氧化活性/%GPVGAAGP80.917 826624.322 210.170.442 630 53 QPGPAGP70.915 976622.306 61-0.05 0.489 161 55 GAPGPVGP80.929 411650.337 810.130.488 151 76肽段氨基酸序列长度肽段评分分子肽段
多肽的生物活性与其所含特定氨基酸的比例和氨基酸位置有关[40]。例如,疏水性氨基酸比例较高的多肽具有较强的抗氧化活性,因为疏水性氨基酸有利于提高肽段在脂质体系中的溶解度,并且能够加强肽段与脂溶性自由基相互作用[41]。Mendis等[42]从巨型鱿鱼皮明胶中分离抗氧化肽时也发现,疏水性氨基酸含量高的多肽抗氧化活性更好。同样地,Agrawal等[43]发现珍珠粟蛋白抗氧化肽的高抗氧化活性与其多肽序列中的疏水性氨基酸Gly、Leu和Pro有关。Ranathunga等[44]发现,星康吉鳗(Conger myriaster)蛋白水解物中纯化的抗氧化肽由Leu、Gly和Val等疏水性氨基酸残基组成,约占序列的55%,表明疏水性氨基酸占比对其抗氧化作用影响较大。综上,疏水性氨基酸比例影响多肽抗氧化活性,GPVGAAGP、GAPGPVGP和QPGPAGP 3 条多肽的疏水性氨基酸占比均在80%以上,推测肽段中的Gly、Pro、Val、Arg可能通过促进其与脂质分子相互作用清除自由基,表现出较高的抗氧化活性。
为进一步从分子水平探究多肽的抗氧化作用机制,将GPVGAAGP、GAPGPVGP和QPGPAGP分别与Keap1蛋白进行分子对接模拟[45]。研究[46]表明,抗氧化肽可以直接作用于Keap1-Nrf2结合位点,与Keap1蛋白结合并释放Nrf2进入细胞核,进而启动抗氧化基因表达。结合能小于0说明小分子可以自由结合靶蛋白,并且结合能越小,越可能与靶蛋白结合[47]。由表8可知,GPVGAAGP、GAPGPVGP、QPGPAGP对Keap1蛋白的结合能分别为-2.76、-5.36、-1.96 kJ/mol,表明3 条多肽能够自发地与Keap1蛋白结合。
表8 3 条抗氧化肽与Keap1的分子对接分析
Table 8 Molecular docking analysis of three antioxidant peptides with Keap1 protein
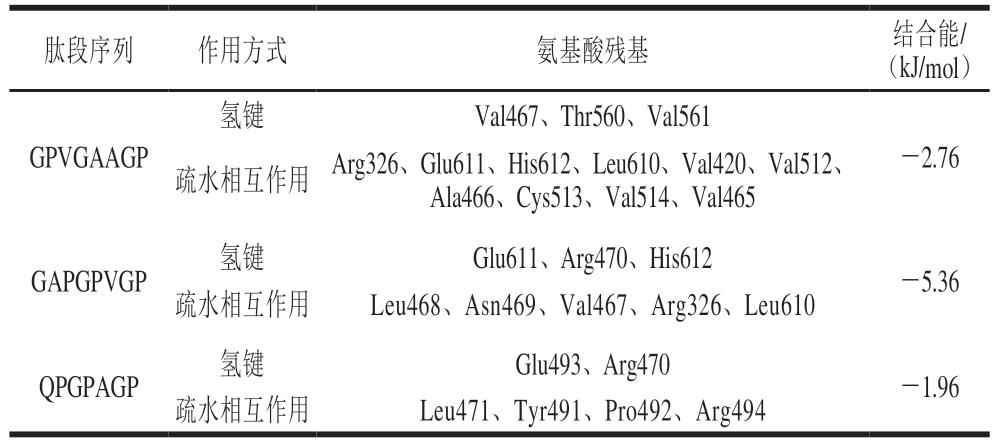
肽段序列作用方式氨基酸残基结合能/(kJ/mol)氢键Val467、Thr560、Val561 GPVGAAGP -2.76疏水相互作用 Arg326、Glu611、His612、Leu610、Val420、Val512、Ala466、Cys513、Val514、Val465 GAPGPVGP氢键Glu611、Arg470、His612-5.36疏水相互作用Leu468、Asn469、Val467、Arg326、Leu610 QPGPAGP氢键Glu493、Arg470-1.96疏水相互作用Leu471、Tyr491、Pro492、Arg494
3 条抗氧化肽与Keap1蛋白结合方式和结合残基如图5所示。GPVGAAGP与Val467、Thr560、Val561残基形成3 个氢键,长度分别为2.85、3.04、2.88 Å,与Arg326、Glu611、His612、Leu610等10 个残基产生疏水相互作用;GAPGPVGP与His612、Glu611、Arg470残基形成3 个氢键,长度分别为3.24、3.03、2.99 Å,存在5 种疏水相互作用;QPGPAGP与Arg470、Glu493残基形成2 个氢键,长度分别为2.78、2.92 Å,存在4 种疏水相互作用,氢键和疏水相互作用对复合物形成至关重要[48]。QPGPAGP和GAPGPVGP与Keap1蛋白在Arg470形成共同的氢键结合位点。分子对接结果表明,3 条抗氧化肽均可能与Keap1蛋白结合释放Nrf2,从而发挥抗氧化活性。
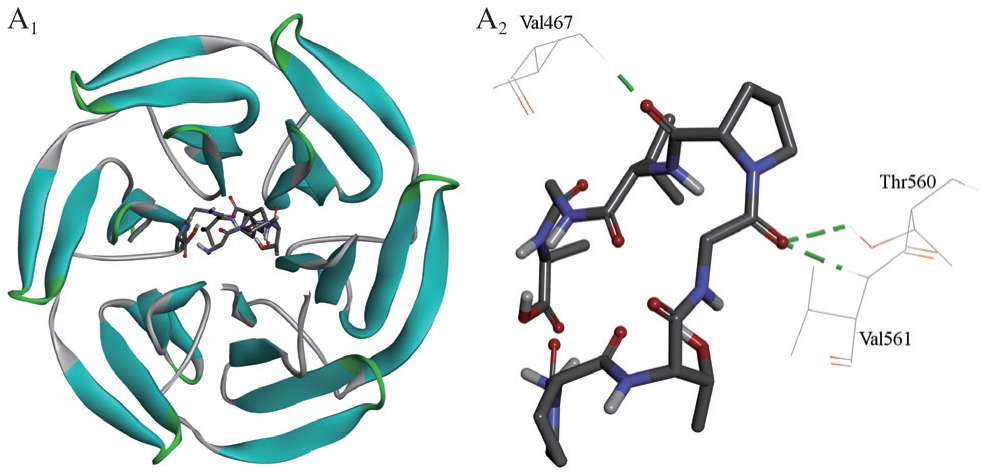
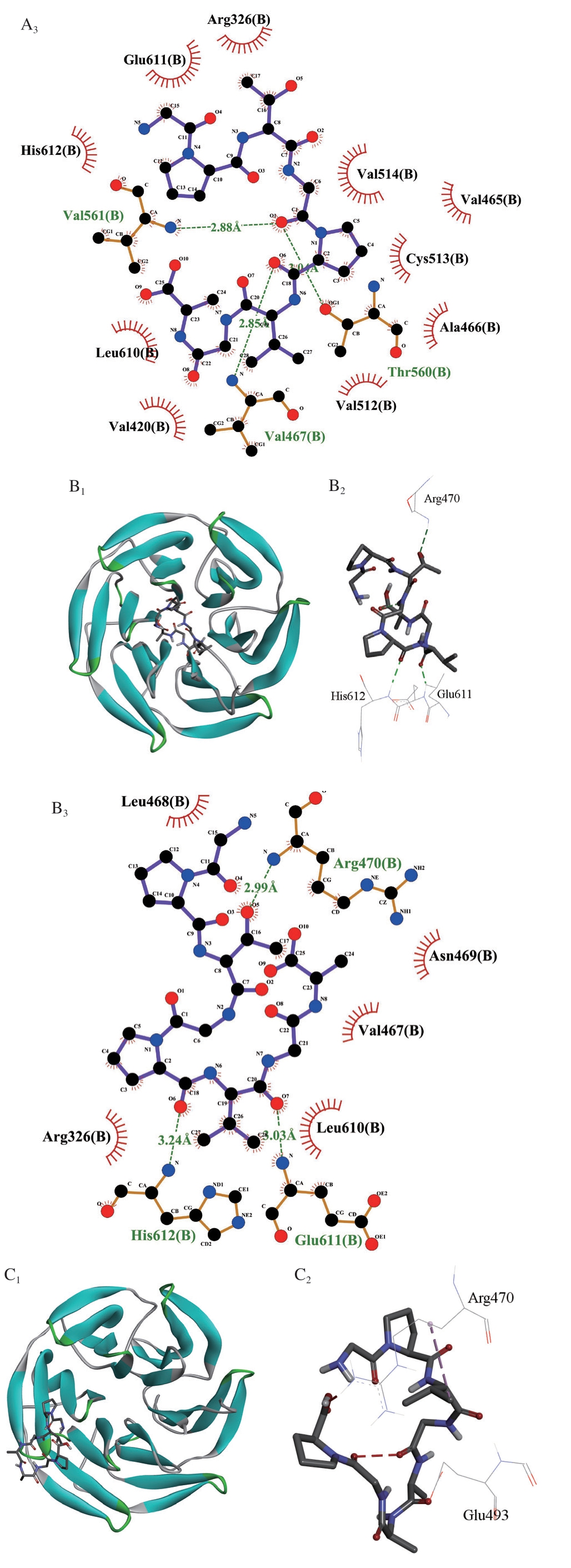
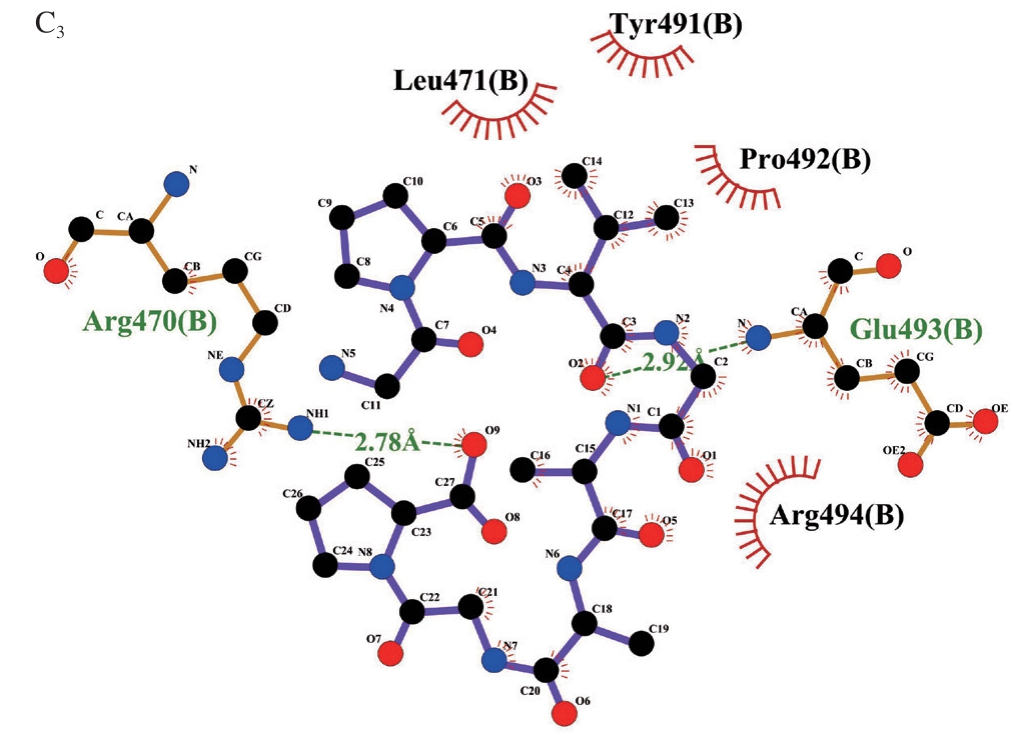
图5 3 条抗氧化肽与Keap1蛋白分子对接图
Fig.5 Molecular docking diagrams between three antioxidant peptides and Keap1 protein
A1~A2、B1~B2、C1~C2.分别为GPVGAAGP、GAPGPVGP、QPGPAGP和Keap1蛋白结合相互作用图;A3、B3、C3.分别为GPVGAAGP、GAPGPVGP、QPGPAGP和Keap1蛋白的氢键和疏水相互作用图。
本研究以ABTS阳离子自由基清除率、DH等为指标确定制备猪骨抗氧化肽的最佳工艺条件,在此工艺条件下,猪骨抗氧化肽ABTS阳离子自由基清除率为88.03%。通过超滤和凝胶色谱分离得到3 个组分(F-1、F-2、F-3),其中F-2组分具有较强的抗氧化活性,ABTS阳离子自由基清除率为94.57%,采用LC-MS/MS从F-2组分中鉴定得到232 条肽段。通过PeptideRanker筛选得到GPVGAAGP、GAPGPVGP和QPGPAGP肽段,其分子质量分别为624.32、650.34、622.31 Da,疏水性氨基酸占比均在80%以上。分子对接结果表明,3 条多肽与Keap1蛋白通过氢键和疏水相互作用产生较好结合能力,说明其具备较强的抗氧化活性。由于分子对接是一种虚拟筛选方法,后续可以通过细胞实验进一步验证多肽的抗氧化活性,深入研究其抗氧化作用机制。
[1] SHEN X, ZHANG M, BHANDARI B.Novel technologies in utilization of byproducts of animal food processing: a review[J].Critical Reviews in Food Science and Nutrition, 2019, 59(21): 3420-3430.DOI:10.1080/10408398.2018.1493428.
[2] 王丽媛, 高艳蕾, 张丽, 等.畜禽副产物的加工利用现状及研究展望[J].食品科技, 2022, 47(6): 174-183.DOI:10.13684/j.cnki.spkj.2022.06.017.
[3] 安玉会, 杨松华, 晋佳路, 等.猪骨蛋白的氨基酸含量分析[J].河南医学研究, 2004(3): 208-209.DOI:10.3969/j.issn.1004-437X.2004.03.003.
[4] 唐宏刚, 肖朝耿, 叶梦迪, 等.猪骨蛋白酶解液美拉德反应产物的抗氧化活性研究[J].浙江农业学报, 2016, 28(8): 1396-1400.DOI:10.3969/j.issn.1004-1524.2016.08.18.
[5] 孔玉婷.猪骨汤胞内抗氧化活性评价模型的研究[D].杭州: 浙江工商大学, 2016: 17-22.
[6] 张强, 李伟华.抗氧化肽的研究现状[J].食品与发酵工业, 2021,47(2): 298-304.DOI:10.13995/j.cnki.11-1802/ts.024999.
[7] 左依瑾, 于子淇, 申雪晴, 等.鱼源抗氧化肽的结构与其功能关系研究进展[J].食品工业科技, 2022, 44(14): 419-429.DOI:10.13386/j.issn1002-0306.2022080200.
[8] BASHIR K M I, SOHN J H, KIM J S, et al.Identification and characterization of novel antioxidant peptides from mackerel (Scomber japonicus) muscle protein hydrolysates[J].Food Chemistry, 2020, 323:126809.DOI:10.1016/j.foodchem.2020.126809.
[9] 白玉, 周中驰, 陈飞, 等.牛骨多肽酶解工艺优化及其抗氧化活性研究[J].食品工业, 2024, 45(1): 41-46.DOI:10.3969/j.issn.1004-471X.spgy202401011.
[10] LIU Q, KONG B H, JIANG L Z, et al.Free radical scavenging activity of porcine plasma protein hydrolysates determined by electron spin resonance spectrometer[J].LWT-Food Science and Technology, 2009,42(5): 956-962.DOI:10.1016/j.lwt.2008.12.007.
[11] YANG G L, QIN S, LI W J.Purification and characterization of a novel angiotensin I-converting enzyme-inhibitory peptide derived from Alaska pollack skins[J].Journal of Food Science, 2021, 86(6): 2457-2467.DOI:10.1111/1750-3841.15754.
[12] 李红梅, 梁振, 吴家颖, 等.抗氧化肽SHW作用于Keap1-Nrf2通路的分子动力学模拟[J].原子与分子物理学报, 2023, 40(4): 58-64.DOI:10.19855/j.1000-0364.2023.041008.
[13] IGBOKWE C J, FENG Y Q, LOUIS H, et al.Novel antioxidant peptides identified from coix seed by molecular docking, quantum chemical calculations and in vitro study in HepG2 cells[J].Food Chemistry,2024, 440: 138234.DOI:10.1016/J.FOODCHEM.2023.138234.
[14] 张周莉.酶解法制备猪肩胛骨抗氧化肽及其分离纯化的研究[D].雅安: 四川农业大学, 2020: 17-20.
[15] RAO P S, BAJAJ R, MANN B.Impact of sequential enzymatic hydrolysis on antioxidant activity and peptide profile of casein hydrolysate[J].Food Science and Technology, 2020, 57(12): 4562-4575.DOI:10.1007/s13197-020-04495-2.
[16] 尹国友, 孙婕, 澹博, 等.双水相萃取韭籽粕多糖的工艺优化及其抗氧化活性研究[J].食品科学技术学报, 2021, 39(2): 134-142.DOI:10.12301/j.issn.2095-6002.2021.02.017.
[17] CARRILLO C, WILCHES-PÉREZ D, HALLMANN E, et al.Organic versus conventional beetroot.Bioactive compounds and antioxidant properties[J].LWT-Food Science and Technology, 2019, 116: 108552.DOI:10.1016/j.lwt.2019.108552.
[18] XIAO F, XU T, LU B Y, et al.Guidelines for antioxidant assays for food components[J].Food Frontiers, 2020, 1(1): 60-69.DOI:10.1002/fft2.10.
[19] 沈晓勇, 林峰, 马涛, 等.双酶法制备饲料级小麦低聚肽的工艺研究[J].饲料研究, 2023, 46(5): 65-69.DOI:10.13557/j.cnki.issn1002-2813.202123.05.014.
[20] FENG Y X, WANG Z C, CHEN J X, et al.Separation, identification,and molecular docking of tyrosinase inhibitory peptides from the hydrolysates of defatted walnut (Juglans regia L.) meal[J].Food Chemistry, 2021, 353: 129471.DOI:10.1016/j.foodchem.2021.129471.
[21] HEGELUND T O, BETUL Y, ISA F M, et al.AnOxPePred: using deep learning for the prediction of antioxidative properties of peptides[J].Scientific Reports, 2020, 10(1): 21471.DOI:10.1038/s41598-020-78319-w.
[22] AN G J, WEI M Y, WANG Z C, et al.Effects of enzymolysis method on the preparation of peptides from wheat flour[J].Food Bioscience,2022, 49: 101956.DOI:10.1016/J.FBIO.2022.101956.
[23] HUANG S F, LI Y L, LI C L, et al.Ultrasound-assisted multienzymatic system for the preparation of ACE inhibitory peptides with low bitterness from corn gluten meal[J].Processes, 2021, 9(12): 2170.DOI:10.3390/PR9122170.
[24] WANG J, WANG Y M, LI L Y, et al.Twelve antioxidant peptides from protein hydrolysate of skipjack tuna (Katsuwonus pelamis) roe prepared by flavourzyme: purification, sequence identification, and activity evaluation[J].Frontiers in Nutrition, 2022, 21(8): 813780.DOI:10.3389/fnut.2021.813780.
[25] LI S S, TAO L C, PENG S Q, et al.Structural and antioxidative properties of royal jelly protein by partial enzymatic hydrolysis[J].Food Science and Human Wellness, 2023, 12(5): 1820-1827.DOI:10.1016/J.FSHW.2023.02.046.
[26] JESIONOWSKI T, ZDARTA J, KRAJEWSKA B.Enzyme immobilization by adsorption: a review[J].Adsorption, 2014, 20(5/6):801-821.DOI:10.1007/s10450-014-9623-y.
[27] YAO Y M, WANG M Y, LIU Y, et al.Insights into the improvement of the enzymatic hydrolysis of bovine bone protein using lipase pretreatment[J].Food Chemistry, 2020, 302: 125199.DOI:10.1016/j.foodchem.2019.125199.
[28] SUN C Z, SHAN Y W, TANG X, et al.Effects of enzymatic hydrolysis on physicochemical property and antioxidant activity of mulberry(Morus atropurpurea Roxb.) leaf protein[J].Food Science & Nutrition,2021, 9(10): 5379-5390.DOI:10.1002/fsn3.2474.
[29] FAN T, HU J G, FU L D, et al.Optimization of enzymolysis ultrasonic assisted extraction of polysaccharides from Momordica charabtia L.by response surface methodology[J].Carbohydrate Polymers, 2015,115: 1-6.DOI:10.1016/j.carbpol.2014.09.009.
[30] ZHU L J, XIONG H G, HUNG X, et al.Identification and molecular mechanisms of novel antioxidant peptides from two sources of eggshell membrane hydrolysates showing cytoprotection against oxidative stress: a combined in silico and in vitro study[J].Food Research International, 2022, 157(1): 111266.DOI:10.1016/J.FOODRES.2022.111266.
[31] PHONGTHAI S, D’AMICO S, SCHOENLECHNER R, et al.Fractionation and antioxidant properties of rice bran protein hydrolysates stimulated by in vitro gastrointestinal digestion[J].Food Chemistry, 2018, 240(3): 156-164.DOI:10.1016/j.foodchem.2017.07.080.
[32] WALI A, WUBULIKASIMU A, YAN H G, et al.Separation and purification of antioxidant peptides from enzymatically prepared scorpion (Buthus martensii Karsch) protein hydrolysates[J].International Journal of Peptide Research and Therapeutics, 2019,26(4): 1-16.DOI:10.1007/s10989-019-09976-3.
[33] 蔡金秀, 夏姗姗, 马佳雯, 等.马面鱼皮胶原抗氧化肽的分离制备及稳定性研究[J].核农学报, 2021, 35(11): 2569-2577.DOI:10.11869/j.issn.100-8551.2021.11-2569.
[34] IRVINE G B.High-performance size-exclusion chromatography of peptides[J].Journal of Biochemical and Biophysical Methods, 2003,56(1/3): 233-242.DOI:10.1016/S0165-022X(03)00061-7.
[35] WU H C, CHEN H M, SHI C Y.Free amino acids and peptides as related to antioxidant properties in protein hydrolysates of mackerel(Scomber austriasicus)[J].Food Research International, 2003,36(9/10): 949-957.DOI:10.1016/s0963-9969(03)00104-2.
[36] XING L J, HU Y Y, HU H Y, et al.Purification and identification of antioxidative peptides from dry-cured Xuanwei ham[J].F o o d C h e m i s t r y, 2 0 1 6, 1 9 4: 9 5 1-9 5 8.D O I:1 0.1 0 1 6/j.foodchem.2015.08.101.
[37] ALEMAN A, GIMENEZ B, PEREZ-SANTIN E, et al.Contribution of Leu and Hyp residues to antioxidant and ACE-inhibitory activities of peptide sequences isolated from squid gelatin hydrolysate[J].Food Chemistry, 2010, 125(2): 334-341.DOI:10.1016/j.foodchem.2010.08.058.
[38] WONG F C, XIAO J, WANG S, et al.Advances on the antioxidant peptides from edible plant sources[J].Trends in Food Science &Technology, 2020, 99: 44-57.DOI:10.1016/j.tifs.2020.02.012.
[39] MA Y Y, XIONG Y L, ZHAI J J, et al.Fractionation and evaluation of radical scavenging peptides from in vitro digests of buckwheat protein[J].Food Chemistry, 2009, 118(0): 582-588.DOI:10.1016/j.foodchem.2009.05.024.
[40] MA Y K, WU Y Y, LI L H.Relationship between primary structure or spatial conformation and functional activity of antioxidant peptides from Pinctada fucata[J].Food Chemistry, 2018, 264: 108-117.DOI:10.1016/j.foodchem.2018.05.006.
[41] MATSUI R, HONDA R, KANOME M, et al.Designing antioxidant peptides based on the antioxidant properties of the amino acid sidechains[J].Food Chemistry, 2018, 245: 750-755.DOI:10.1016/j.foodchem.2017.11.119.
[42] MENDIS E, RAJAPAKSE N, BYUN H G, et al.Investigation of jumbo squid (Dosidicus gigas) skin gelatin peptides for their in vitro antioxidant effects[J].Life Sciences, 2005, 77(17): 2166-2178.DOI:10.1016/j.lfs.2005.03.016.
[43] AGRAWAL H, JOSHI R, GUPTA M.Isolation, purification and characterization of antioxidative peptide of pearl millet (Pennisetum glaucum) protein hydrolysate[J].Food Chemistry, 2016, 204: 365-372.DOI:10.1016/j.foodchem.2016.02.127.
[44] RANATHUNGA S, RAJAPAKSE N, KIM S K.Purification and characterization of antioxidative peptide derived from muscle of conger eel (Conger myriaster)[J].European Food Research and Technology,2006, 222(3/4): 310-315.DOI:10.1007/s00217-005-0079-x.
[45] LI C L, MORA L, TOLDRÁ F.Structure-function relationship of small peptides generated during the ripening of Spanish dry cured ham: peptidome, molecular stability and computational modelling[J].Food Chemistry, 2022, 375: 131673.DOI:10.1016/j/foodchem.2021.131673.
[46] LIU W Y, FANG L, FENG X W, et al.In vitro antioxidant and angiotensin I-converting enzyme inhibitory properties of peptides derived from corn gluten meal[J].European Food Research and Technology, 2020, 246: 2017-2027.DOI:10.1007/s00217-020-03552-6.
[47] SUN Y Y, SUN H Q, PAN L C, et al.Preparation, structure and α-glucosidase inhibitory of oligosaccharides by enzymatic hydrolysis from Annona squamosa polysaccharide[J].Industrial Crops and Products, 2022, 77: 114468.DOI:10.1016/j.indcrop.2021.114468.
[48] MIRZAEI M, MIRDAMADI S, EHSANI M R, et al.Production of antioxidant and ACE-inhibitory peptides from Kluyveromyces marxianus protein hydrolysates: purification and molecular docking[J].Journal of Food and Drug Analysis, 2018, 6(2): 696-705.DOI:10.1016/j.jfda.2017.07.008.