肉类及其制品是优质蛋白质、氨基酸和维生素的重要来源。随着全球人口增长和生活水平提高,全球肉类产量从2016年的3.171 7亿 t增加到2024年的3.506 4亿 t[1]。然而,肉品的丰富营养是微生物的理想培养基,新鲜肉品的高水分活度(>0.998)及中性至微酸性的pH值为各种细菌和真菌提供了理想的生长环境,加之脂质氧化和酶促自溶作用,共同促进了肉品的腐败。全球每年有7 300~9 100万 t肉类因腐败而被浪费[2],这不仅造成了巨大的经济损失,还引发了严重的食品安全问题。根据世界卫生组织报道,每年约有6亿 人患食源性疾病,其中有42万 人因食用不安全食品而死亡,其中由微生物性致病因子引起的病例占总发病人数的60%以上[3]。在我国,肉类是引发食源性疾病暴发的第二大常见食品类别,占所有食源性疾病暴发总数的16.4%,因此对肉品中微生物的检测尤为重要[4]。
目前,肉品微生物的检测主要依赖于经典的检测方法,如培养法、免疫学方法、生化反应、聚合酶链式反应(polymerase chain reaction,PCR)检测及荧光检测等,虽然这些方法操作简单、成本较低,但存在检测时间长、灵敏度低和操作过程复杂等局限,难以满足现代食品特别是肉品安全的快速检测和溯源需求。近年来,随着高通量测序(high-throughput sequencing,HTS)、数字PCR(digital PCR,dPCR)、基质辅助激光解吸电离飞行时间质谱(matrix-assisted laser desorption ionization time of flight mass spectrometry,MALDI-TOF MS)、红外光谱、拉曼光谱和高光谱等新技术的出现,肉品微生物检测在速度、灵敏度和特异性方面得到显著提升。HTS可以提供详尽的基因组信息,dPCR适用于低丰度病原体的检测,MALDI-TOF MS可实现快速、高度精确的鉴定,而红外光谱、拉曼光谱和高光谱技术则通过无损检测手段促进了实时质量安全的监控。这些新技术不仅克服了经典方法的多种局限性,还通过更精准、快捷的分析手段,大幅提高检测效率。当然,新的检测技术也面临着挑战,如设备成本高、操作复杂及缺乏统一的标准化流程。
基于此,本文对肉品中微生物检测经典方法的优势和不足进行总结,着重介绍一系列新型检测技术的原理、优势,探讨其在未来食品安全检测中的应用前景,旨在为提高肉品安全的监测水平提供技术支持。
1 肉品微生物经典检测技术
1.1 传统培养法
传统培养法仍是检测肉品微生物的主要标准。该方法通过将待检样本接种在特定的培养基上,并在适宜的条件下孵育形成菌落,再根据菌落的形态、颜色和大小初步鉴定微生物种类。细菌和真菌都能够在培养基上生长,形成可见菌落后进行初步检测,并根据需要进行其他下游检测。该方法无需复杂的设备和技术,成本较低,适合各种实验室条件,可生成基于培养基的食品病原体定性和定量数据。目前,GB 4789系列《食品安全国家标准 食品微生物学检验》均是以传统微生物学培养为基础的经典检验方法,培养法仍是食品微生物检测的金标准。
传统培养法的显著缺点是操作繁琐,需提前配制培养基,稀释管、枪头等用品需要高压灭菌,样品的前处理、增菌和分离培养通常需要48 h以上,致病菌和真菌检测常需要1 周甚至更长的时间,因此不适用于食源性病原菌的现场快速检测。此外,一些可培养的细菌(如伤寒杆菌和大肠杆菌)可能因环境压力导致其以具有活力但不可培养的形式存在,使检测产生假阴性结果[5]。某些微生物可能在特定培养基上无法生长或生长缓慢,且低浓度的微生物难以被检测到。
1.2 免疫学方法
免疫学检测方法是通过抗原-抗体的特异性结合来识别待测成分。通常采用酶联、荧光或胶体金等方式放大信号。检测时,颜色深浅与待检物质的量成比例,可直接根据颜色深浅进行样品分析。常见的方法如酶联免疫吸附测定(enzyme linked immunosorbent assay,ELISA)因其高通量、良好的特异性和稳健性及操作简便而被广泛使用[6]。王利刚[7]利用全自动ELISA荧光分析仪法对生鲜肉中单核细胞增生李斯特氏菌进行检测,将检测时间从传统检测周期所需的4~5 d缩短至51 h。免疫学检测方法的主要优点是操作简便,比传统培养法更快,通常可以在数小时内得到结果,可以检测毒素,且能够特异性地识别目标微生物,适用于检测难以培养的微生物。ELISA试剂盒法已被广泛应用于葡萄球菌肠毒素、黄曲霉毒素B1等微生物相关毒素的测定。
然而传统ELISA技术仍存在一些缺点,其检测限(最低102 CFU/mL)不足以达到许多蛋白质生物标志物的临床阈值,难以检测微生物污染早期阶段的低浓度抗原。此外,食品中的脂肪、盐类、碳水化合物等共存成分可能会抑制抗体与抗原的结合,导致ELISA检测受限。加之ELISA试剂盒价格较高,导致其检测成本高昂。
1.3 生化测试
生化测试方法是通过检测微生物在特定生化反应中的反应产物或相关酶活性鉴定微生物的种类和数量。生化反应可以通过检测细菌对各种基质的代谢作用及其代谢产物鉴别细菌的种属。例如,糖发酵试验、吲哚试验、甲基红试验、Voges-Proskauer试验、硫化氢试验、明胶液化试验和柠檬酸盐利用试验等,这些生化试验通常在特定的培养基中进行,可以提供关于微生物代谢特性的重要信息,有助于微生物的鉴定和分类[8]。生化测试检测方法具有检测特异性较高、成本较低、可了解微生物的代谢特性和生理功能的优点。目前,商品化的微生物生化鉴定试剂盒和配套仪器设备可将多个生化测试集成并实现自动化培养和分析鉴别,如梅里埃VITEK 2 Compact的全自动药敏分析仪、梅里埃AIP 20E等。
生化测试检测的缺点在于检测时间较长,通常需要24 h以上才能完成。对于复杂的微生物群体,可能需要多种生化测试来确认,难以在生产加工现场进行检测。此外,结果解读依赖专业人士,主观因素可能影响准确性。
1.4 PCR检测
PCR可以对特定的DNA和RNA系列进行扩增,因其特异性高、灵敏度高、重现性好而被广泛应用。PCR提供了更准确、灵敏和快速的单一细菌或基因检测,避免传统方法的表型特征模棱两可的情况。Heo等[9]利用hlyA基因设计特异性引物,建立的PCR法可以区分单核细胞增生李斯特氏菌和其他3 种同属异种菌。PCR技术的应用极大优化了检测步骤,缩短了检测时间。目前,GB 4789.6—2016《食品安全国家标准 食品微生物学检验》及GB 4789.40—2024《食品安全国家标准 食品微生物学检验 克罗诺杆菌检验》中致泻大肠埃希氏菌、克罗诺杆菌等致病菌可使用PCR和电泳的方式进行检测。
PCR系列技术在用于病原体检测的过程中也存在一些不足,如易受PCR抑制剂影响,且检测前需要分离出单个菌落并对其进行确定。PCR仪器和特异性引物费用较高,且无法对失去生物活性的菌株进行识别。此外,PCR反应的高灵敏度可能导致DNA被污染从而引起假阳性,失去活性的病菌DNA在体外扩增也可能导致假阳性的结果[10]。
PCR技术还有许多变化,如实时定量PCR、多重PCR等,可以定量检测样品中微生物数量,或同时一次性检测多种病原微生物。
1.5 荧光检测技术
腺苷三磷酸(adenosine triphosphate,ATP)荧光检测技术是一种基于生物发光反应的快速检测方法,被广泛应用于肉制品检测领域。该技术利用荧光素酶催化ATP和荧光素之间的反应产生荧光信号,通过测定荧光的强度间接反映样品中的ATP含量,从而推断出食品中的微生物或有机残留水平。ATP荧光检测技术具有检测灵敏度高、数据处理速度快、特异性强、操作简便等优点。Liu Zhenning等[11]提出了一种基于S1核酸酶、6-羧基荧光素胺标记的单链DNA和氧化石墨烯建立的新型传感器,用于检测ATP和评估牛肉样品的新鲜度。结果表明,在最佳条件下,荧光与20~3 500 μmol/L的ATP浓度之间存在线性相关性,检测限为3.2 μmol/L,表明该改良荧光技术的灵敏度和准确性高于紫外-可见光谱分析。ATP荧光检测技术的缺点也很明显,它无法区分不同类型的微生物(如病原菌与非病原菌),其检测结果只能反映总ATP含量,而不能具体指示污染来源。环境中残留的非微生物ATP或其他含ATP的物质可能干扰检测结果,导致结果出现假阳性。
经典检测技术的优势和不足如表1所示。
表1 经典检测技术的优势和不足
Table 1 Strengths and weaknesses of classical detection techniques

方法优势不足参考文献传统培养法操作简单,适应性高,可获得活菌 培养周期长,难以检测难培养微生物[5]生化测试特异性较高,可了解微生物代谢特性和生理功能检测时间较长,复杂群体,需要多种生化测试[8]PCR检测特异性高,灵敏度高,重现性好耗时,成本高,受PCR抑制剂影响,需要DNA纯化[10]荧光检测技术灵敏度高,数据处理速度快,特异性强,操作简便等无法提供微生物的种类信息,易受环境干扰[11]
表2 微生物检测新技术的优缺点、检测时长及检测限
Table 2 Advantages, disadvantages, time consumption, and limits of detection of new microbial detection techniques
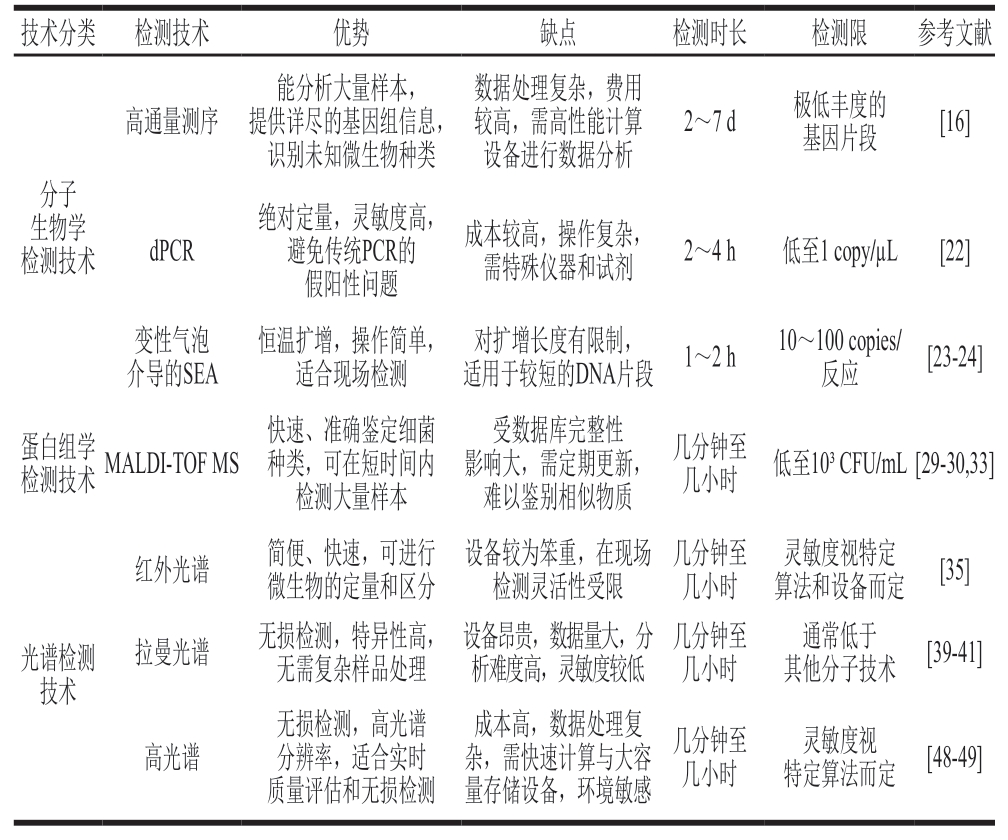
技术分类检测技术优势缺点检测时长检测限参考文献高通量测序能分析大量样本,提供详尽的基因组信息,识别未知微生物种类数据处理复杂,费用较高,需高性能计算设备进行数据分析2~7 d极低丰度的基因片段[16]分子生物学检测技术dPCR绝对定量,灵敏度高,避免传统PCR的假阳性问题成本较高,操作复杂,需特殊仪器和试剂2~4 h低至1 copy/µL[22]变性气泡介导的SEA恒温扩增,操作简单,适合现场检测适用于较短的DNA片段 1~2 h10~100 copies/反应[23-24]对扩增长度有限制,蛋白组学检测技术MALDI-TOF MS快速、准确鉴定细菌种类,可在短时间内检测大量样本受数据库完整性影响大,需定期更新,难以鉴别相似物质几分钟至几小时 低至10³ CFU/mL [29-30,33]红外光谱简便、快速,可进行微生物的定量和区分设备较为笨重,在现场检测灵活性受限几分钟至几小时灵敏度视特定算法和设备而定 [35]光谱检测技术拉曼光谱无损检测,特异性高,无需复杂样品处理设备昂贵,数据量大,分析难度高,灵敏度较低几分钟至几小时通常低于其他分子技术 [39-41]高光谱无损检测,高光谱分辨率,适合实时质量评估和无损检测成本高,数据处理复杂,需快速计算与大容量存储设备,环境敏感几分钟至几小时灵敏度视特定算法而定 [48-49]
2 肉品微生物检测新技术
2.1 基于分子生物学的检测方法
2.1.1 HTS技术
HTS技术也被称为下一代测序(next-generation sequencing,NGS)技术,是现代分子生物学的重要工具。这项技术能够同时测定数十万至数百万条DNA分子序列,极大扩展了基因组学的应用范围,是目前应用最广泛的测序方法[12]。
NGS技术显著提高了测序速度和效率,能同时处理多个样本,生成大量基因组数据。在肉品微生物检测中,NGS主要有2 种应用方式:一是通过对微生物进行分离培养,对单个微生物进行全基因组测序。例如,Wang Qin等[13]利用该技术完成了中国零售肉类中大肠杆菌的测序,报道了mcr-10在零售肉类中的质粒介导耐药基因的传播,揭示了肉品中的大肠杆菌可能通过食物链传播抗性基因,该研究表明,NGS技术在揭示微生物耐药性、毒力因子及遗传变异信息方面显示出巨大潜力。二是无需对微生物进行分离培养即可快速识别肉品中微生物种类的宏组学技术。例如,de Matos等[14]利用NGS技术分析牛肉中细菌多样性,确定了关键细菌种类的相对丰度,与传统方法相比,NGS提供了更具体和精确的微生物群落信息。Park等[15]则利用NGS技术全面分析鸡肉供应链各环节的微生物群落,识别微生物的来源和扩散路径,从而提高食品安全管理效率。然而,该技术实验及分析周期长,对实验人员技术要求高,且需要高效的计算和分析方法处理庞大的数据量,否则会影响结果的准确性。此外,不同实验室的测序平台、样本处理、数据分析方法的差异也可能影响结果的可比性。短读长测序平台还可能产生一定比例的测序错误,并且样本间的交叉污染也可能进一步影响数据的可靠性[16]。
2.1.2 dPCR技术
dPCR是一种用于核酸分子绝对定量的技术,该技术将核酸样品分配到数万个独立微反应单元中,每个单元尽可能只含1 个目标模板分子,然后进行独立PCR扩增。通过读取荧光信号判断结果,阳性为“1”,阴性为“0”,最后统计阳性个数和比例。由于反应孔中可能含有零个或多个模板分子,dPCR通过泊松统计法校准数据,计算拷贝数和浓度[17],其反应原理如图1所示。
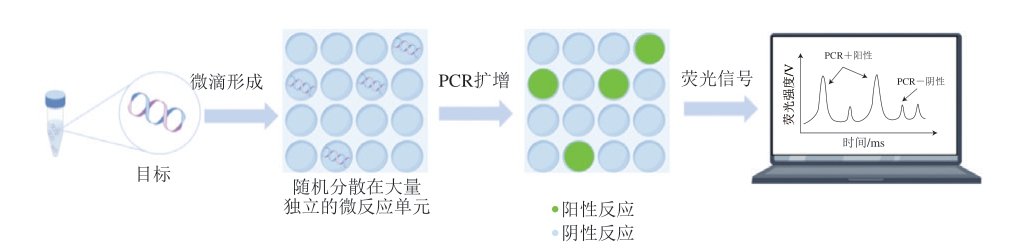
图1 dPCR工作原理图
Fig. 1 Schematic diagram of the working principle of dPCR
与传统的实时荧光定量PCR(real-time fluorescence quantitative PCR,real-time PCR)相比,dPCR无需借助内参基因、标准品或标准曲线即可直接实现核酸的绝对定量分析;泊松分布的应用使得dPCR能够检测到样本中的微小差异,特别适用于复杂背景序列中低丰度目的序列的定量检测。dPCR对样本进行极限稀释,检测灵敏度可达单个核酸分子,减少了靶标分子对扩增试剂的竞争。因此,dPCR在低浓度病原体的检测中表现出显著优势,即使在复杂样品基质中也能保持高灵敏度和抗抑制剂能力[18]。
我国目前对食源性致病菌的检测主要依赖于耗时较长的传统培养法和real-time PCR技术等,但在食品基质复杂、目标分子含量低的情况下,这些方法的检测结果可能存在偏差。dPCR无需建立标准曲线即可进行绝对定量分析,缩短了检测时间,在食源性致病菌的检测中已经得到广泛应用。Öz等[19]开发了一种基于微滴式dPCR(droplet dPCR,ddPCR)的检测方法,用于快速检测生碎肉样品中的沙门氏菌属。该方法通过短时预增菌步骤,结合特异性的引物和探针设计,成功实现了对沙门氏菌属的准确检测,检测限达到1.39 CFU/mL。Kong Jiaqi等[20]将NGS与ddPCR相结合,用于准确检测肉品中的大肠杆菌O157:H7和鼠伤寒沙门氏菌血清型,在鸡肉表面的检测限为3~4(lg(CFU/g)),在鸡肉等食品中可检测到低至2~3(lg(CFU/mL))的病原菌。此外,Suo Yuanjie 等[21]开发了一种基于适配体的dPCR芯片用于鼠伤寒沙门氏菌检测,能够在复杂食品基质中直接定量细菌,检测限低至90 CFU/反应,整个过程仅需约2 h。目前,ddPCR方法已在一系列进出口行业标准中被应用于食品致病菌和病毒检测,作为快速检测放行的依据。
然而,dPCR仍存在一些局限性,如试剂和设备价格昂贵,使用成本较高;液滴的稳定性与可控性相对较差,难以避免液滴的少量融合[22];大部分仪器对结果采用终点检测法,无法监控扩增过程中的实时荧光数据,影响了数据的可信度。随着技术的进步和成本的降低,dPCR技术有望在肉品微生物检测中得到更广泛的应用。
2.1.3 变性气泡介导的链交换扩增(strand exchange amplification,SEA)技术
SEA技术是一种基于DNA呼吸作用引起的变性泡介导的恒温核酸扩增技术,于2016年被提出。SEA引入了链置换酶(strand displacement enzyme,SDE),该酶在低温下具有高度特异性和高效率的链交换活性。在SEA反应中,引物与待扩增的目标序列结合,SDE将引物与目标序列进行链交换,从而生成新的扩增产物,通过不断循环此过程,实现恒温下靶核酸区域的指数式扩增,SEA反应原理如图2所示。
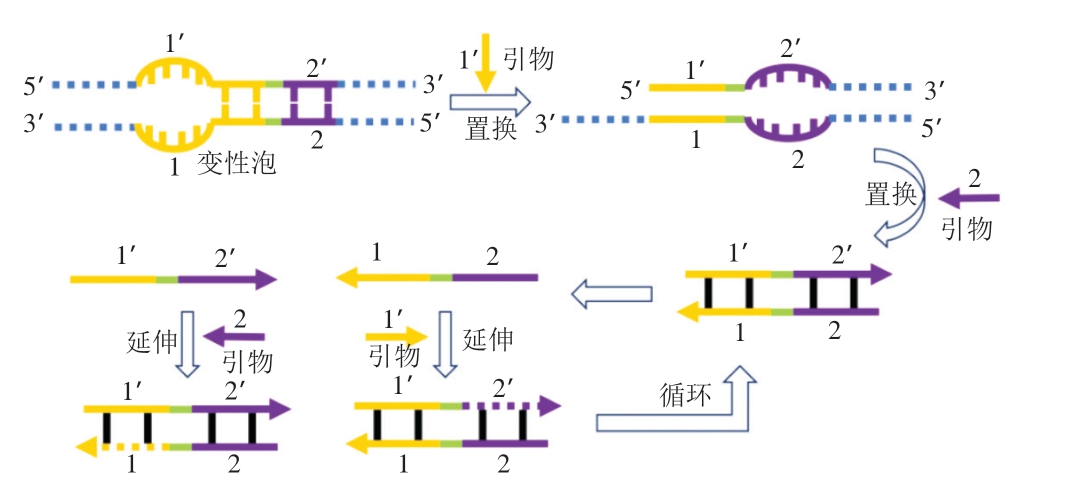
图2 SEA反应原理示意图
Fig. 2 Schematic diagram of the principle of strand exchange amplification
SEA技术为肉品微生物检测提供了一种快速、灵敏且成本效益高的检测方法[23]。基于SEA技术,研究人员开发了交错SEA(staggered SEA,SSEA)和加速SEA技术,进一步提高了检测速度和灵敏度。这些简单、快速、低成本且高灵敏的技术已被广泛应用于食品安全领域的即时分子诊断,尤其在大肠杆菌、李斯特菌和金黄色葡萄球菌等食源性病原菌的检测中表现出显著优势[24]。Zhang Meilin等[25]利用SEA技术有效检测了低至1.0×10-13 mol/L的基因组DNA,无需对样品进行复杂的预处理,即可实现基因组DNA、培养液和菌落的实时荧光检测,大大提高了检测效率。金黄色葡萄球菌污染已成为世界性的食品安全问题,SEA特别适用于金黄色葡萄球菌的现场快速检测和大规模筛查[26]。Zhang Jian等[27]开发的SSEA技术在1 h内实现了低至1.0×10³ CFU/g的金黄色葡萄球菌检测,展示了其在食源性致病菌检测中的应用潜力。
SEA技术也存在一定的局限性,由于SDE的限制,SEA技术对扩增长度有一定的限制,通常适用于较短的目标序列,且对于新使用者来说,获得最优的引物较为困难,这些因素限制了其应用范围。随着技术的改进,SEA有望在食品安全检测中发挥更重要的作用。
2.2 基于蛋白组的检测方法(MALDI-TOF MS技术)
MALDI-TOF MS技术是一种用于微生物快速鉴定的技术,该技术通过检测微生物细胞内特定蛋白质的质谱图谱来实现微生物的鉴定。这些蛋白质(如管家蛋白和核糖体蛋白)被提取并离子化后,根据其质量-电荷比在飞行管中飞行并被检测器检测,形成特征性的质谱图,随后通过将这些质谱图与已知微生物的数据库进行比对,利用算法评估匹配,从而实现对微生物的快速、准确鉴定[28],其反应原理如图3所示。
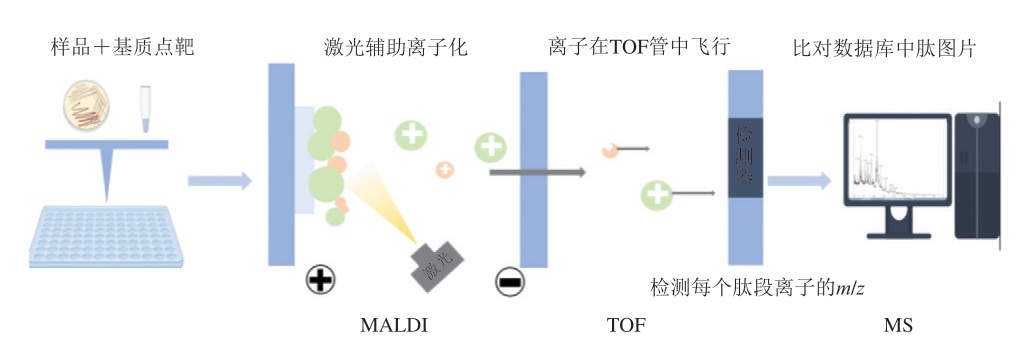
图3 MALDI-TOF MS检测原理图
Fig. 3 Schematic diagram of the working principle of MALDI-TOF MS
MALDI-TOF MS通过分析微生物的蛋白质组成进行鉴定,由于这些蛋白质在物种内部通常具有高度的保守性和独特性,MALDI-TOF MS能够提供具有高度确定性的鉴定结果[29]。此外,MALDI-TOF MS技术所需样品和制备步骤少,数据处理时间短,可在短时间内对大量样本进行检测,因此常被用于微生物的快速鉴定。该技术已被成熟应用于单一细菌的快速鉴定,准确率高且检测时间短,在1 h内的物种鉴定正确率为84%~94%,被证明是一种非常快速、经济且可靠的细菌鉴定工具[30]。Sapugahawatte等[31]利用MALDI-TOF MS从香港湿货市场的鱼类和猪肉中鉴定出多重耐药病原体。Wang Chen等[32]利用该技术以98.5%的准确率鉴定了从日本和埃及采集的食品样本中的肠球菌。GB/T 33682—2017《基质辅助激光解析电离飞行时间质谱鉴别微生物方法通则》也给出了MALDI-TOF MS应用于微生物鉴定检测中的应用原则和要求,有利于未来该方法在微生物鉴定领域的推广应用。
MALDI-TOF MS技术在应用中也有一定的局限性,首先是数据库中参考谱图的完整性和准确性直接影响物种的识别效果,因此还需不断更新图谱库以增加鉴定的准确性;此外,MALDI-TOF MS谱图的重现性受样品培养、制备和保存及所使用的基质类型影响;当物种具有近似相同的谱图时(如大肠杆菌和志贺氏杆菌[33]),仍需通过表型方法和血清学实验等进一步区分。随着数据库的完善和样本处理技术的进步,MALDI-TOF MS有望逐步取代传统的生化鉴定方法。
2.3 基于光谱技术的检测方法
2.3.1 红外光谱技术
红外光谱通过测量物质对特定频率红外光的吸收或发射情况获取物质的化学结构和组成信息。红外覆盖频率范围为12 500~33 cm-1,可分为近中红外(12 500~400 cm-1)和远红外(400~33 cm-1)[34]。近红外和中红外光谱在化学分析和微生物检测中被广泛应用,因为微生物的细胞膜和细胞壁含有独特的化学成分,如核酸、蛋白质等,它们在红外光谱中有特定的吸收特征[35]。尽管微生物的红外光谱可能表现出相似性,借助光谱预处理技术和化学计量学方法(如偏最小二乘回归(partial least squares regression,PLSR)、基于支持向量机(support vector machine,SVM)回归和人工神经网络等),仍然可以实现微生物的准确定量分析和区分。此外,采用主成分分析(principal component analysis,PCA)、偏最小二乘判别分析等方法可以对微生物进行有效的识别和分类。通常通过计算相关系数和均方根误差确保模型的准确性和可靠性,以评估预测模型的性能[36]。
由于红外光谱操作简便、分析速度快且应用范围广泛,已成为成熟的微生物检测手段。目前,已建立多种微生物的红外光谱数据库,并且商业化的红外光谱微生物检测设备也已问世。然而,大多数研究仍依赖于昂贵且笨重的台式仪器,限制了其在现场微生物检测中的灵活性和适应性。同时,近红外光谱设备在应用于先前未分析过的样本时,通常缺乏稳健性和准确性。因此,建立更全面的样本数据库和使用先进的算法对于实现稳定性和高准确性至关重要。在Zhang Zhepeng[37]、Zhang Fan[38]等的研究中,通过改进即时报学习模型,提出了一种稳健的近红外光谱方法,用于检测猪肉中的总挥发性盐基氮(total volatile basic nitrogen,TVB-N)含量。该方法结合了自研的双波段传感器(可见光/短波近红外光谱仪和长波近红外光谱仪),用来采集猪肉的光谱反射率,通过即时报学习策略和最小二乘SVM预测模型,显著提高了对未知样本的预测精度,特别是在新批次样本的预测中,模型预测决定系数达到0.92。这项研究证实近红外光谱技术在快速、无损检测肉类变质方面的应用潜力,特别是在那些对检测精度和模型实时更新有高要求的场景中。通过扩展样本库和优化算法,可以进一步提高模型的鲁棒性和预测的准确性,这表明结合现代算法和便携式设备的红外光谱技术在未来微生物检测和精准防控中具有巨大潜力。
2.3.2 拉曼光谱技术
拉曼光谱技术在肉品微生物检测领域得到了广泛应用,该技术通过检测肉品样本的拉曼散射光,能够获得肉品中微生物的分子结构和组成信息,从而检测肉品中微生物的种类、数量和活性。这为快速评估肉品的质量和安全提供了依据[39],其检测流程如图4所示。
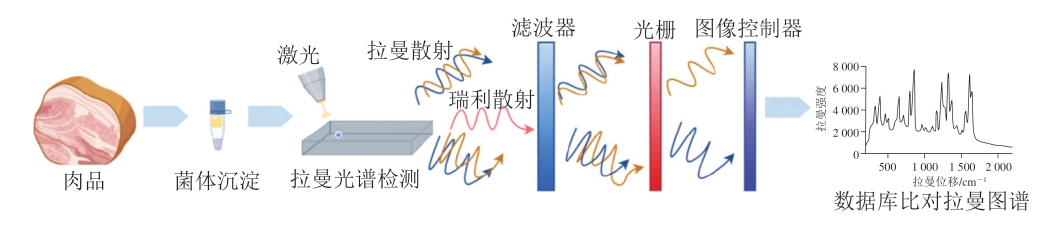
图4 拉曼光谱用于肉品中微生物检测流程图
Fig. 4 Flow chart of applying Raman spectroscopy for microbial detection in meat products
与传统的微生物培养方法相比,拉曼光谱检测技术速度快,特异性高,且无需对样品进行复杂处理,具有显著优势[40]。拉曼光谱技术因其对肉品贮藏期间化学变化的高灵敏度监测,成为评估肉品新鲜度的关键工具。这些化学变化包括蛋白质分解和脂质氧化,通常由微生物活动引起,与肉品腐败过程密切相关。结合拉曼光谱数据和多元统计分析方法(PLSR和PCA),可以预测肉品中的TVB-N含量,进而评估肉品腐败程度。通过正交信号校正和卷积神经网络(convolutional neural network,CNN)等机器学习算法,拉曼光谱技术不仅提升了微生物种类识别的准确性,还能有效区分不同腐败阶段的微生物[41]。例如,便携式拉曼光谱仪结合CNN在鲈鱼鱼片新鲜度检测中展现了快速、无损的检测潜力,提供了食品安全和质量评估的高效工具。特征选择与方差分析的结合,显著提升了CNN模型的分类准确性和计算效率,为肉类产品的微生物安全评估提供了新途径[42]。此外,Liu Shijie等[43]开发了一种基于Ag@AuNP阵列的表面增强拉曼散射光谱技术,实现了对5 种常见细菌孢子的快速、准确鉴定,为肉制品中的孢子监测提供了前景广阔的技术策略。
高昂的设备成本限制了拉曼光谱在肉类产业中的广泛应用[44]。此外,数据量大、分析难度高、灵敏度低等问题使其推广应用面临挑战。未来的研究应致力于开发更轻便、小型化的拉曼光谱设备,并探索成本效益更高的解决方案,以实现在线实时监测和与现有设备的集成。
2.3.3 高光谱技术
高光谱成像(hyperspectral imaging,HSI)技术(图5)是一种集成成像与光谱分析的先进手段,能够捕获每个像素点在不同波长下的光谱信息,生成包含空间和波长维度的三维数据集,即超立方体。与传统的成像或光谱技术相比,HSI的独特之处在于其能够同时测量图像中每个像素的空间信息和光谱参数,捕捉从可见光到近红外或短波红外光谱(0.4~2.5 μm)范围内的连续光谱信息[45]。
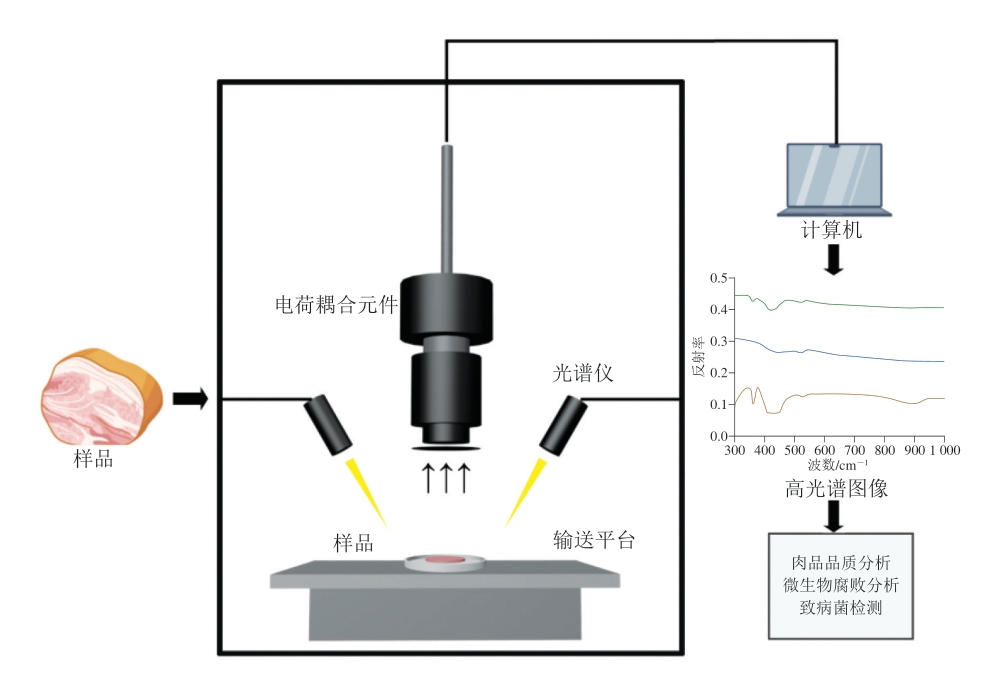
图5 HSI系统示意图
Fig. 5 Schematic diagram of hyperspectral imaging system
当表面带有微生物的食品或液体培养基暴露在HSI系统的光源下,光线与样品相互作用,产生反射或透射光。由于微生物细胞结构和化学组成(如蛋白质、脂类、核酸等)的差异,它们在特定波长范围内会表现出特定的光谱响应。通过这些光谱特征,可以将不同类型的微生物区分开来。例如,不同种类的细菌或真菌在特定波长下的光谱差异可用于分类[46]。
作为一种非侵入性、非接触性、非破坏性的技术,HSI的高光谱分辨率使其在快速、无损监测微生物腐败方面具有显著优势。与耗时的传统化学方法相比,HSI的处理时间更短,更适合于实时质量评估。此外,HSI能够精确识别图像中的关键分析区域,同时获取空间和光谱信息,提供更准确、全面的数据分析,为后续实验研究奠定了坚实的基础[47]。
HSI在预测总活菌数(total viable count,TVC)方面显示出巨大潜力。Li Huanhuan等[48]通过结合猪肉样本的高光谱数据和比色传感器响应,应用多变量分析方法,建立高效SVM的TVC预测模型,其模型的预测均方根误差为2.991 3,这显示出HSI与比色传感器相结合的多变量分析方法在无损检测猪肉质量与安全方面具有巨大潜力。此外,HSI也被用于其他微生物的快速无损检测。Zhao Jiexi等[49]利用HSI结合AdaBoost-RT算法对冷藏鸡肉中的假单胞菌进行检测,通过获取329~1 113 nm波段的高光谱数据,建立了新鲜鸡胸肉中定性检测金黄色葡萄球菌的方法。该研究将HSI与PCA、多种特征选择技术(如竞争自适应重加权采样、遗传算法和连续投影算法)、SVM算法相结合,开发出分类模型。HSI在食品质量和安全评估,尤其是评估肉类新鲜度和微生物风险方面具有较好的应用潜力[50]。
尽管HSI技术具有明显优势,但也存在一些局限性。HSI系统的购置成本较高,且由于产生大量数据,需要高速计算机进行数据处理和大容量存储设备。图像采集过程中,信号可能受到环境因素如散射和照明的影响,可能导致信噪比降低。此外,通常难以使用光谱数据在等效图像中检测和识别到不同的物体。尽管存在这些挑战,HSI技术在食品检测领域的应用前景依然广阔,值得进一步的研究与开发。
3 结 语
在食品检测过程中,经典检测方法仍然是应用最广泛的方法,某些检测新技术则成为辅助手段。这些技术如NGS、dPCR、SEA、MALDI-TOF MS和红外光谱、拉曼光谱、HSI等,不仅在检测速度、灵敏度和特异性方面表现出显著优势,还大大提高了检测效率和准确性。其中,NGS能够同时分析多个样本的基因组信息,识别未知微生物种类,为揭示微生物的耐药性和毒力因子提供了新的视角。dPCR技术凭借其高灵敏度和绝对定量的优势,成为低丰度病原体检测的理想选择。SEA技术因其恒温扩增的特性,非常适合现场快速检测。MALDITOF MS则通过质谱分析微生物的蛋白质谱图,实现细菌的快速鉴定。红外光谱技术则实现了微生物的定量分析和区分,能够在短时间内提供准确的分析结果。而拉曼光谱、HSI技术以其非破坏、非入侵性和快速检测的优点,实现了样品的无损检测。
尽管这些新技术在肉品微生物检测中展现出广阔的应用前景,但其在实际应用中仍面临一些挑战。如NGS和dPCR技术的应用成本较高,数据处理复杂,设备成本高;MALDI-TOF MS受限于数据库的完整性,难以鉴别相似物种;而拉曼光谱技术则由于设备昂贵和数据分析难度大,在肉类产业中的推广应用受到限制。此外,HSI和红外光谱虽然以其无损、快速检测的优势广泛应用于食品质量评估,但它们的购置成本较高,数据处理复杂,且容易受到环境因素的影响,从而限制了其在实际生产中的大规模应用。未来的研究应着眼于降低技术成本,提高其在实际生产环境中的应用可行性,特别是开发小型化、集成化的设备,以实现实时在线监测,从而更有效地保障食品安全。
[1] Organisation for Economic Co-operation and Development and Food and Agriculture Organization of the United Nations. OECD-FAO Agricultural Outlook 2024—2033[R]. Paris, France and Rome: OECD and FAO, 2024.
[2] Food and Agriculture Organization of the United Nations. FAO platform on food loss and waste[R]. Rome: FAO, 2024.
[3] World Health Organization. WHO estimates of the global burden of foodborne diseases: foodborne diseases burden epidemiology reference group 2007—2015[R]. Geneva: WHO, 2015.
[4] LI W W, PIRES S M, LIU Z T, et al. Surveillance of foodborne disease outbreaks in China, 2003—2017[J]. Food Control, 2020, 118: 107359.DOI:10.1016/j.foodcont.2020.107359.
[5] FODDAI A C G, GRANT I R. Methods for detection of viable foodborne pathogens: current state-of-art and future prospects[J].Applied Microbiology and Biotechnology, 2020, 104(10): 4281-4288.DOI:10.1007/s00253-020-10542-x.
[6] PORTILHO A I, GIMENES LIMA G, DE GASPARI E. Enzymelinked immunosorbent assay: an adaptable methodology to study SARS-CoV-2 humoral and cellular immune responses[J]. Journal of Clinical Medicine, 2022, 11(6): 1503. DOI:10.3390/jcm11061503.
[7] 王利刚. 生鲜牛肉中单核细胞增生李斯特氏菌检验方法的比较[J]. 肉类工业, 2018(2): 54-57. DOI:10.3969/j.issn.1008-5467.2018.02.011.
[8] CHAUHAN A, JINDAL T. Biochemical and molecular methods for bacterial identification[M]//CHAUHAN A, JINDAL T. Microbiological methods for environment, food and pharmaceutical analysis. Cham:Springer International Publishing, 2020: 425-468. DOI:10.1007/978-3-030-52024-3_10.
[9] HEO E J, SONG B R, PARK H J, et al. Rapid detection of Listeria monocytogenes by real-time PCR in processed meat and dairy products[J]. Journal of Food Protection, 2014, 77(3): 453-458.DOI:10.4315/0362-028X.JFP-13-318.
[10] 王丹丹, 刘鸣畅, 杨艳歌, 等. 食源性致病菌快速检测技术研究进展[J].食品科学, 2022, 43(3): 276-285. DOI:10.7506/spkx1002-6630-20201105-048.
[11] LIU Z N, ZHONG Y H, HU Y, et al. Fluorescence strategy for sensitive detection of adenosine triphosphate in terms of evaluating meat freshness[J]. Food Chemistry, 2019, 270: 573-578. DOI:10.1016/j.foodchem.2018.07.041.
[12] SATAM H, JOSHI K, MANGROLIA U, et al. Next-generation sequencing technology: current trends and advancements[J]. Biology,2023, 12(7): 997. DOI:10.3390/biology12070997.
[13] WANG Q, HAN Y Y, ZHANG T J, et al. Whole-genome sequencing of Escherichia coli from retail meat in China reveals the dissemination of clinically important antimicrobial resistance genes[J]. International Journal of Food Microbiology, 2024, 415: 110634. DOI:10.1016/j.ijfoodmicro.2024.110634.
[14] DE MATOS L G, DA SILVA ABREU A C, ALONSO V P P, et al.Comparison of bacterial diversity in wet- and dry-aged beef using traditional microbiology and next generation sequencing[J]. The Microbe, 2024, 2: 100035. DOI:10.1016/j.microb.2024.100035.
[15] PARK J, BAE D, KIM S A. Microbial trace investigation throughout the entire chicken supply chain based on metagenomic high-throughput sequencing[J]. Food Research International, 2023, 169: 112775.DOI:10.1016/j.foodres.2023.112775.
[16] 刁振丽, 李金明. 宏基因组高通量测序技术的临床应用: 现状、挑战与前景[J]. 协和医学杂志, 2023, 14(5): 905-910. DOI:10.12290/xhyxzz.2023-0089.
[17] SANCHA DOMINGUEZ L, COTOS SUÁREZ A, SÁNCHEZ LEDESMA M, et al. Present and future applications of digital PCR in infectious diseases diagnosis[J]. Diagnostics, 2024, 14(9): 931.DOI:10.3390/diagnostics14090931.
[18] 秦爱, 王娟, 邓方进, 等. 数字聚合酶链式反应技术在食品安全核酸检测领域中的研究进展及标准化现状[J]. 食品科学, 2024, 45(18):350-360. DOI:10.3969/j.issn.1008-0589.2004.03.009.
[19] ÖZ Y Y, SÖNMEZ Ö İ, KARAMAN S, et al. Rapid and sensitive detection of Salmonella spp. in raw minced meat samples using droplet digital PCR[J]. European Food Research and Technology, 2020,246(10): 1895-1907. DOI:10.1007/s00217-020-03531-x.
[20] KONG J Q, FAN C C, LIAO X J, et al. Accurate detection of Escherichia coli O157:H7 and Salmonella enterica serovar typhimurium based on the combination of next-generation sequencing and droplet digital PCR[J]. LWT-Food Science and Technology, 2022,168: 113913. DOI:10.1016/j.lwt.2022.113913.
[21] SUO Y J, YIN W H, ZHU Q Y, et al. A specific and sensitive aptamerbased digital PCR chip for Salmonella typhimurium detection[J].Biosensors, 2022, 12(7): 458. DOI:10.3390/bios12070458.
[22] BRAZELTON DE CÁRDENAS J N, HAYDEN R T. Applications of digital PCR in clinical microbiology[J]. Advanced Techniques in Diagnostic Microbiology, 2018, 2: 685-698. DOI:10.1007/978-3-319-95111-9_29.
[23] LIN S, ZHANG X, TIAN A N, et al. Boron nitride nanoplate-based strand exchange amplification with enhanced sensitivity and rapidity for quantitative detection of Staphylococcus aureus in food samples[J].Analytical Methods, 2024, 16(6): 892-898. DOI:10.1039/d3ay02076b.
[24] DENG J, LI Y, SHI W Q, et al. Primer design strategy for denaturation bubble-mediated strand exchange amplification[J]. Analytical Biochemistry, 2020, 593: 113593. DOI:10.1016/j.ab.2020.113593.
[25] ZHANG M L, WANG X D, HAN L Z, et al. Rapid detection of foodborne pathogen Listeria monocytogenes by strand exchange amplification[J]. Analytical Biochemistry, 2018, 545: 38-42.DOI:10.1016/j.ab.2018.01.013.
[26] LIU C Y, SHI C, LI M Z, et al. Rapid and simple detection of viable foodborne pathogen Staphylococcus aureus[J]. Frontiers in Chemistry,2019, 7: 124. DOI:10.3389/fchem.2019.00124.
[27] ZHANG J, HAN X N, WANG Y, et al. Rapid and sensitive detection of Staphylococcus aureus via an all-in-one staggered strand exchange amplification platform[J]. Analyst, 2023, 148(9): 1970-1977.DOI:10.1039/D2AN02092K.
[28] 尹盼盼, 白雯静, 李海燕, 等. 基质辅助激光解吸电离飞行时间质谱在微生物鉴定中的研究进展[J]. 微生物学通报, 2023, 50(10): 4655-4666. DOI:10.13344/j.microbiol.china.230127.
[29] SINGHAL N, KUMAR M, KANAUJIA P K, et al. MALDI-TOF mass spectrometry: an emerging technology for microbial identification and diagnosis[J]. Frontiers in Microbiology, 2015, 6: 791. DOI:10.3389/fmicb.2015.00791.
[30] SENG P, DRANCOURT M, GOURIET F, et al. Ongoing revolution in bacteriology: routine identification of bacteria by matrix-assisted laser desorption ionization time-of-flight mass spectrometry[J]. Clinical Infectious Diseases, 2009, 49(4): 543-551. DOI:10.1086/600885.
[31] SAPUGAHAWATTE D N, LI C, ZHU C, et al. Prevalence and characteristics of extended-spectrum-β-lactamase-producing and carbapenemase-producing enterobacteriaceae from freshwater fish and pork in wet markets of Hong Kong[J]. mSphere, 2020, 5(2): 00107-20.DOI:10.1128/msphere.00107-20.
[32] WANG C, EL-TELBANY M, LIN Y Z, et al. Identification of Enterococcus spp. from food sources by matrix-assisted laser desorption ionization-time of flight mass spectrometry and characterization of virulence factors, antibiotic resistance, and biofilm formation[J]. International Journal of Food Microbiology, 2024, 420:110768. DOI:10.1016/j.ijfoodmicro.2024.110768.
[33] ASHFAQ M Y, DA’NA D A, AL-GHOUTI M A. Application of MALDI-TOF MS for identification of environmental bacteria: a review[J]. Journal of Environmental Management, 2022, 305: 114359.DOI:10.1016/j.jenvman.2021.114359.
[34] ZHANG J H, LI F W, SHEN S X, et al. More simple, efficient and accurate food research promoted by intermolecular interaction approaches: a review[J]. Food Chemistry, 2023, 416: 135726.DOI:10.1016/j.foodchem.2023.135726.
[35] SHAPAVAL V, SCHMITT J, MØRETRØ T, et al. Characterization of food spoilage fungi by FTIR spectroscopy[J]. Journal of Applied Microbiology, 2013, 114(3): 788-796. DOI:10.1111/jam.12092.
[36] CHENG J H, SUN D W. Partial least squares regression (PLSR)applied to NIR and HSI spectral data modeling to predict chemical properties of fish muscle[J]. Food Engineering Reviews, 2017, 9(1):36-49. DOI:10.1007/s12393-016-9147-1.
[37] ZHANG Z P, SUN Y, SANG S Y, et al. Emerging approach for fish freshness evaluation: principle, application and challenges[J]. Foods,2022, 11(13): 1897. DOI:10.3390/foods11131897.
[38] ZHANG F, KANG T H, SUN J F, et al. Improving TVB-N prediction in pork using portable spectroscopy with just-in-time learning model updating method[J]. Meat Science, 2022, 188: 108801. DOI:10.1016/j.meatsci.2022.108801.
[39] 韩爱云, 张振冉, 解立斌, 等. 拉曼光谱技术在肉类掺假检测方面的应用研究进展[J]. 肉类研究, 2021, 35(7): 50-54. DOI:10.7506/rlyj1001-8123-20210316-073.
[40] JAAFREH S, VALLER O, KREYENSCHMIDT J, et al. In vitro discrimination and classification of microbial flora of poultry using two dispersive Raman spectrometers (microscope and Portable Fiber-Optic systems) in tandem with chemometric analysis[J]. Talanta, 2019,202: 411-425. DOI:10.1016/j.talanta.2019.04.082.
[41] LIU Q S, DONG P C, FENGOU L C, et al. Preliminary investigation into the prediction of indicators of beef spoilage using Raman and Fourier transform infrared spectroscopy[J]. Meat Science, 2023, 200:109168. DOI:10.1016/j.meatsci.2023.109168.
[42] WANG K Q, YUE Z L, LIN H, et al. Rapid classification of the freshness grades of sea bass (Lateolabrax japonicus) fillets using a portable Raman spectrometer with machine learning method[J].Microchemical Journal, 2023, 192: 108948. DOI:10.1016/j.microc.2023.108948.
[43] LIU S J, ZHU Y D, ZHAO L J, et al. Characteristic substance analysis and rapid detection of bacteria spores in cooked meat products by surface enhanced Raman scattering based on Ag@AuNP array substrate[J]. Analytica Chimica Acta, 2024, 1308: 342616.DOI:10.1016/j.aca.2024.342616.
[44] WANG Y M, FANG L R, WANG Y H, et al. Current trends of Raman spectroscopy in clinic settings: opportunities and challenges[J].Advanced Science, 2024, 11(7): e2300668. DOI:10.1002/advs.202300668.
[45] ELMASRY G, KAMRUZZAMAN M, SUN D W, et al. Principles and applications of hyperspectral imaging in quality evaluation of agro-food products: a review[J]. Critical Reviews in Food Science and Nutrition,2012, 52(11): 999-1023. DOI:10.1080/10408398.2010.543495.
[46] SONI A, DIXIT Y, REIS M M, et al. Hyperspectral imaging and machine learning in food microbiology: developments and challenges in detection of bacterial, fungal, and viral contaminants[J].Comprehensive Reviews in Food Science and Food Safety, 2022,21(4): 3717-3745. DOI:10.1111/1541-4337.12983.
[47] LU G L, FEI B W. Medical hyperspectral imaging: a review[J].Journal of Biomedical Optics, 2014, 19(1): 10901. DOI:10.1117/1.JBO.19.1.010901.
[48] LI H H, KUTSANEDZIE F, ZHAO J W, et al. Quantifying total viable count in pork meat using combined hyperspectral imaging and artificial olfaction techniques[J]. Food Analytical Methods, 2016, 9(11): 3015-3024. DOI:10.1007/s12161-016-0475-9.
[49] ZHAO J X, MEHMOOD A, DONG Q L, et al. Rapid assessment of chilled chicken spoilage based on hyperspectral imaging technology and AdaBoost-RT[J]. Food Analytical Methods, 2023, 16(9): 1504-1511. DOI:10.1007/s12161-023-02501-9.
[50] QIU R C, ZHAO Y L, KONG D D, et al. Development and comparison of classification models on VIS-NIR hyperspectral imaging spectra for qualitative detection of the Staphylococcus aureus in fresh chicken breast[J]. Spectrochimica Acta Part A: Molecular and Biomolecular Spectroscopy, 2023, 285: 121838. DOI:10.1016/j.saa.2022.121838.