沙门氏菌是世界上最常见的食源性病原体之一,可引起人和动物的多种疾病,如胃肠炎、副伤寒和伤寒等[1-2],目前已经有2 600多种血清型被确认。受污染的动物源食品,如家禽肉、鸡蛋和乳制品,被认为是沙门氏菌感染的主要来源[3-4]。据世界卫生组织报道,沙门氏菌每年导致全球9 380多万例胃肠炎,以及155 000多人死亡[5]。肠炎沙门氏菌与鼠伤寒沙门氏菌一直被列为世界上与人类疾病相关的最常见的2 种血清型。这2 种血清型占全球人类沙门氏菌病的比例超过40%,在中国比例超过30%[6],在美国这一比例约为20%[7]。肠炎沙门氏菌感染的临床表现多样,包括自限性小肠结肠炎、急性胰腺炎及具有高死亡率的侵袭性感染[8]。鼠伤寒沙门氏菌感染能够导致肠胃炎及血流感染,可感染肝脏及脾脏等器官[9]。
过去几十年,随着抗生素的频繁使用,耐药性沙门氏菌的出现及传播对公共卫生构成了极大威胁,且多重耐药菌株也已经普遍存在。研究表明,我国禽源沙门氏菌对氨苄西林、四环素、萘啶酸等的耐药率均超过50%[10];在美国,同时耐受氨苄西林、链霉素、新诺明和四环素的多重耐药菌株比例从2009—2013年的1.1%上升到2014—2018年的2.6%[11]。沙门氏菌还携带有多种毒力基因,如毒力岛、鞭毛、菌毛和肠毒素等,毒力基因复杂多样;正是由于沙门氏菌多种毒力基因的表达导致沙门氏菌致病性的多样化,同时由于沙门氏菌携带多种质粒等可移动原件,导致其耐药基因及毒力基因的传播[12]。
本研究利用全基因组测序手段分析2020—2023年68 株分离自生畜肉中的沙门氏菌,鉴定血清型,检测药物敏感性,从全基因组水平分析其耐药基因和毒力因子以及序列型(sequence typing,ST)与耐药和毒力的关系,为了解生畜肉中沙门氏菌的流行情况及其防控提供依据。
1 材料与方法
1.1 材料与试剂
本研究所用沙门氏菌菌株分离自2020—2023年风险监测项目生畜肉,包括牛肉、羊肉和猪肉,其中2020年采集的159 份样本分离出13 株、2021年采集的262 份样本分离出20 株、2022年采集的275 份样本分离出15 株、2023年采集的256 份样本分离出20 株。
1.2 仪器与设备
GelDoc XR Biorad凝胶成像系统 伯乐生命医学产品(上海)有限公司;DH209L电热恒温培养箱 天津泰斯特仪器有限公司;Phoenix-M50全自动微生物鉴定药敏分析系统、Phoenix革兰氏阴性药敏检测板 碧迪医疗器械(上海)有限公司;沙门氏菌鉴定分型诊断血清套装 丹麦国家血清研究院;022070平板计数琼脂干粉广东环凯微生物科技有限公司。
1.3 方法
1.3.1 血清型鉴定
按照血清凝集方法,对沙门氏菌分离株进行血清型鉴定;根据得到的O、H1和H2抗原数据,按照考夫曼-怀特沙门氏菌属抗原表判定沙门氏菌分离株血清型。
1.3.2 药敏实验
利用全自动微生物鉴定药敏分析系统对68 株沙门氏菌进行青霉素类(氨苄西林(ampicillin,AMP))、β-内酰胺类(氨苄西林/舒巴坦(ampicillin/sulbactam,AMS))、头孢类(头孢噻肟(cefotaxime,CTX)、头孢他啶(ceftazidime,CAZ))、磺胺类(复方新诺明(cotrimoxazole,SXT))、四环素类(四环素(tetracycline,TE))、氨基糖苷类(阿米卡星(amikacin,AMI))、大环内酯类(阿奇霉素(azithromycin,AZM))、碳氢霉烯类(厄他培南(ertapenem,ETP)、美罗培南(meropenem,MEM))、苯丙醇类(氯霉素(chloramphenicol,CHL))、氟喹诺酮类(环丙沙星(ciprofloxacin,CIP)、萘啶酸(nalixic acid,NAL))10 大类、13 种抗菌药物进行最小抑菌浓度实验。耐3 类及以上药物的菌株为多耐药菌株,耐5 类及以上药物的菌株为高耐药菌株,药敏结果参照美国临床和实验室标准化协会规定的标准判定是否耐药。
1.3.3 全基因组多位点序列分型(complete genome multilocus sequencetyping,cgMLST)实验
按照Stepan等[13]的方法将68 株全基因组测序后的沙门氏菌上传至http://enterobase.warwick.ac.uk/species/senterica/allele_st_search网站,比对aroC、dnaD、hemD、hisD、purE、sucA和thrA 7 个管家基因,查询管家基因的基因位点编号,由7 个管家基因位点编号判断分离株的ST。将68 株沙门氏菌的全基因组测序拼接草图上传至http://bacdb.cn网站[14],用cgMLST V2算法构建最小生成树和聚类树,将全基因组序列上传至https://cge.cbs.dtu.dk//services/ResFinder/进行耐药基因的注释(一致性≥90%,覆盖度≥60%),毒力基因的注释与分类在http://www.mgc.ac.cn/cgi-bin/VFs/v5/main.cgi网站完成。
1.4 数据处理
数据处理与分析利用Origin 8.0和Chiplot在线作图软件完成[15],差异显著性分析采用卡方检验法。
2 结果与分析
2.1 血清型与多位点序列分型(multilcous seq uence typing,MLST)结果
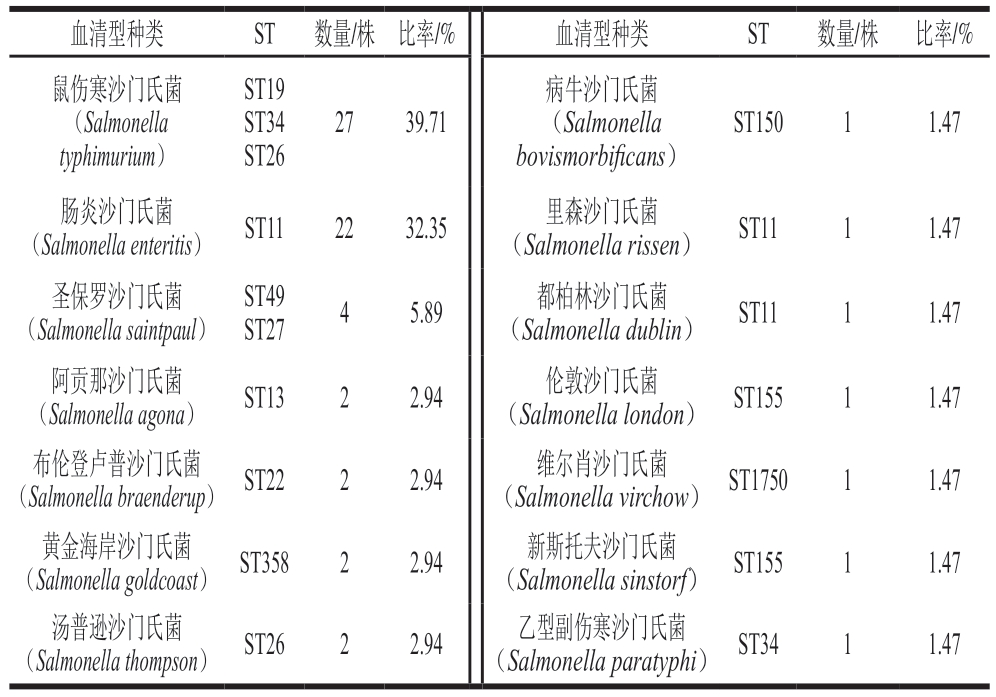
如表1所示,68 株沙门氏菌分离株经鉴定分属14 种血清型。其中,27 株为鼠伤寒沙门氏菌,占比39.71%,22 株为肠炎沙门氏菌,占比32.35%,为2 种优势血清型;其次为圣保罗沙门氏菌(4/68,5.89%),阿贡那沙门氏菌、布伦登卢普沙门氏菌、黄金海岸沙门氏菌和汤普逊沙门氏菌各2 株,占比2.94%,病牛沙门氏菌、里森沙门氏菌、都柏林沙门氏菌、伦敦沙门氏菌、维尔肖沙门氏菌、新斯托夫沙门氏菌和乙型副伤寒沙门氏菌均为1 株,占比1.47%。68 株沙门氏菌经比对鉴定有12 种ST,鼠伤寒沙门氏菌(n=27)的ST主要为ST19(n=15)和ST34(n=11),另外还有ST26(n=1);肠炎沙门氏菌(n=22)的ST为ST11(n=22);圣保罗沙门氏菌(n=4)的ST有ST49(n=2)和ST27(n=2);阿贡那沙门氏菌(n=2)、布伦登卢普沙门氏菌(n=2)、黄金海岸沙门氏菌(n=2)和汤普逊沙门氏菌(n=2)的ST分别为ST13(n=2)、ST22(n=2)、ST358(n=2)、ST26(n=2)。
表1 2020—2023年68 株沙门氏菌血清型
Table 1 Serotypes of 68 Salmonella strain
如图1所示,本研究从生畜肉中分离到的沙门氏菌血清型以鼠伤寒沙门氏菌占比最多,其次为肠炎沙门氏菌,这与吴海晶等[16]从猪肉中分离到的鼠伤寒沙门氏菌占比最多保持一致,其余血清型存在一定的差异性,本研究中发现的沙门氏菌优势血清型为鼠伤寒沙门氏菌和肠炎沙门氏菌,流行ST为ST19、ST34和ST11,这与张捷[17]、毛旭建[18]等报道我国流行的沙门氏菌优势血清型基本相同,与Turcotte等[19]报道美国分离的沙门氏菌ST19、ST11、ST32型基本相同,血清型存在一定差异。由此可知,我国生畜肉中目前流行的沙门氏菌以鼠伤寒沙门氏菌和肠炎沙门氏菌为主,ST19和ST11为流行沙门氏菌ST。
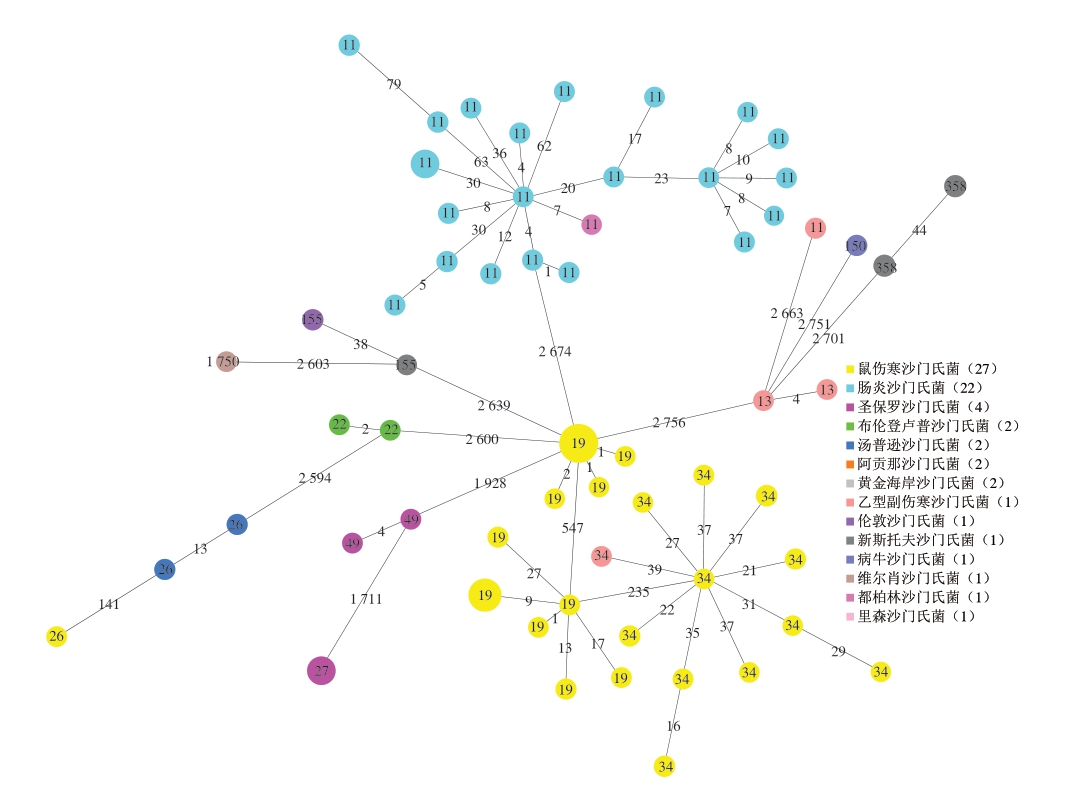
图1 2020—2023年68 株沙门氏菌最小生成树
Fig.1 Minimum spanning tree of 68 Salmonella strains
圆圈内数字代表对应的菌株ST。
2.2 耐药性与耐药基因测定结果
如图2所示,68 株沙门氏菌对AMP的耐药率最高,为76.47%;其次为TET和NAL,耐药率为55.88%和52.94%;对CHL、SXT和AMS的耐药率均在30%以上,对CTX和AZM的耐药率约为20%,对CAZ和CIP的耐药率约为15%;68 株沙门氏菌对碳氢霉烯类ETP和MEM以及氨基糖苷类AMI均无耐药性;虽然对CIP的耐药率较低,但是中介占比较高,达到64.71%,对AMS的耐药中介占比为41.18%。
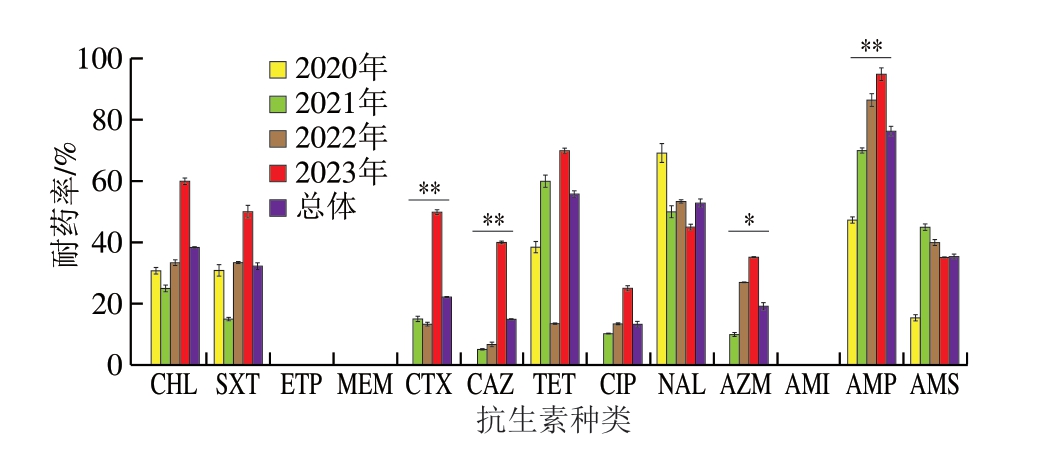
图2 2020—2023年68 株沙门氏菌耐药情况
Fig.2 Drug resistance of 68 Salmonella strains
2020年与2023年相比,*.差异显著(P<0.05);**.差异极显著(P<0.01)。
如表2所示,2020—2023年共47 株为多耐药菌株,占69.12%,其中2020年6 株(46.15%)、2021年13 株(65.00%)、2022年10 株(66.67%)、2023年18 株(90.00%);2020—2023年共16 株为高耐药菌株,占23.53%,其中2020年0 株(0%)、2021年4 株(20.00%)、2022年3 株(20%)、2023年9 株(45.00%);多耐药和高耐药菌株占比呈现出一定的上升态势。耐药谱为CHL-SXT-TET-AMP的菌株有10 株(14.71%),是耐药谱中占比最多的,有6 株菌对检测用药物全部敏感。
表2 2020—2023年68 株沙门氏菌多重耐药情况
Table 2 Multi-drug resistance of 68 Salmonella strains
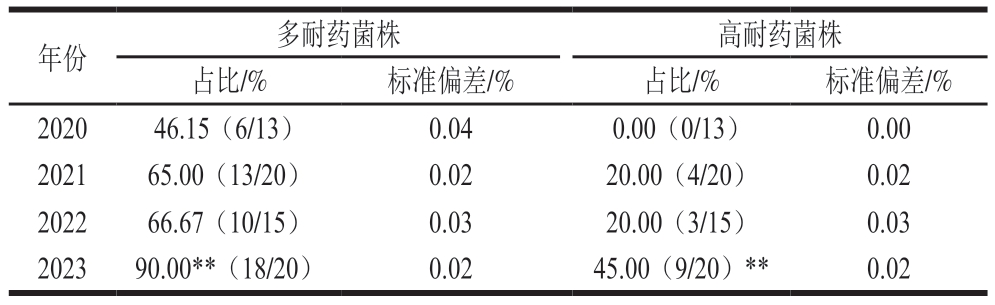
注:2020年与2023年相比,**.差异极显著(P<0.01)。
本研究选取10 大类13 种抗生素进行药物敏感实验,结果表明,沙门氏菌对AMP、TET和NAL耐药较严重,3 重及以上耐药菌株占比69.12%(47/68),说明生畜肉中沙门氏菌耐药严重,这与朱坤鹏等[20]报道广东鸡肉源沙门氏菌耐药性相似,与王雪娇等[21]报道天津市滨海新区食源性疾病主动监测沙门氏菌耐药结果也类似。本研究中所分离的沙门氏菌对碳氢霉烯类ETP和MEM以及氨基糖苷类AMI均无耐药性,这与刘建忠等[22]报道的福建泉州引起食源性腹泻的沙门氏菌对AMI耐药严重不一致,这说明沙门氏菌的耐药性与地域有一定关系。由此可见,生畜肉中沙门氏菌对青霉素类AMP、四环素类TET和NAL耐药严重,提示在生畜养殖中要加强对这3 类抗生素的使用管理。同时本研究中的沙门氏菌对碳氢霉烯类ETP和MEM无耐药性,提示由沙门氏菌引起的生畜疾病中可合理使用碳氢霉烯类抗生素治疗管理。
如表3所示,大部分菌株对检测用的抗生素耐药性均由1~2 种耐药基因所介导。对CHL耐药的26 株沙门氏菌中,出现3 种耐药基因型,主要基因型为floR。对SXT耐药的22 株沙门氏菌中,出现4 种耐药基因型,主要为sul2。对碳氢霉烯类ETP和MEM耐药菌株数量为0,同时也没有检测到相应的耐药基因。对头孢类CTX和CAZ耐药的菌株分别为15、10 株,出现耐药基因型5 种,主要为blaCTX-M-14和blaCTX-M-55。对TET耐药的38 株沙门氏菌中发现3 种基因型,主要为tet(A)。对氟喹诺酮类CIP和NAL耐药的菌株分别为9、36 株,CIP的耐药基因型有3 种,qnrS1为主要基因型,未检测到NAL的对应耐药基因。对AZM和AMI耐药的菌株与基因型一致性较低,对AZM耐药但只有1 株检测到对应耐药基因,68 株沙门氏菌均携带aac(6’)-Iaa基因,但对AMI均无耐药性。对AMP和AMS耐药的菌株分别检出8、4 种基因型,均以blaTEM-1B为主要基因型。
表3 68 株沙门氏菌耐药基因型分布
Table 3 Distribution of drug resistance genotypes of 68 Salmonella strains
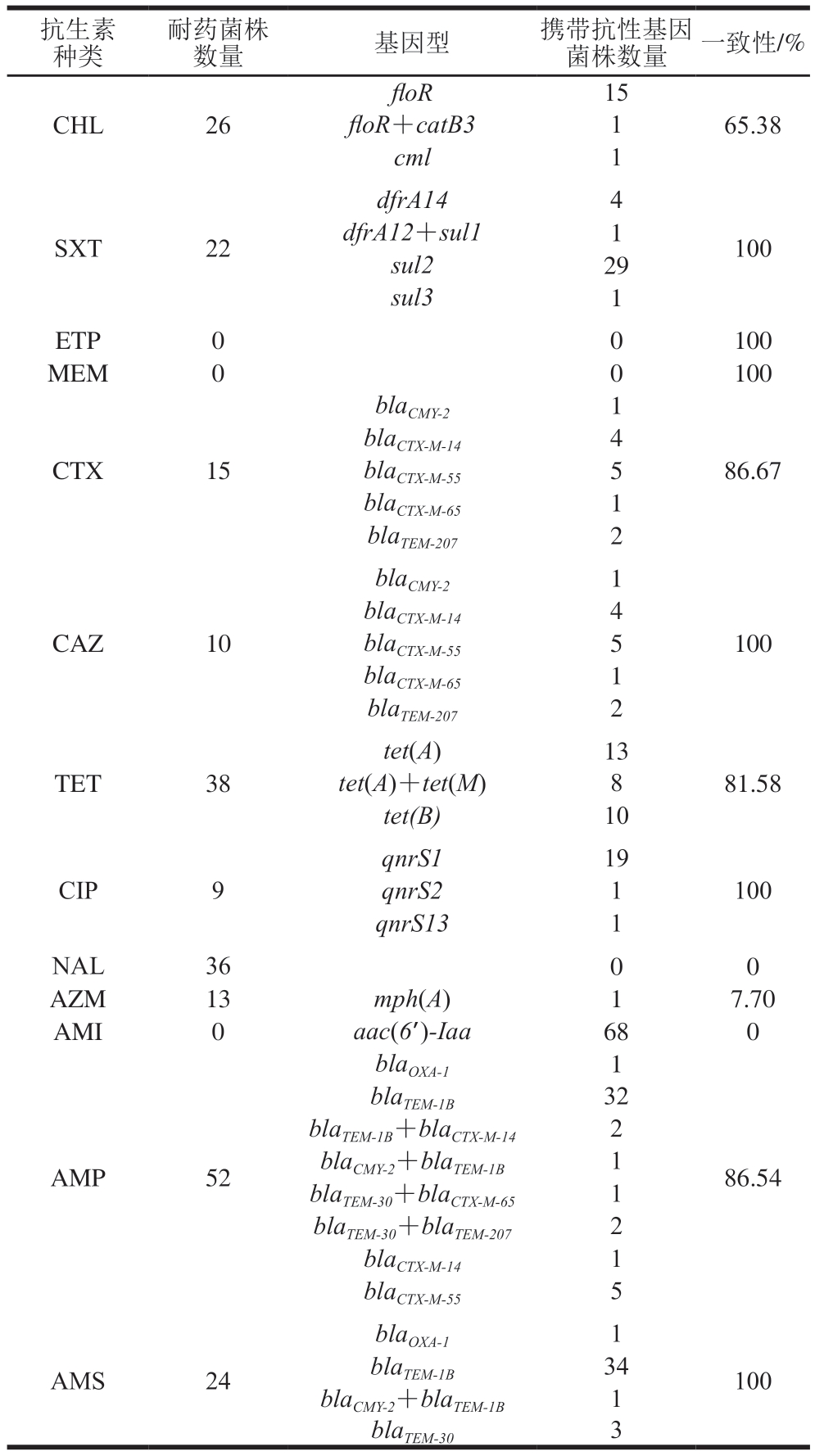
本研究中大部分沙门氏菌的耐药表型均由1~2 种耐药基因所介导,即大部分菌株的耐药性由基因水平介导。这与华德等[23]报道的沙门氏菌耐药基因与耐药表型基本一致的结论相符,该研究中的63 株沙门氏菌均携带aac(6’)-Iaa基因,只有6 株具有对氨基糖苷类庆大霉素的耐药性,与本研究中结果类似;这表明aac(6’)-Iaa基因在氨基糖苷类药物耐药中不是起到主要作用。本研究中68 株沙门氏菌对AZM耐药的有13 株,耐药率为19.12%,只有1 株发现相关的耐药基因,这与Chiou等[24]报道76 株AZM耐药沙门氏菌只有1 株未检测到相关耐药基因结果不一致,但他们也发现有26 株中介耐药菌株未检测到相关耐药基因;AZM和碳氢霉烯类药物是临床上治疗多重耐药沙门氏菌引起的侵袭性沙门氏菌病的最后手段,非基因水平介导的AZM耐药机制值得深入探究。由此可知,生畜中沙门氏菌的耐药基本由基因水平介导,需要加强在生畜养殖中抗生素的合理使用和管理,同时对于非基因水平介导的抗生素抗性需要进一步研究其机制,不同菌株间是否具有转移性和共性。
2.3 全基因组毒力因子分析
为了解68 株沙门氏菌的致病性谱,将68 株沙门氏菌全基因组测序结果上传至VFDB网站,所得注释结果经统计后如图3所示,68 株沙门氏菌所携带的毒力基因按照功能可分为11 大类,分别为:菌毛吸附、镁离子转运、非菌毛吸附、调控相关、分泌系统、前噬菌体、压力适应性、血清抗性、毒素、转运蛋白相关因子和侵袭素。其中,68 株沙门氏菌均携带的毒力因子有菌毛吸附相关:Agf、Bcf、Fim、Stb、Std、Sth、Sti,镁离子转运相关:Mg2+ transport,非菌毛类吸附相关:MisL、SinH,调控因子相关:PhoPQ,分泌系统相关:TTSS(SPI-1 encode)、TTSS(SPI-2 encode)、TTSS effectors、TTSS-1 effectors、TTSS-2 effectors;67 株携带Saf、Stf、Mig-14;65 株携带Lpf、ShdA;63 株携带RatB;57 株携带SodCI;这些毒力因子被大多数沙门氏菌所携带。
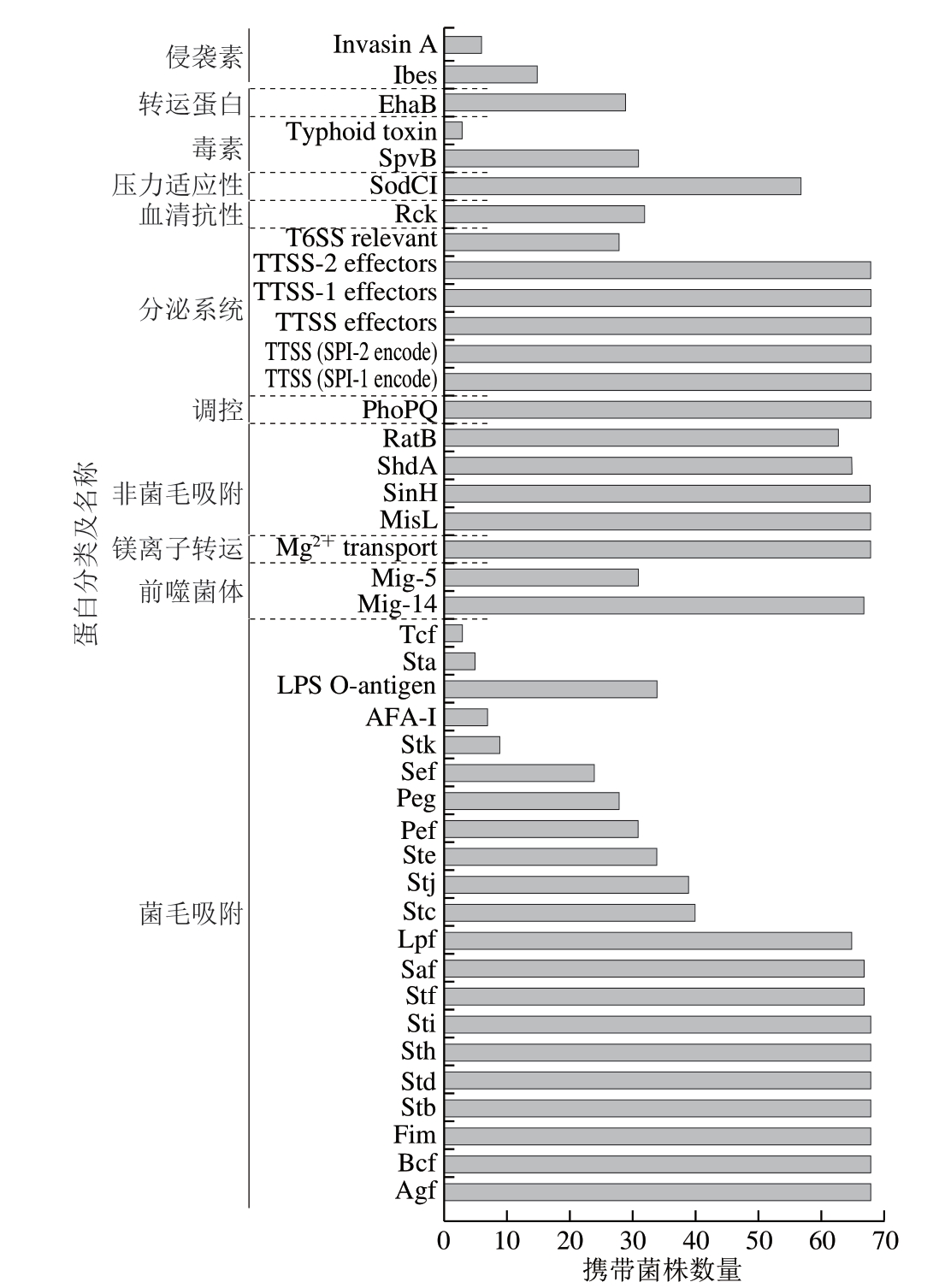
图3 68 株沙门氏菌毒力因子携带情况
Fig.3 Virulence factors of 68 Salmonella strains
此外,3 株携带由cdtB基因编码的伤寒毒素,其中2 株黄金海岸沙门氏菌和1 株病牛沙门氏菌,31 株携带由spvB基因编码的SpvB毒力因子,其中8 株鼠伤寒沙门氏菌占比29.63%(8/27),22 株肠炎沙门氏菌占比100%(22/22),1 株都柏林沙门氏菌;6 株携带Invasin A,其中4 株肠炎沙门氏菌、1 株鼠伤寒沙门氏菌、1 株都柏林沙门氏菌;15 株携带Ibes,其中12 株鼠伤寒沙门氏菌占比44.44%(12/27)、1 株乙型副伤寒沙门氏菌、1 株里森沙门氏菌、1 株都柏林沙门氏菌;7 株携带AFA-I,其中4 株圣保罗沙门氏菌占比100%(4/4)、1 株鼠伤寒沙门氏菌、1 株新斯托夫沙门氏菌、1 株阿贡那沙门氏菌,35 株携带LPS O-抗原,其中21 株肠炎沙门氏菌占比95.45%(21/22),3 株鼠伤寒沙门氏菌;28 株携带EhaB,其中肠炎沙门氏菌21 株,占比95.45%(21/22),29 株携带与VI型分泌系统相关的毒力因子,其中肠炎沙门氏菌22 株,占比100%(22/22),鼠伤寒沙门氏菌2 株。
本研究中68 株沙门氏菌主要毒力因子与吸附、转运、分泌系统及压力适应性等相关,这与Yan Shigan等[25]报道的243 株沙门氏菌毒力因子中与黏附、效应子递送系统、免疫调节、运动和营养/代谢因素相关的菌株占84.63%结果类似;梁丽等[26]报道的鼠伤寒沙门氏菌毒力因子主要与细菌黏附、毒素、分泌系统、血清抗性和免疫逃避等相关,不同菌株之间毒力基因也存在显著差异;与本研究中沙门氏菌毒力因子大部分相同、少部分为个别菌株特有结果一致。这表明沙门氏菌大部分毒力因子趋于稳定,少量毒力因子变化较大。变化较大的毒力因子主要与侵袭、转运、毒素及吸附相关,需要加强对这几类毒力因子的监测分析。
2.4 ST与耐药和毒力关联性分析
已有研究表明,沙门氏菌的毒力和耐药存在一定的关联性。为探究本研究中68 株沙门氏菌ST、毒力和耐药的关系,将沙门氏菌携带的毒力基因和耐药基因、实际耐药重数和ST统计后结合单核苷酸多态性进化树进行分析。如图4所示,系统发育树将68 株沙门氏菌分为4大支,部分ST19为一支、部分ST19与ST34为一支、ST11为一支、其他ST为一支;总体上携带耐药基因数量与实际耐药重数成正比;ST11携带毒力因子最多,ST34携带毒力因子最少。具体来看,ST19进化分为2 个方向:一是携带更少毒力因子,耐药性增强,二是携带更多毒力因子,耐药性减弱;ST34携带毒力因子少但耐药性强;ST11则是携带毒力因子多的同时耐药性也强;ST49耐药性弱,携带毒力因子数量较多;ST27携带耐药基因和毒力因子数量均较多;ST358耐药性强但携带毒力因子数量少;ST13携带毒力因子多且耐药性强;ST155携带耐药基因数量较少但其实际耐药重数较多,且携带毒力因子数量较多;ST22耐药性弱且携带毒力因子较少;2 株ST26耐药性强,携带耐药基因数量较少,1 株ST26则耐药性相对较弱,但毒力因子携带数量较多。
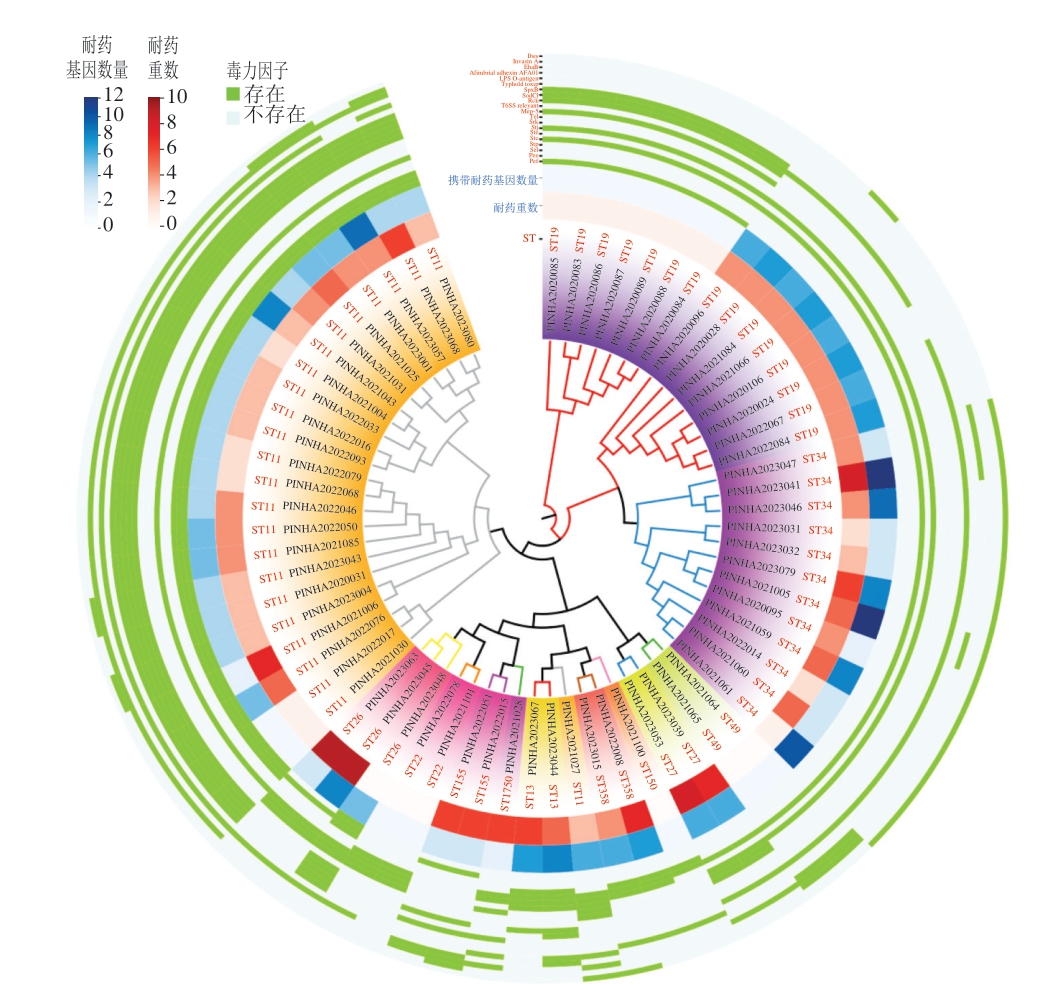
图4 68 株沙门氏菌ST与毒力和耐药性关系图
Fig.4 Relationship between ST and virulence and drug resistance of 68 strains of Salmonella
据报道,沙门氏菌的毒力与携带的毒力因子数量成正比[27-29],这说明ST11型肠炎沙门氏菌实际毒力很强。Wang Xufeng等[30]通过沙门氏菌感染小鼠肠道发现,鼠伤寒沙门氏菌和肠炎沙门氏菌对小鼠肠道的损伤强于其他几种血清型沙门氏菌。Li Yan等[31]报道,在研究的8 种血清型沙门氏菌中肠炎沙门氏菌携带最多种类的质粒,也就意味着肠炎沙门氏菌携带更多种类的耐药基因和毒力基因,与本研究结果一致。这表明肠炎沙门氏菌在向毒力增强和耐药性增强的方向进化,虽然鼠伤寒沙门氏菌和肠炎沙门氏菌是与人类感染密切相关的2 种沙门氏菌,从本研究中的耐药和毒力分析结果来看,肠炎沙门氏菌的威胁性更大。以往监测中主要关注其耐药性的变化,从本研究结果来看,一方面要监测其抗生素抗性的变化,另一方面要监测其毒力基因携带情况的变化。
3 结论
本研究对分离自生畜肉中的68 株沙门氏菌的分型结果表明,鼠伤寒沙门氏菌和肠炎沙门氏菌是生畜肉中沙门氏菌的主要血清型,ST19、ST34和ST11是流行ST。药物敏感性试验结果表明,68 株沙门氏菌多重耐药菌株占比69.12%(47/68),对氨苄西林(76.47%)、四环素(55.88%)和萘啶酸(52.94%)耐药率较高,大部分菌株对所检测用的抗生素耐药性均由1~2 种耐药基因介导。毒力因子分析表明,沙门氏菌携带多种类型的毒力因子,与细菌黏附、毒素、分泌系统、血清抗性和免疫逃避等相关的毒力因子最多。ST与耐药和毒力关联性分析表明,ST11型肠炎沙门氏菌携带更多的毒力因子且耐药性强。综上,市售生畜肉中沙门氏菌血清型和ST分布广泛,耐药性严重同时携带多种毒力因子,提示要加强对畜类饲养环节抗生素的合理使用和生产销售环节的管理。
[1] 罗锦强,骆庆明.儿童非伤寒沙门氏菌感染的临床特点及其对抗菌药物的耐药性临床研究[J].黑龙江医药,2023,36(4): 891-893.DOI:10.14035/j.cnki.hljyy.2023.04.050.
[2] 张楚,王晨蕾,殷荣荣,等.2020—2021年云南省食源性沙门氏菌感染腹泻流行病学特征分析[J].中国人兽共患病学报,2024,40(2):111-115.DOI:10.3969/j.issn.1002-2694.2024.00.017.
[3] XIAO X N,BAI L,WANG S,et al.Chlorine tolerance and crossresistance to antibiotics in poultry-associated Salmonella isolates in China[J].Frontiers in Microbiology,2022,12: 833743.DOI:10.3389/fmicb.2021.833743.
[4] 谭菊,王永娟,张雨馨,等.泰州地区鸡胚源沙门氏菌的分离鉴定及耐药基因和毒力基因的检测[J].黑龙江畜牧兽医,2023,672(12):64-71.DOI:10.13881/j.cnki.hljxmsy.2022.07.0275.
[5] WU L J,LUO Y,SHI G L,et al.Prevalence,clinical characteristics and changes of antibiotic resistance in children with non-typhoidal Salmonella infections from 2009—2018 in Chongqing,China[J].Infection and Drug Resistance,2021,14(1): 1403-1413.DOI:10.2147/IDR.S301318.
[6] ZHAN Z,XU X,GU Z,et al.Molecular epidemiology and antimicrobial resistance of invasive non-typhoidal Salmonella in China,2007—2016[J].Infection and Drug Resistance,2019,12(12):2885-2897.DOI:10.2147/IDR.S210961.
[7] CAPUANO F,MANCUSI A,CAPPARELLI R,et al.Characterization of drug resistance and virulotypes of Salmonella strains isolated from food and humans[J].Foodborne Pathogens and Disease,2013,10(11):963-968.DOI:10.1089/fpd.2013.1511.
[8] 侯悦,刘洁.肠炎沙门氏菌中侵袭相关毒力基因研究进展[J].食品安全质量检测学报,2021,12(10): 3867-3873.DOI:10.19812/j.cnki.jfsq11-5956/ts.2021.10.001.
[9] 吴娅芳,王英林,田亚晨,等.鼠伤寒沙门氏菌感染机制及其检测方法研究进展[J].工业微生物,2022,52(3): 41-48.DOI:10.3969/j.issn.1001-6678.2022.03.007.
[10] 沈海玉,窦新红,秦爱建.江苏部分地区鸡胚源沙门氏菌的分离鉴定及耐药性分析[J].中国家禽,2023,45(10): 109-115.DOI:10.16372/j.issn.1004-6364.2023.10.016.
[11] PLUMB I D,BROWN A C,STOKES E K,et al.Increased multidrug-resistant Salmonella enterica I Serotype 4,[5],12:i:-infections associated with pork,United States,2009–2018[J].Emerging Infectious Diseases,2023,29(2): 314-322.DOI:10.3201/EID2902.220950.
[12] 王天,徐红红,张林吉,等.质粒介导的印第安纳沙门氏菌耐药机制及分子溯源研究[J].中国人兽共患病学报,2022,38(7): 619-625.DOI:10.3969/j.issn.1002-2694.2022.00.076.
[13]STEPAN R M,SHERWOOD J S,PETERMANN S R,et al.Molecular and comparative analysis of Salmonella enterica senftenberg from humans and animals using PFGE,MLST and NARMS[J].BMC Microbiology,2011,11: 153-162.DOI:10.1186/1471-2180-11-153.
[14] ZHI R,YE F.BacWGSTdb,a database for genotyping and source tracking bacterial pathogens[J].Nucleic Acids Research,2016,44(D1):682-687.DOI:10.1093/nar/gkv1004.
[15] XIE J,CHEN Y,CAI G,et al.Tree visualization by one table(tvBOT): a web application for visualizing,modifying and annotating phylogenetic trees[J].Nucleic Acids Research,2023,51(W1): 587-592.DOI:10.1093/nar/gkad359.
[16] 吴海晶,程逸宇,沈威,等.南京地区猪肉源中沙门氏菌分子分型及耐药性分析[J].现代食品科技,2023,39(10): 307-314.DOI:10.13982/j.mfst.1673-9078.2023.10.1362.
[17] 张捷,畅晓晖,柳明,等.禽类中沙门氏菌MLST分型及毒力基因筛查[J].食品研究与开发,2021,42(13): 174-179.DOI:10.12161/j.issn.1005-6521.2021.13.026.
[18] 毛旭建,屠博文,薛银刚,等.食源与人源性沙门氏菌的血清和耐药水平差异分析[J].公共卫生与预防医学,2021,32(3): 63-67.DOI:10.3969/j.issn.1006-2483.2021.03.015.
[19] TURCOTTE M R,SMITH J T,LI J F,et al.Genome characteristics of clinical Salmonella enterica population from a state public health laboratory,New Hampshire,USA,2017–2020[J].BMC Genomics,2022,23(1): 537.DOI:10.1186/s12864-022-08769-1.
[20] 朱坤鹏,向莹,邱少富,等.2018年广东省广州市鸡源和猪源鼠伤寒沙门菌耐药性及分子特征分析[J].疾病监测,2023,38(5): 567-573.DOI:10.3784/jbjc.202302150043.
[21] 王雪娇,刘钦,赵苗苗,等.2016—2020年天津市滨海新区食源性疾病主动监测沙门氏菌分析[J].现代疾病预防控制,2023,34(5): 396-399.DOI:10.13515/j.cnki.hnjpm.1006-8414.2023.05.018.
[22] 刘建忠,叶聿程,陈韵妍,等.2013—2020年福建省泉州市食源性疾病沙门氏菌血清型及耐药分析[J].预防医学论坛,2023,29(12):915-918.DOI:10.16406/j.pmt.issn.1672-9153.2023.12.07.
[23] 华德,王鲁彦,邝仕壮,等.2020年海南省人源沙门菌耐药性及携带耐药基因分析[J].疾病监测,2023,38(6): 722-728.DOI:10.3784/jbjc.202210110440.
[24] CHIOU C S,HONG Y P,WANG Y W,et al.Antimicrobial resistance and mechanisms of azithromycin resistance in nontyphoidal Salmonella isolates in Taiwan,2017 to 2018[J].Microbiology Spectrum,2023,11(1): e0336422.DOI:10.1128/spectrum.03364-22.
[25] YAN S G,LIU X,LI C Y,et al.Genomic virulence genes profile analysis of Salmonella enterica isolates from animal and human in China from 2004 to 2019[J].Microbial Pathogenesis,2022,173:105808.DOI:10.1016/j.micpath.2022.105808.
[26] 梁丽,石继春,陈驰,等.不同时期分离的鼠伤寒沙门氏菌全基因组学分析研究[J].中国药事,2022,36(12): 1403-1413.DOI:10.16153/j.1002-7777.2022.12.010.
[27] 程琼,庞瑞亮,王若晨,等.不同源沙门氏菌对小鼠致病力的比较与毒力基因检测[J].中国人兽共患病学报,2013,29(5): 460-465.DOI:10.3969/cjz.j.issn.1002-2694.2013.05.008.
[28] 施开创,黎宗强,屈素洁,等.鸡源沙门氏菌致病基因与致病性的相关性研究[J].基因组学与应用生物学,2020,39(6): 2513-2520.DOI:10.13417/j.gab.039.002513.
[29] 刘理慧,储锦华,隋雨欣,等.沙门氏菌中主要毒力因子的研究进展[J].生物技术通报,2022,38(9): 72-83.DOI:10.13560/j.cnki.biotech.bull.1985.2022-0687.
[30] WANG X F,XU H H,WANG Y,et al.Systematic evaluation of potential pathogenicity of Salmonella indiana[J].Veterinary Microbiology,2020,247: 108759.DOI:10.1016/j.vetmic.2020.108759.
[31] LI Y,KANG X,ED-DRA A,et al.Genome-based assessment of antimicrobial resistance and virulence potential of isolates of nonpullorum/gallinarum Salmonella serovars recovered from dead poultry in China[J].Microbiology Spectrum,2022,10(4): e0096522.DOI:10.1128/spectrum.00965-22.