近年来,鸡肉及鸡肉制品颇受人们欢迎,鸡肉主要分为热鲜肉、冰鲜肉和冷冻肉,冰鲜鸡肉是宰后将胴体迅速冷却到0~4 ℃,并且在加工、运输和销售过程,温度始终保持0~4 ℃的生鲜鸡肉。冰鲜鸡肉以品质鲜嫩、高营养等优点,成为人们生活中必不可少的肉食选择,鸡肉已然成为餐桌上的主要肉食[1]。由于冰鲜鸡肉中蛋白质含量、水分活度较高,是微生物生长繁殖的天然培养基,因此在屠宰、分割、预冷等生产加工环节以及车辆运输和贮藏、销售过程中极易受到微生物污染[2-3]。相关研究发现,冰鲜鸡肉在冷藏条件下(0~4 ℃)不到1 周就会发生腐败,主要表现为发酸、发黏、有菌斑、外观明显变色等[4-6]。腐败菌在肉品贮藏过程中增殖,产生异味,是影响产品货架期的主要微生物[7-8]。
本文通过分析冰鲜鸡肉中的腐败微生物,结合相关研究确定冰鲜鸡肉中优势腐败菌,并综述针对腐败优势菌的传统检测方法和现代检测新技术的研究进展,为冰鲜鸡肉质量检测、贮藏保鲜及高效防腐提供理论基础。
1 冰鲜鸡肉优势腐败菌
由于不同肉的组织形态、屠宰加工方式等不同,众多学者对不同肉的菌群分布及生长趋势研究发现,不同肉中腐败菌群类别及生长趋势显著不同[9-10]。目前很多学者对冰鲜鸡肉腐败优势菌已有相关研究,传统培养方式下优势腐败菌相差不明显,并且变化趋势相一致。孙彦雨[3]结合聚合酶链式反应(polymerase chain reaction,PCR)和变性梯度凝胶电泳(denaturing gradient gel electrophoresis,DGGE)2 种方法形成PCR-DGGE技术,研究冰鲜鸡肉贮藏过程中不同时期的菌相变化,得出乳酸菌、肉杆菌、热杀索丝菌、腐败希瓦氏菌等是冰鲜鸡肉主要优势菌。梁慧等[11]研究表明,从冷鲜鸡肉初始菌中分离出主导菌群为乳酸菌和葡萄球菌,二者所占比例最大,为35%。刘朏[12]采用传统培养结合PCR-DGGE,确定冷鲜鸡优势菌为肠杆菌、假单胞菌、葡萄球菌、不动杆菌、莫拉克斯和洋葱伯克霍尔德属。张秋勤[13]通过结合传统培养和PCR-DGGE技术,确定鸡肉的优势腐败菌为不动杆菌、假单胞菌、肉杆菌和魏斯氏菌属。通过上述研究结果可知,导致冰鲜鸡肉腐败的微生物主要为假单胞菌属、嗜热环丝菌、气单胞菌、乳杆菌属和肠杆菌科等[14]。鉴于传统分离技术和检测新技术之间存在差异,综合2 种检测方法确定导致冰鲜鸡肉腐败的优势菌为热杀索丝菌、假单胞菌、腐败希瓦氏菌、乳酸菌、肉杆菌、不动杆菌属、莫拉氏菌、气单胞菌属及魏斯氏菌属。根据《伯杰氏系统细菌学手册》,冰鲜鸡肉中的优势腐败微生物特性如表1所示。
表1 冰鲜鸡肉中优势腐败菌及其特征
Table 1 Characteristics of dominant spoilage bacteria in chilled chicken
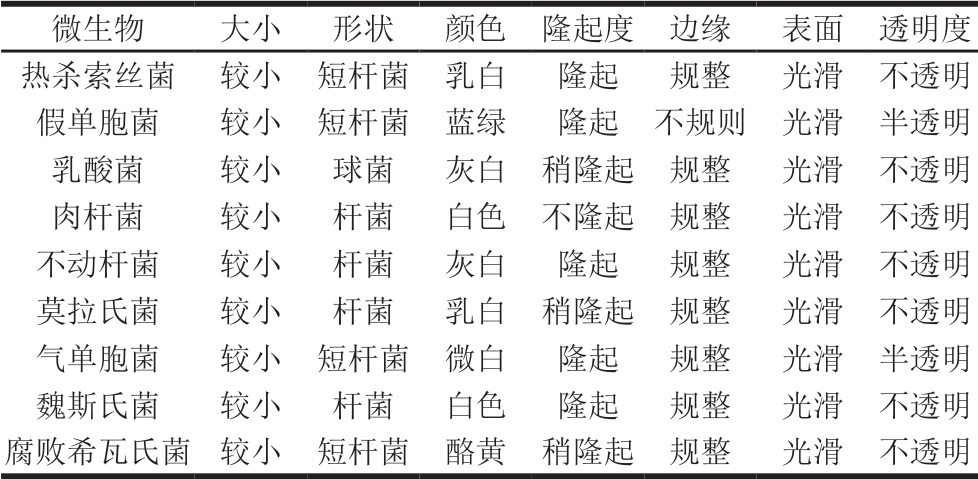
微生物大小形状颜色隆起度边缘表面透明度热杀索丝菌较小短杆菌乳白隆起规整光滑不透明假单胞菌较小短杆菌蓝绿隆起不规则光滑半透明乳酸菌较小球菌灰白稍隆起规整光滑不透明肉杆菌较小杆菌白色不隆起规整光滑不透明不动杆菌较小杆菌灰白隆起规整光滑不透明莫拉氏菌较小杆菌乳白稍隆起规整光滑不透明气单胞菌较小短杆菌微白隆起规整光滑半透明魏斯氏菌较小杆菌白色隆起规整光滑不透明腐败希瓦氏菌较小短杆菌酪黄稍隆起规整光滑不透明
2 传统微生物检测方法
传统微生物检测依据国标4789系列标准,如GB 4789.2—2016《食品安全国家标准 食品微生物学检验菌落总数测定》、GB 4789.35—2016《食品安全国家标准 食品微生物学检验 乳酸菌检验》、GB 4789.15—2012《食品安全国家标准 食品微生物学检验 霉菌和酵母菌计数》、GB 4789.28—2013《食品安全国家标准 食品微生物学检验 培养基和试剂的质量要求》、GB 4789.4—2016《食品安全国家标准 食品微生物学检验 沙门氏菌检验》、GB 4789.14—2016《食品安全国家标准 食品微生物学检验 蜡样芽孢杆菌检验》及GB 4789.13—2012《食品安全国家标准 食品微生物学检验 产气夹膜梭菌检验》。
传统微生物检测主要是为微生物提供适宜的生长环境,如需氧或兼性厌氧条件下pH 6.0~7.5、35~37 ℃培养18~48 h,以及所必需的营养物质,如水分、碳源、氮源等。传统培养包含前增菌培养和分离培养2 个步骤,根据微生物种类,选择不同培养基进行接种、培养,根据菌落形态特征、革兰氏染色结果等再进行下一步的分离和鉴定[15]。传统检测作为经典方法,检测结果准确、直观、成本低、所需设备少,为肉制品加工企业检测首选。但是传统检测方法中存在一些不确定因素,如环境中的微生物对制备的培养基及整个检测过程都有污染风险,进而影响结果;在培养、分离、鉴定过程中,致病菌可能污染空气环境,导致检验人员受到影响,造成一定的生物危害。传统检测方法操作繁琐,需提前配制药品,使用的稀释管、枪头等用品需要高压灭菌,样品处理、稀释、培养、计数等一般需要48 h以上,才能得到分离纯化的单个菌落,再进行鉴定,耗时耗力。而且传统方法鉴定过程不确定因素较多,易被污染,结果准确率低,能够分离和鉴别的菌株种类有限,不能够充分反映所检测样品中微生物的真实性[16]。要想得到准确检测结果的同时缩短检测时间、降低检测成本,就需要充分结合传统分离和现代检测2 种方法,这也是未来食品检测发展的需要。
3 现代检测新技术
3.1 分子检测技术
3.1.1 分子印迹技术
分子印迹技术即利用分子印迹聚合物(molecularly imprinted polymers,MIPs),模拟酶与底物或者抗体与抗原之间的相互作用,对印迹分子进行专一识别的技术[17-18]。基于分子印迹技术制备而成的高分子聚合物可通过三维空间结构和分子间作用力实现与模板分子或类似物的特异性识别,该技术具有专一性、结果准确度高、检测过程快速、整体实用性高等优点,在活性成分分离、模拟酶催化危害物降解等方面进行了广泛研究[19-20]。
Hu Tianliang等[21]基于该原理以大肠杆菌O157:H7为检测模板,建立大肠杆菌O157:H7的电化学发光检测方法。在最优条件下,检测范围为10~107 CFU/mL,检出限为8 CFU/mL。Idil等[22]以大肠杆菌为模板,通过微接触紫外引发印迹聚合方法,制备大肠杆菌MIPs金电极传感器的同时建立电容式电化学检测方法。大肠杆菌的检测范围为102~107 CFU/mL,检出限为70 CFU/mL。Wu Jikui等[23]以大肠杆菌为模板,吡咯为单体,采用电化学聚合方法制备玻璃电极MIPs电化学传感器,并建立了阻抗型电化学检测方法,该方法可在1 h内实现对大肠杆菌O157:H7的检测,检测定量限为103 CFU/mL,回收率为96.0%~107.9%。Fu Kaiyue等[24]采用微接触印迹法在玻璃板上制备副溶血弧菌MIPs膜,该印迹膜对副溶血弧菌具有较好的选择吸附性,捕获率达62.9%。
3.1.2 PCR检测技术
PCR技术的基本原理与DNA复制过程高度相似,是特定DNA分子片段的生物学技术。其特异性来源于与靶序列两端互补的寡核苷酸引物[25-27]。可以通过破碎细胞、提取核酸、PCR扩增、凝胶电泳纯化、核酸测序及数据库比对分析等过程进行鉴定。PCR技术具有样品前处理操作简单、检测周期短、灵敏度高等优点[28]。
DGGE被称为DNA指纹图谱技术,采用PCR-DGGE技术研究鸡肉中微生物,在准确分析微生物菌群及其生长多样性的同时,还能体现微生物在不同生长时期的详细变化[3]。实时荧光定量PCR技术通过扩增反应,在监测荧光信号的基础上,绘制扩增曲线进行深入分析,在实际检测过程中具有极高的精准性和准确度[29-31]。
Li等[32]运用PCR-DGGE技术研究冷藏猪肉,分离出假单胞菌、热杀索丝菌和乳酸菌是主要腐败菌。孙彦雨[3]通过PCR-DGGE技术和传统技术的融合,确定乳酸菌、肉杆菌、热杀索丝菌和腐败希瓦氏菌等是冰鲜鸡肉主要优势菌。李凤珠等[33]采用TaqMan探针和SYBR Green染料2 种方法,实现对金黄色葡萄球菌的快速定量检测,对目前已知变种菌株和相关菌株的检测特异性接近100%。杨曼琼[34]通过荧光定量PCR等技术实现假单胞菌的快速有效检测。
3.1.3 等温扩增技术
等温扩增技术是指可以在恒定的温度下对特定核酸片段进行扩增的技术,本文介绍环介导等温扩增技术和交叉引物扩增技术[35]。
环介导等温扩增技术是一种在恒温条件下进行核酸扩增、能在体外迅速完成对靶序列上亿次扩增的痕量检测技术[36]。环介导等温扩增技术检测方式多样,关键在于找到合适的靶基因并设计相应的特异性引物,从而实现定性检测。Ledlod等[37]通过结合环介导等温扩增技术和双向侧流试纸方法,可快速检测肉及肉制品中单核细胞增生李斯特氏菌,检出限达20 CFU/g。Chen Xingxing等[38]开发了基于等温扩增技术的猪肉制品金黄色葡萄球菌快检方法,检出限达50 CFU/mL。张晋豪等[39]以冷鲜鸡新鲜度与假单胞菌数量的高度正相关性为基础,建立的检测方法对假单胞菌特异性、扩增性显著提高,在最优条件下灵敏度为3.05×102 CFU/mL。
交叉引物恒温扩增(cross priming amplification,CPA)技术能够实现高效扩增目标核酸,但是需要在恒定的温度条件下,通过特殊设计的引物和探针才能实现[40]。CPA技术具有特异性高、操作过程简单、恒温扩增过程迅速、不易被污染、成本低等优点[41]。向勇[42]根据铜绿假单胞菌(Pseudomonas aeruginosa,PA)的特异性基因建立的PA-CPA方法操作简便、特异性强、准确度高,在62 ℃恒温扩增45 min,灵敏度是常规PCR的100 倍,而且重复性和稳定性良好,可用于铜绿假单胞菌的快速检测。
3.1.4 高通量测序技术
高通量测序技术又称为下一代测序技术或大规模平行测序技术[43-44],可同时测序大量核酸分子,该技术在检测普通微生物的同时还能检测到那些不易培养、丰度低以及难以分离的微生物[45-46],与传统微生物学方法相比,能够更好地解释微生物基因的组成和多样性,具有检测快速、通量高、灵敏度高、能够实现样品间平行分析等优点[47]。随着测序技术的发展和成本的下降,高通量测序技术在肉及肉制品腐败微生物检测及菌群多样性方面有着广泛应用[48]。
Xi La等[49]利用Illunima MiSeq高通量测序技术分析麦曲样品中真菌的群落结构和多样性,麦曲中的主要菌门为子囊菌门,平均相对含量达到90.21%。Dong Yun等[50]通过MiSeq高通量测序,分离出腊肉中的主要细菌门为硬壁菌门和变形菌门,平均相对含量分别为54.05%和44.28%。田建军等[51]采用Illumina MiSeq技术分析风干肉制品中细菌16S rRNA V4区基因序列,在自然发酵和人工调控肉制品中分别鉴定出241 个和102 个细菌属,细菌多样性在属水平上差异显著(P<0.05)。
3.2 光谱检测技术
3.2.1 高光谱成像技术
高光谱成像技术是通过融合光谱和图像2 种技术,可以对样品快速、无损、高效检测的光电分析技术,检测过程中能够同时得到样品的光谱和图像信息[52]。光谱波段涵盖范围广,分辨率达到纳米级别,是一种融合光学、计算机、信号处理学的无损、快速、准确、高效的现代化技术[53-54]。
王慧等[55]通过高光谱数据分析所建立鸡肉嫩度预测模型的相关系数为0.94。何鸿举等[56]利用900~1 700 nm近红外高光谱成像系统对冷鲜鸡胸肉的乳酸菌含量进行检测,通过连续投影算法(successive projections algorithm,SPA),根据筛选出的最优波长,建立的SPAPLS模型预测相关系数达到0.949,验证集均方根误差为0.439(lg(CFU/g))。Feng Yaoze等[57]对鸡肉中的肠杆菌科进行研究,通过使用近红外高光谱成像建立的鸡肉中肠杆菌科预测模型相关系数为0.93。何鸿举等[58]基于近红外高光谱成像技术,通过使用基线校正(baseline correction,BC)、偏最小二乘(partial least squares,PLS)算法等,研究热杀索丝菌含量在鸡胸肉冷藏过程中的变化,通过BC预处理,构建的全波段PLS热杀索丝菌预测模型相关系数接近1.0。
3.2.2 近红外光谱技术
近红外光谱是对波长760~2 600 nm、可见光谱区及中红外光谱区间电磁波的描述[59]。近红外光谱技术是一种能够实现预测样品化学组成及其含量的无损检测技术,并且可以关联样品本身的性质特征,来建立校正模型。近红外光谱源于分子振动,可反映分子化学键基频振动的倍频和合频吸收,结合化学计量学方法PLS[60]用于食品质量检测,能够实现快速、无损、多组分同时检测,广泛应用于鲜猪肉、鲜鸡肉等肉类及肉制品的检测中[61]。
Alexandrakis等[62]利用近红外光谱技术定性检测鸡胸肉中李斯特菌、荧光假单胞菌、恶臭假单胞菌和大肠杆菌。张雷蕾[63]通过近红外高光谱成像技术,建立冷却猪肉中假单胞菌的预测模型,相关系数为0.948。王名星[64]运用嗅觉可视化及近红外光谱技术,通过多传感器信息融合技术分离鉴定鸡肉中假单胞菌,建立的反向传播人工神经网络模型识别效果显著提升。
3.2.3 拉曼光谱技术
拉曼光谱技术是一种分子水平的光谱分析技术。样品被激光照射后,大部分光以与入射光相同的频率发生散射(瑞利散射),但是仍有少量入射光以不同于入射光的频率发生散射,即拉曼散射[65]。拉曼光谱技术具有样品前处理简单无损、检测过程快速、全程操作简便等特点。但是拉曼光谱易受干扰,导致信号较弱,实现痕量物质的快速检测可通过表面增强拉曼,即通过贵金属表面糙化处理进而增强拉曼信号[66]。
Ma Xiaoyuan等[67]在多刺纳米金粒子上修饰巯基化适配体作为表面增强拉曼纳米探针,检测猪肉中沙门氏菌,最优条件下检测限可达4 CFU/mL。Zhang Hui等[68]用适配体固定化磁性纳米金粒子捕获猪肉中金黄色葡萄球菌,优化后表面增强拉曼法检出限为35 CFU/mL,加标回收率为94.12%~102.75%。Argyri等[69]使用拉曼光谱结合偏最小二乘回归分析透氧和气调2 种包装的牛肉馅5 ℃贮藏期间的腐败情况,精确检测出菌落总数、乳酸菌数和肠杆菌数。杨鸿博[70]采用拉曼光谱技术预测贮藏21 d的牛肉中菌落总数和乳酸菌数的能力相关系数为0.75~0.99,验证集均方根误差为0.38~0.61。
3.3 免疫分析法
3.3.1 酶联免疫法
酶联免疫吸附测定(enzyme-linked immunosorbent assay,ELISA)是在保持抗体(抗原)活性的基础上连接到固相载体或酶上制成的功能化载体或酶标抗体(酶标抗原)[71]。通过建立功能化载体以催化底物检测体系,逐步实现利用颜色变化判断致病菌的种类及数量。该方法还可以检测抗体、其他大分子及小分子物质[72]。但是存在试剂盒需低温存放、昂贵、寿命短、检测中酶的活性易受环境影响、重复性和准确性差等问题。
谢珲等[73]建立黄曲霉毒素B1(aflation B1,AFB1)间接竞争ELISA检测法,检测限为1.04 μg/L,通过该方法与进口试剂盒检测结果相比,具有较好的重复性,可以用于快速筛检饲料中AFB1。姚永忠等[74]在食品质量安全检测工作中,采用生物素-亲和素斑点-ELISA法检测李氏杆菌,检测灵敏度达到104 个/mL,并且整体检测过程易于操作,不易受环境污染,检测结果准确度高。
3.3.2 胶体金免疫层析法
胶体金免疫层析法是将已知的抗原或抗体固定在具有特异性的载体上,利用抗原-抗体之间的显色反应,通过标记、免疫反应及色层分析法等形成的新型免疫检测技术[75]。其优势为检测结果直观、样品前处理方便、抗原-抗体特异性强、仪器和试剂价格较低等[76]。Song Chunmei等[77]开发的胶体金免疫层析试纸条,可用于检测肉冻等食品中志贺氏菌和大肠杆菌O157:H7,具有检测快速、结果准确度高、价格低廉的优点。
检测新技术应用优缺点详见表2。
表2 检测新技术在腐败菌中的应用
Table 2 Application of new technologies in detecting spoilage bacteria

新技术检测微生物检测样品优点缺点参考文献分子印迹技术假单胞菌、肠杆菌食品具有特异识别性、准确、快速非特异性吸附问题突出、基质对吸附性能及敏感度影响大[17,21,24]技术假单胞菌冷鲜鸡肉快速、简便、灵敏度高预处理复杂、试剂昂贵、人员要求较高[35,39,42]高通量测序技术等温扩增假单胞菌、不动杆菌、乳酸菌、魏斯氏菌肉制品检测快速、通量高、灵敏度高、能够实现样品间平行分析引物要求高、数据量大[50-51]拉曼光谱技术假单胞菌、肠杆菌、乳酸菌牛肉易操作、准确度高、重复性和稳定性好引物设计要求较高、易形成气溶胶、造成假阳性[65-66,70]高光谱成像技术乳酸菌、热杀索丝菌、肠杆菌、假单胞菌冷鲜鸡肉无损、快速、准确、高效数据量大、信息多、数据分析较困难[52,56,58]光谱技术假单胞菌鸡肉快速、无损、多组分同步检测近红外光谱带宽大、强度低、特征峰重叠、易受干扰[59,61]PCR技术乳酸菌、热杀索丝菌、腐败希瓦氏菌、肉杆菌、假单胞菌冰鲜鸡肉操作简便、快速、准确信号易受干扰、影响光谱形态、应用有选择性[26-27,31]免疫分析法假单胞菌饲料简便快速、特异性强、稳定性好、检测范围广试剂昂贵、需低温存放、酶活性易受影响、重复性、准确性差[71,73,75]
4 结 语
本文综述冰鲜鸡肉中的腐败微生物,确定冰鲜鸡肉中优势腐败菌,分析不同类型检测新技术的研究进展及应用情况,均能够实现快速、无损、高效检测,但是新技术仍有诸多不足。PCR检测技术样品前处理复杂,检测过程易受环境和操作污染,所需试剂较贵、对检测人员要求较高;高光谱技术采集信息更全面的同时数据量大、干扰信息多、数据分析较繁琐;近红外光谱技术检测过程中易出现光谱带宽大、强度低、特征峰重叠、检测易受干扰等情况。在我国,新技术、仪器商品化程度低,多数处于实验室研究阶段,不能满足实际生产检测需求。目前许多食品生产企业在实际生产中腐败菌、致病菌等微生物的分离检测仍以传统方法为主,新技术使用率较低。
未来我国微生物检测发展方向应为操作简便化、仪器产品化,通过各类学科技术的交叉与引进,形成现代化食品微生物检测技术体系,满足社会、企业等实际生产需要,不断提高新技术的整体适用性,推动微生物检测的发展,并广泛应用到食品卫生检测的各个领域。
[1] 胡三梅.肉及肉制品微生物检测新技术研究进展[J].肉类研究,2022, 36(5): 62-67.DOI:10.7506/rlyj1001-8123-20220328-027.
[2] 樊静.鸡肉及其生鲜调理制品货架期预测模型的建立[D].郑州: 河南农业大学, 2016: 2-48.
[3] 孙彦雨.冰鲜鸡肉腐败微生物分析及其减菌剂的研究[D].南京: 南京农业大学, 2011: 8-60.
[4] LEE K, PARK H, BAEK S, et al.Colorimetric array freshness indicator and digital color processing for monitoring the freshness of packaged chicken breast[J].Food Packaging and Shelf Life, 2019, 22:100408.DOI:10.1016/j.fpsl.2019.100408.
[5] 侯芮.引起鸡肉腐败的微生物及延缓腐败的措施[J].科技风,2012(14): 227.DOI:10.19392/j.cnki.1671-7341.2012.14.190.
[6] 张德权, 惠腾, 王振宇.我国肉品加工科技现状及趋势[J].肉类研究,2020, 34(1): 1-8.DOI:10.7506/rlyj1001-8123-20191029-256.
[7] KATIYO W, DE K H L, COOREY R, et al.Sensory implications of chicken meat spoilage in relation to microbial and physicochemical characteristics during refrigerated storage[J].LWT-Food Science and Technology, 2020, 128: 109468.DOI:10.1016/j.lwt.2020.109468.
[8] YE Keping, JIANG Jian, WANG Yuefan, et al.Microbial analysis of MAP pot-stewed duck wings under different conditions during 15 ℃storage[J].Journal of Food Science and Technology, 2017, 54(5):1073-1079.DOI:10.1007/s13197.
[9] SHAO L T, CHEN S S, WANG H H, et al.Advances in understanding the predominance, phenotypes, and mechanisms of bacteria related to meat spoilage[J].Trends in Food Science and Technology, 2021, 118:822-832.DOI:10.1016/j.tifs.2021.11.007.
[10] YE Keping, LIU Mei, LIU Jia, et al.Microbial diversity of different modified atmosphere packed pot-stewed duck wings products during 8 ℃storage[J].Letters in Applied Microbiology, 2017, 64(3): 225-230.DOI:10.1111/lam.12704.
[11] 梁慧, 于立梅, 陈秀兰, 等.鸡胸肉冷藏过程中腐败菌分析及其品质变化研究[J].食品与发酵工业, 2016, 42(10): 184-188.DOI:10.13995/j.cnki.11-1802/ts.201610030.
[12] 刘朏.不同贮藏温度对冷鲜鸡微生物和肉品品质的影响研究[D].杨凌: 西北农林科技大学, 2015: 8-13.
[13] 张秋勤.生鲜鸡肉中腐败菌群体感应信号分子研究[D].南京: 南京农业大学, 2014: 1-29.
[14] 赖宏刚, 蒋云升, 张元嵩, 等.真空包装冷鲜鸡中腐败菌微生物的分离鉴定[J].江苏农业科学, 2018, 46(17): 198-201.DOI:10.15889/j.issn1002-1302.2018.17.053.
[15] 李乐.致病微生物新型检测方法及预防体系应用研究[D].长沙: 中南大学, 2014: 6-15.
[16] GAROFALO C, BANCALARI E, MILANOVIĆ V, et al.Study of the bacterial diversity of foods: PCR-DGGE versus LH-PCR[J].International Journal of Food Microbiology, 2017, 242(2): 24-36.DOI:10.1016/j.ijfoodmicro.2016.11.008.
[17] 任涛涛, 陈金媛, 靳雨婷, 等.分子印迹技术在食品腐败菌与致病微生物检测中的应用[J].中国食品学报, 2022, 22(6): 435-444.DOI:10.16429/j.1009-7848.2022.06.044.
[18] GUO Mingkun, GAO Yuhang, CHEN Ge, et al.Recent advances and future trends in the detection of contaminants by molecularly imprinted polymers in food samples[J].Frontiers in Chemistry, 2020, 8: 616326.DOI:10.3389/fchem.2020.616326.
[19] 鹿萌, 张若楠, 刘慧芳, 等.分子印迹技术在新烟碱类农药残留分析中的应用研究进展[J].食品工业科技, 2019, 40(21): 307-314.DOI:10.13386/j.issn1002-0306.2019.21.050.
[20] MAHMOUDPOUR M, TORBATI M, MOUSAVI M M, et al.Nanomaterial-based molecularly imprinted polymers for pesticides detection: recent trends and future prospects[J].TrAC Trends in Analytical Chemistry, 2020, 129: 115943.DOI:10.1016/j.trac.2020.115943.
[21] HU Tianliang, CHEN Run, WANG Qiang, et al.Recent advances and applications of molecularly imprinted polymers in solid-phase extraction for real sample analysis[J].Journal of Separation Science,2021, 44(1): 274-309.DOI:10.1016/j.trac.2020.115943.
[22] IDIL N, HEDSTROM M, DENIZLI A, et al.Whole cell based microcontact imprinted capacitive biosensor for the detection of Escherichia coli[J].Biosensors and Bioelectronics, 2017, 87: 807-815.
[23] WU Jikui, WANG Ruinan, LU Yunfei, et al.Facile preparation of a bacteria imprinted artificial receptor for highly selective bacterial recognition and label-free impedimetric detection[J].Analytical Chemistry, 2019, 91(1): 1027-1033.DOI:10.1021/acs.analchem.8b04314.
[24] FU Kaiyue, ZHANG Huiwen, GUO Yuanyuan, et al.Rapid and selective recognition of Vibrio parahaemolyticus assisted by perfluo-rinated alkoxysilane modified molecularly imprinted polymer film[J].RAC Advance, 2020, 24(10): 14305-14312.DOI:10.1039/D0RA00306A.
[25] HOWITT S H, BLACKSHAW D, FONTAINE E, et al.Comparison of traditional microbiological culture and 16S polymerase chain reaction analyses for identification of preoperative airway colonization for patients undergoing lung resection[J].Journal of Critical Care, 2018,46(8): 84-87.DOI:10.1016/j.jcrc.2018.04.013.
[26] 熊苏玥, 米瑞芳, 陈曦, 等.多重PCR同时检测食品中4 种细菌与常见霉菌[J].食品科学, 2019, 40(4): 305-311.DOI:10.7506/spkx1002-6630-20180521-283.
[27] 覃璐琪, 梁晓琳, 潘海博, 等.采用实时荧光定量PCR法同时定性定量检测发酵乳中多种益生菌[J].食品与发酵工业, 2020, 46(15):238-244.DOI:10.13995/j.cnki.11-1802/ts.023849.
[28] 王春梅.实时荧光定量技术在食品微生物检测和研究中的应用[J].食品科技, 2022(5): 126-128.DOI:10.16043/j.cnki.cfs.2022.13.024.
[29] 王菲, 张玲, 杨佳怡.基于实时荧光定量PCR检测的沙门氏菌标准物质验证[J].食品与发酵工业, 2020, 46(8): 219-225.DOI:10.13995/j.cnki.11-1802/ts.022950.
[30] 范维, 高晓月, 李贺楠, 等.3 种致病菌多重real-time PCR检测方法的建立及其在散装即食肉制品中的应用[J].食品科学, 2022, 43(2):332-338.DOI:10.7506/spkx1002-6630-20210416-230.
[31] 牛犇, 洪斌, 穆丽丽.荧光定量PCR在预测微生物学中的应用[J].食品工业科技, 2019, 40(5): 275-282.DOI:10.13386/j.issn1002-0306.2019.05.046.
[32] LI M Y, ZHOU G H, XU X L, et al.Changes of bacterial diversity and main flora in chilled pork during storage using PCR-DGGE[J].Food Microbiology, 2006, 23(7): 607-611.DOI:10.1016/j.fm.2006.01.004.
[33] 李凤珠, 张玉礼, 杨吉霞.运用实时荧光定量PCR法研究榨菜腌制过程中细菌和真菌数量变化[J].食品与发酵工业, 2019, 45(18): 58-64.DOI:10.13995/j.cnki.11-1802/ts.020771.
[34] 杨曼琼.荧光实时定量PCR检测铜绿假单胞菌oprI基因方法学的建立及运用[D].长沙: 中南大学, 2007: 6-37.
[35] 曹科峰.环介导等温扩增技术检测铜绿假单胞菌方法的建立及其应用[D].合肥: 安徽医科大学, 2017: 16-43.
[36] 郭建平, 万佳佳, 陆兆新, 等.基于环介导等温扩增技术的金黄色葡萄球菌快速检测研究[J].食品科学, 2019, 40(20): 325-331.DOI:10.7506/spkx1002-6630-20180830-332.
[37] LEDLOD S, BUNRODDITH K, AREEKIT S, et al.Development of a duplex lateral flflow dipstick test for the detection and differentiation of Listeria spp.and Listeria monocytogenes in meat products based on loopmediated isothermal amplifification[J].Journal of Chromatography B,2020, 1139: 121834.DOI:10.1016/j.jchromb.2019.121834.
[38] CHEN Xingxing, WU Xiaoli, GAN Min, et al.Rapid detection of Staphylococcus aureus in dairy and meat foods by combination of capture with silica-coated magnetic nanoparticles and thermophilic helicase-dependent isothermal amplification[J].Journal of Dairy Science, 2015, 98(3): 1563-1570.DOI:10.3168/jds.2014-8828.
[39] 张晋豪, 王浩东, 刘欣悦, 等.基于LAMP技术快速检测冷鲜鸡新鲜度[J/OL].食品科学, 2022.https://kns.cnki.net/kcms/detail/11.2206.TS.20220613.1054.063.html.
[40] 何景, 程楠, 许文涛.食品微生物新型快速筛查技术研究进展[J].食品科学, 2015, 36(13): 288-292.DOI:10.7506/spkx1002-6630-201513053.
[41] 冯涛.动物源性成分交叉引物等温扩增快速检测方法的研究和建立[D].杭州: 中国计量大学, 2016: 3-27.
[42] 向勇.种禽场铜绿假单胞菌的流行特点及其CPA检测方法的建立[D].广州: 华南农业大学, 2019: 43-67.
[43] DALMASSO A, DEL RIO M D L D S, CIVERA T, et al.Characterization of microbiota in Plaisentif cheese by high-throughput sequencing[J].LWT-Food Science and Technology, 2016, 69(2):490-496.DOI:10.1016/j.lwt.2016.02.004.
[44] CAPORASO J G, LAUBER C L, WALTERS W A, et al.Ultra-highthroughput microbial community analysis on the Illumina HiSeq and MiSeq platforms[J].The ISME Journal, 2012, 6(8): 1621-1624.DOI:10.1038/ismej.2012.8.
[45] LABRIE S J, EL HADDAD L, TREMBLAY D M, et al.First complete genome sequence of Staphylococcus xylosus, a meat starter culture and a host to propagate Staphylococcus aureus phages[J].Genome Announcements, 2014, 2(4): e00671-14.DOI:10.1128/genomeA.00671-14.
[46] FIORE-DONNO A M, RIXEN C, RIPPIN M, et al.New barcoded primers for efficient retrieval of cercozoan sequences in highthroughput environmental diversity surveys, with emphasis on worldwide biological soil crusts[J].Molecular Ecology Resources,2018, 18(2): 229-239.DOI:10.1111/1755-0998.12729.
[47] DIAS M F, REIS M P, ACURCIO L B, et al.Changes in mouse gut bacterial community in response to different types of drinking water[J].Water Research, 2018, 132: 79-89.DOI:10.1016/j.watres.2017.12.052.
[48] SUSKIND D L, COHEN S A, BRITTNACHER M J, et al.Clinical and fecal microbial changes with diet therapy in active inflammatory bowel disease[J].Journal of Clinical Gastroenterology, 2018, 52(2):155-163.DOI:10.1097/MCG.0000000000000772.
[49] XI La, KONG Xiangcong, YANG Shaoyong, et al.Fungal diversity analysis of wheat qu in yellow wine based on Illunima Miseq sequencing technology[J].Food Research and Development, 2022,43(12): 185-190.DOI:10.12161/j.issn.1005-6521.2022.12.024.
[50] DONG Yun, WANG Yurong, WANG Yao, et al.Evaluation of bacterial diversity in Chinese bacon from Enshi by denatured gradient gel electrophoresis and Miseq high-throughput sequencing[J].Meat Research,2018, 32(10): 37-42.DOI:10.7506/rlyj1001-8123-201810007.
[51] 田建军, 张开屏, 杨明阳, 等.应用Illumina MiSeq测序技术比较风干肉中细菌多样性和微生物安全性[J].食品科学, 2019, 40(8): 33-40.DOI:10.7506/spkx1002-6630-20180504-043.
[52] 庄齐斌, 郑晓春, 杨德勇, 等.基于高光谱反射特性的猪肉新鲜度和腐败程度的对比分析[J].食品科学, 2021, 42(16): 254-260.DOI:10.7506/spkx1002-6630-20200310-156.
[53] 马无锡.基于高光谱成像技术的鱼肉新鲜度无损检测方法研究[J].信息系统工程, 2020(9): 125-126.
[54] 赵婕秀, 董庆利, 陈培琴, 等.高光谱成像技术在动物源性食品微生物污染快速检测中的应用进展[J].食品工业科技, 2022, 43(7):467-473.DOI:10.13386/j.issn1002-0306.2021050236.
[55] 王慧, 何鸿举, 张海曼, 等.高光谱成像技术快速预测冷鲜鸡胸肉的嫩度[J].海南师范大学学报(自然科学版), 2018, 31(2): 164-170.
[56] 何鸿举, 蒋圣启, 王魏, 等.高光谱快速预测冷鲜鸡胸肉中乳酸菌[J].食品工业科技, 2020, 41(12): 227-233.DOI:10.13386/j.issn1002-0306.2020.12.037.
[57] FENG Yaoze, ELMASRY G, SUN Dawen, et al.Near-infrared hyperspectral imaging and partial least squares regression for rapid and reagentless determination of Enterobacteriaceae on chicken fillets[J].Food Chemistry, 2013, 138(2/3): 1829-1836.DOI:10.1016/j.foodchem.2012.11.040.
[58] 何鸿举, 蒋圣启, 马汉军, 等.基于NIR高光谱技术快速预测冷鲜鸡肉热杀索丝菌含量[J].食品工业科技, 2020, 41(13): 241-252.DOI:10.13386/j.issn1002-0306.2020.13.038.
[59] 何鸿举, 朱亚东, 王慧, 等.近红外光谱技术在生鲜禽肉质量检测中应用的研究进展[J].食品科学, 2019, 40(21): 317-323.DOI:10.7506/spkx1002-6630-20181119-218.
[60] 刘彦俊, 王力.偏最小二乘回归模型在EEG特征选择的应用[J].计算机工程与应用, 2021(19): 218-223.
[61] 莫嘉琪.近红外光谱技术在食品微生物检测中的应用[J].现代食品, 2021, 13(39): 140-142.DOI:10.16736/j.cnki.cn41-1434/ts.2021.13.039.
[62] ALEXANDRAKIS D, DOWNEYG, SCANNELL A G M, et al.Detection and identifification of selected bacteria, inoculated on chicken breast, using near infrared spectroscopy and chemometrics[J].Sensing and Instrumentation for Food Quality and Safety, 2011, 5(2):57-62.DOI:10.1007/s11694-011-9111-y.
[63] 张雷蕾.冷却肉微生物污染及食用安全的光学无损评定研究[D].北京: 中国农业大学, 2015: 19-34.
[64] 王名星.基于嗅觉可视化和近红外光谱技术的鸡肉中假单胞菌快速识别研究[D].镇江: 江苏大学, 2017: 27-44.
[65] 陶站华, 刘军贤, 师德强, 等.基于单细胞拉曼光谱技术的葡萄球菌黄素生物合成分析[J].分析化学, 2016, 44(3): 456-461.DOI:10.11895/j.issn.0253-3820.150729.
[66] 李欢欢.牛奶中主要有害污染物的表面增强拉曼光谱检测方法研究[D].镇江: 江苏大学, 2018: 13-46.
[67] MA Xiaoyuan, XU Xumin, XIA Yu, et al.SERS aptasensor for Salmonella typhimurium detection based on spiny gold nanoparticles[J].Food Control, 2018, 84: 232-237.DOI:10.1016/j.foodcont.2017.07.016.
[68] ZHANG Hui, MA Xiaoyuan, LIU Ying, et al.Gold nanoparticles enhanced SERS aptasensor for the simultaneous detection of Salmonella typhimurium and Staphylococcus aureus[J].Biosensors and Bioelectronics, 2015, 74: 872-877.DOI:10.1016/j.bios.2015.07.033.
[69] ARGYRI A A, JARVIS R M, WEDGE D, et al.A comparison of Raman and FT-IR spectroscopy for the prediction of meat spoilage[J].Food Control, 2013, 29(2): 461-470.DOI:10.1016/j.foodcont.2012.05.040.
[70] 杨鸿博.拉曼光谱预测牛肉中微生物的初步探究[D].泰安: 山东农业大学, 2020: 6-42.
[71] 范孝英.酶联免疫吸附(ELISA)法在食品微生物检测中的应用分析[J].食品科技, 2021(3): 152-154.DOI:10.16043/j.cnki.cfs.2021.09.083.
[72] 高毅云, 刘传桂.酶联免疫吸附测定法与免疫胶体金技术在沙眼衣原体检测中的应用比较[J].中国医学创新, 2015, (17): 128-130.
[73] 谢珲, 章先.黄曲霉毒素B1单克降抗体的制备及间接竞争ELISA检测技术研究[J].微生物学通报, 2015(10): 2033-2040.
[74] 姚永忠, 刘明远.BA-Dot-EIdSA快速检测食品中单核白细胞增多症李氏杆菌[J].山西农业大学学报, 1993(12): 3-5.
[75] 孔玉方, 王慧煜, 韩雪清.胶体金免疫层析技术在食源性致病微生物检测中的应用[J].中国兽医杂志, 2019, 55(4): 82-84.
[76] 朱芳茜, 何扩, 张秀媛, 等.量子点标记荧光免疫试纸条可视化检测肉品中志贺氏菌研究[J].食品研究与开发, 2021, 42(3): 136-140.DOI:10.12161/j.issn.1005-6521.2021.03.022.
[77] SONG Chunmei, LIU Cheng, WU Shuyan, et al.Development of alateral flow colloidal gold immunoassay strip for the simultaneous detection of Shigella boydii, and Escherichia coli O157:H7 in bread, milk and jelly samples[J].Food Control, 2016, 59: 345-351.DOI:10.1016/j.foodcont.2015.06.012.