肉品富含蛋白质、维生素和许多其他对健康有益的营养物质,在人类饮食中扮演着重要角色。随着社会经济的发展,人们对肉品的需求量不断增加。全球肉品消费量已从1961年的7 000 万t增长到2021年的3.28 亿t[1-2],到2050年预计将超过4.64 亿t[3]。另一方面,肉品营养丰富的特性也使其成为微生物的天然培养基,在加工、贮藏、运输及销售过程中极易受到致病菌、腐败菌等有害微生物的污染,进而对消费者的健康产生威胁。因此,针对肉品中的有害微生物进行精准、有效的检测、预测和控制,对整个肉品行业至关重要。
过去的20年里,食品行业得到了巨大的发展,信息化、自动化程度不断提升,食品安全监管体系逐步完善,这也使得包括肉品在内的整个食品行业产生了规模空前的数据流[4]。机器学习方法可以从海量、复杂的数据中提取关键信息,学习数据集中各变量间的隐藏关系,现已逐渐成为食品安全领域数据密集型分析任务的强有力工具。
本文通过归纳近年来国内外相关研究,总结了机器学习方法在肉品中有害微生物的检测和预测建模中所起到的关键作用,分析了该法在实际应用时的不足之处,并展望了该法在肉类微生物安全研究中的应用前景。
1 机器学习方法简介
机器学习这一概念最初由Arthur Samuel于20世纪50年代提出:“机器学习是一个研究领域,让计算机无需进行明确编程就具备学习能力”。1997年,Tom Mitchell又为其提出了一个更加工程化的概念:“一个计算机程序利用经验E来学习任务T,性能是P,如果针对任务T的性能P随着经验E不断增长,则任务T被称之为机器学习”。现如今,大多数被广泛使用的机器学习算法或模型都是在20世纪60—90年代提出的,如K-近邻、随机森林、神经网络等。随着大数据时代的到来,得益于数据量和计算机计算能力的指数级增长,机器学习在各个领域的应用都得到了史无前例的发展。
作为人工智能领域的一个重要分支,不同于传统算法,机器学习在解决实际问题时不再试图编写详细且明确的规则或计算机指令。相反,机器学习系统从具体的实例中训练模型,并不断优化模型参数,以提高对新实例的预测准确性。机器学习的数据驱动性和规则未知性使其特别适用于以下几类问题[5]:1)有解决方案但解决方案需要进行大量人工微调或需要遵循大量规则的问题;2)通过传统的数学方法无法得出显式解的复杂问题;3)不断有新实例或新数据产生而使旧规则失效的问题;4)拥有大量数据且需要从中获取更多规律的问题。
在训练过程中,机器学习系统可能会受到来自人类的指导或监督。通常,根据是否受到监督,机器学习中的问题大致可以分为有监督学习、无监督学习和强化学习[5]。有监督学习要求所有提供给算法的输入必须具有相应的输出,即人为地为输入数据建立期望的或基于事实的标签。常见的分类和回归问题就是2 类典型的有监督学习任务。与有监督学习不同,无监督学习任务中的训练数据都是不具有标签的,并要求算法自主发现输入数据中的规律。聚类分析、主成分分析(principal components analysis,PCA)和异常值检测是常见的无监督学习任务。与前2 类机器学习任务不同,强化学习是一种非常复杂的学习系统,它可以在给定情况下找到完成任务目标的最优方案。强化学习基于“回报-惩罚”机制实现学习过程,即算法目标随着时间的推移实现回报最大化。
为了适应具有不同复杂程度的问题,人们提出了许多基于不同理论的机器学习算法。图1总结了4 种代表性的基础机器学习算法[6]。K-means是一种常用的基于欧氏距离的聚类算法,属于无监督学习[7],其可以动态地将相似的观察结果划分成簇,并根据目标间的欧氏距离判断相似度(图1A)。支持向量机(support vector machine,SVM)是一种功能强大且全面的机器学习算法[8],它能够执行线性或非线性分类、回归,甚至是异常值检测任务。SVM算法使用实线或平面将不同类别的观察数据分成不同的类,特别适用于中小型复杂数据集的分类(图1B)。决策树(decision tree,DT)算法[9]可以根据输入数据的不同特征将实例递归地划分为不同的类,以生成类似流程图的树结构,DT算法中根节点和叶节点表示对某一属性的1 次测试(图1C)。人工神经网络(artificial neural network,ANN)通常由人工神经元多层相互连接组成,以模拟真实的生物神经元在动物大脑中的协同工作[10]。这些人工神经元接收输入信号,并通过权重和输入的线性组合以及一个非线性激活函数将输出信号传入到下一层中的人工神经元(图1D)。ANN算法一般由1 个输入层、1 个或多个隐含层和1 个输出层组成,而一个包含许多隐含层的神经网络被称作深度神经网络,即深度学习的核心[10]。
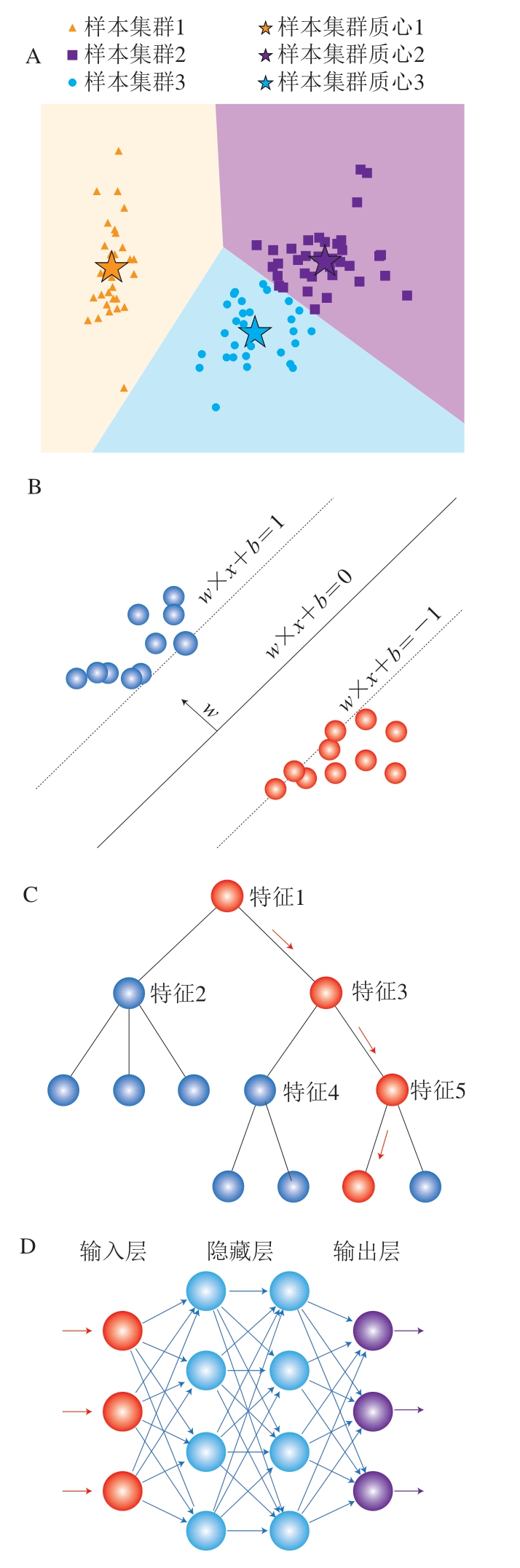
图1 4 种代表性的机器学习算法示意图[6]Fig.1 Schematic diagrams of four machine learning models[6]
A.K-means算法;B.SVM算法(x表示输入数据,w和b表示SVM算法超平面参数);C.DT算法;D.ANN算法。
2 机器学习在肉品有害微生物检测中的应用
传统的微生物检测方法,如平板计数法、聚合酶链式反应法、免疫分析法等,通常耗时、有损且需要专业操作人员,因此传统检测方法并不能很好地满足现代肉品行业的需求[11-12]。随着计算机科学和光学技术的发展,光谱技术、计算机视觉、生物传感器等新型检测技术逐步进入研究人员的视野,其中光谱技术因其快速、无损和高效的优点,在微生物检测领域脱颖而出[11,13-14]。然而,光谱技术在检测过程中往往会产生大量复杂且难以解释的数据,如何剔除检测数据中的冗余信息并提取关键特征是光谱技术在实际应用时的难点。如前所述,机器学习方法特别适合于大量复杂数据的分析工作,且该法已在国内外许多研究中与光谱技术一起应用于肉品中有害微生物的快速检测。Argyri等[15]比较傅里叶变换红外光谱和拉曼光谱在预测牛肉中微生物数量时的性能差异。在数据分析时,该研究使用了多种不同的机器学习方法,包括偏最小二乘回归(partial least square regression,PLSR)、遗传算法(genetic algorithm,GA)、ANN算法、支持向量回归(support vector regression,SVR)等。结果表明,PLSR和SVR模型的预测效果普遍优于其他模型。Duan Cui等[16]使用便携式近红外光谱对比目鱼鱼片中的总细菌数进行无损快速检测,将GA和ANN算法应用于600~1 100 nm的红外光谱数据分析,并建立了相应的预测模型。结果表明,2 种机器学习算法的均方根误差(root mean square error,RMSE)均小于其他模型。董小栋等[17]利用高光谱成像技术和SVR模型对香肠中菌落总数进行定量预测和数据可视化,SVR模型的相关系数高达0.977 7。除了对微生物数量进行定量分析外,利用光谱技术结合无监督机器学习算法还可以实现对肉品中有害微生物的定性区分。孙颖颖[18]使用基于纳米银颗粒的表面增强拉曼光谱结合PCA和层次聚类分析(hierarchical clustering analysis,HCA),实现了对牛肉样品中的鼠伤寒沙门氏菌、单核细胞增生李斯特菌(以下简称单增李斯特菌)、金黄色葡萄球菌、大肠杆菌O157:H7的分类,对于不同致病菌的分类准确率达到93%。类似的结果也见于Xie Yunfei[19]、Witkowska[20]等的相关研究中。表1总结了部分光谱技术和机器学习方法在肉品有害微生物检测中的应用研究。
表1 光谱技术和机器学习方法在肉品有害微生物检测中的应用实例
Table 1 Application of spectroscopy combined with machine learning in detection of harmful microorganism in meat
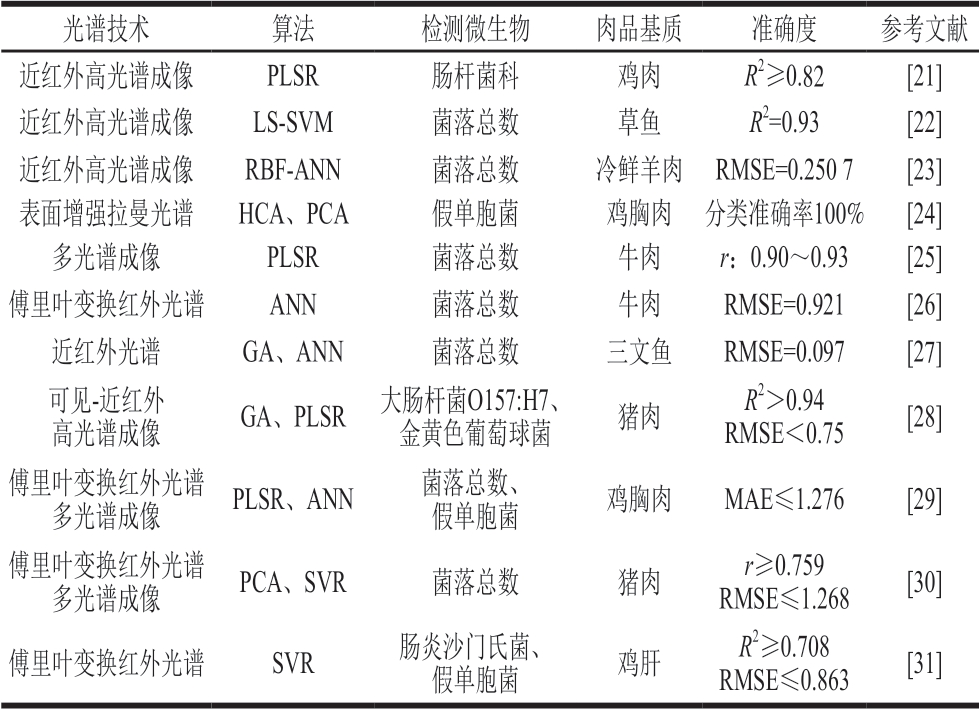
注:LS-SVM.最小二乘SVM(least square-SVM);RBF-ANN.径向基底ANN(radial basis function-ANN);r.相关系数;R2.决定系数;MAE.平均绝对误差(mean absolute error)。
光谱技术算法检测微生物肉品基质准确度参考文献近红外高光谱成像PLSR肠杆菌科鸡肉R2≥0.82[21]近红外高光谱成像LS-SVM菌落总数草鱼R2=0.93[22]近红外高光谱成像RBF-ANN菌落总数冷鲜羊肉RMSE=0.250 7[23]表面增强拉曼光谱HCA、PCA假单胞菌鸡胸肉分类准确率100%[24]多光谱成像PLSR菌落总数牛肉r:0.90~0.93[25]傅里叶变换红外光谱ANN菌落总数牛肉RMSE=0.921[26]近红外光谱GA、ANN菌落总数三文鱼RMSE=0.097[27]可见-近红外高光谱成像GA、PLSR大肠杆菌O157:H7、金黄色葡萄球菌猪肉R2>0.94 RMSE<0.75[28]傅里叶变换红外光谱多光谱成像PLSR、ANN菌落总数、假单胞菌鸡胸肉MAE≤1.276[29]傅里叶变换红外光谱多光谱成像PCA、SVR菌落总数猪肉r≥0.759 RMSE≤1.268[30]傅里叶变换红外光谱SVR肠炎沙门氏菌、假单胞菌鸡肝R2≥0.708 RMSE≤0.863[31]
光谱技术和机器学习方法的结合可以定性或定量检测不同肉品基质中的有害微生物,在国内外的研究中也日趋成熟。然而,现阶段与光谱检测技术相关联的机器学习算法大多较为基础,在分析涉及多种肉品基质或微生物类别的大型数据集时,难免会占用大量的计算资源,分析效率低下,并严重影响检测准确性[32]。因此,开发更加复杂且先进的机器学习算法以匹配日益增大的数据量,是拓展光谱检测技术实际应用前景的重点和难点。
3 机器学习在肉品有害微生物预测建模中的应用
食源性致病菌及其引起的食源性疾病是全球食品安全面临的重要挑战之一[33]。根据Li Weiwei等[34]的研究,2003—2017年间我国因肉品暴发的食源性疾病中有近半数是由食源性致病菌污染所引起。此外,根据国家食品安全风险评估中心和疾病预防控制中心2020—2021年的相关监测数据,肉品中的沙门氏菌、大肠杆菌、副溶血性弧菌等依然是我国亟需控制的重要食源性致病因子[35-36]。针对此类食品安全问题,在肉品工业中应采取更有效的措施严格控制肉品中致病菌的污染水平,并结合预测微生物学建模估算其在不同加工及流通条件下的生长/失活特性,以降低因食源性致病菌所引发的食源性疾病暴发风险。
预测微生物学是一门在微生物学、数学、统计学和应用计算机科学基础上建立起来的学科,它的目的是研究和设计一系列能描述和预测食品微生物在特定条件下生长和衰亡的模型[37]。根据不同的分类标准可将预测微生物学模型分为不同的类别。其中,由Whiting和Buchanan划分的一级、二级和三级模型在预测微生物学中应用较为广泛[38]。一级模型主要描述微生物量(如微生物细胞的数目、浊度、形成的毒素浓度等响应值)与时间之间的函数关系。二级模型亦称次级模型,通过建立一级模型中的各项参数与环境条件之间的函数关系,其可以很好地表示环境条件对微生物行为的影响。三级模型主要指建立在一级生长/失活模型和二级生长/失活模型之上的电脑应用软件程序。
传统的预测微生物学模型在描述食源性致病菌的生长失活行为及环境条件与致病菌特定行为的定量关系时,通常使用经验式的数学回归方法[39]。通过传统模型得到的预测结果大多是确定性的点估计[40-41],然而由于变异性的存在,实际的致病菌行为通常是某个特定的范围,而不是单一的确定值。在食品供应链中,影响微生物行为的变异性来源主要包括加工、贮藏等环节中环境条件的变异性、菌株变异性、单细胞异质性等[42]。由于忽视了微生物行为的变异性,传统的确定性模型已被证明并不适用于描述所有场景下微生物的行为特性[43-45]。近年来,更加注重微生物行为变异性的随机模型得到研究人员的关注,这类模型通过概率分布的方式更好地体现了变异性对微生物行为的影响[46-47]。然而,由于通常难以将模型的预测结果与实际的观测结果进行对比且依赖大量的理论化假设,关于随机模型的可用性与准确性依然存在争议。不同于确定性模型和随机模型,机器学习方法可以在不作出过多假设和相关机制未知的情况下,准确捕捉输入和输出之间的复杂规则。因此,机器学习方法在预测微生物学领域中也已经得到越来越多的应用。范志文等[48]使用反向传播神经网络(back propagation ANN,BP-ANN)构建了酱牛肉中金黄色葡萄球菌在不同温度和不同初始菌量下的生长模型。结果表明,经过超参数优化之后的BP-ANN相较于传统的Gompertz模型表现出更低的预测误差。Oscar等[49-52]使用前馈神经网络、广义回归神经网络建立了鸡肉中多种不同血清型沙门氏菌的生长模型,均取得了较好的预测效果。除了生长建模,机器学习算法还可应用于食源性致病菌的失活建模研究中。相较于三阶多项式回归模型,ANN算法更精确地描述了香肠中单增李斯特菌在不同环境条件下的失活行为[53]。Gosukonda等[54]在传统BP-ANN的基础上,引入卡尔曼滤波算法,并成功描述了牛肉表面低压电流对大肠杆菌O157:H7失活行为的影响,该模型成功体现出微生物在失活过程中的变异性和不确定性。表2总结了机器学习方法在肉品有害微生物预测建模中的部分应用。
表2 机器学习方法在肉品中有害微生物预测建模中的应用实例
Table 2 Selected examples of the application of machine learning in predictive modeling of harmful microorganism in meat
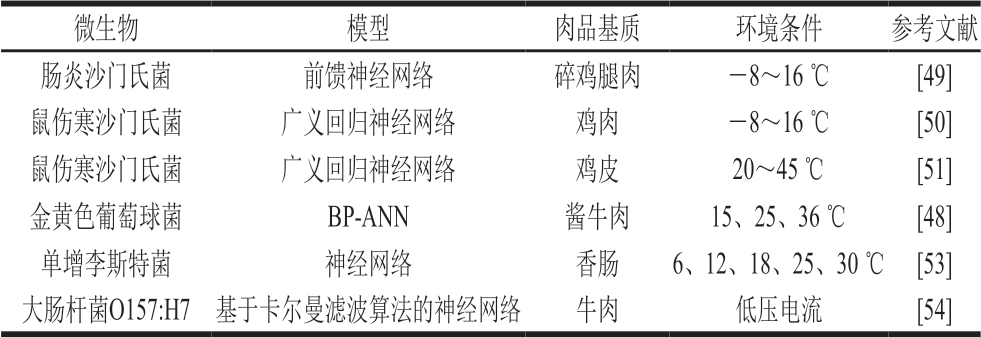
微生物模型肉品基质环境条件参考文献肠炎沙门氏菌前馈神经网络碎鸡腿肉-8~16 ℃[49]鼠伤寒沙门氏菌广义回归神经网络鸡肉-8~16 ℃[50]鼠伤寒沙门氏菌广义回归神经网络鸡皮20~45 ℃[51]金黄色葡萄球菌BP-ANN酱牛肉15、25、36 ℃[48]单增李斯特菌神经网络香肠6、12、18、25、30 ℃[53]大肠杆菌O157:H7基于卡尔曼滤波算法的神经网络牛肉低压电流[54]
机器学习算法因其较强的灵活性和较高的预测精度而在预测微生物学相关领域得到广泛应用。现阶段,限制机器学习模型性能的主要因素是数据集的大小和质量。如果数据集体积过小且具有大量的缺失值,机器学习模型便很容易产生过拟合现象,从而导致预测准确度的下降。因此,在未来的研究中应结合大型微生物行为数据库(如Combase数据库),对机器学习算法进行预训练,从而提升其在包括肉品在内的多种食品基质中的适用性。
此外,由于机器学习模型的“黑盒”属性,研究人员往往只能得到单一的预测结果,无法得知模型在预测过程中如何做出决策以及数据集中每个输入特征如何对最终预测产生影响。因此,在未来的研究中,应利用特征重要性分析等方法,提升机器学习方法在预测微生物行为时的可解释性。
4 结 语
随着大数据时代的到来,机器学习已在各个领域内展现出其独有的魅力。利用机器学习方法从海量数据中提取关键信息,对肉品有害微生物进行精准识别与预测,是实现肉类微生物安全的重要途径。本文综述了机器学习方法在肉品有害微生物检测和预测建模方向的研究进展,分析了该法的优势和不足,以期为今后该领域内的研究提供一定参考。
以下,针对机器学习方法在肉类微生物安全领域内应用时所体现出的不足之处提出展望:1)针对光谱检测技术中可能出现的高维复杂数据集,可结合深度学习方法和迁移学习的思想,提高重要特征的识别效率,并加快模型的训练速度,从而使得光谱检测技术可以更好地应用于肉品有害微生物的检测;2)应加快大型微生物行为数据库的构建和完善,充分发挥机器学习的优势,建立适用范围更广、预测精度更高的肉品微生物预测模型;3)推进机器学习在肉品有害微生物控制中的应用,通过机器学习方法,优化现阶段肉品杀菌工艺中的相关参数,从“检测”“预测”“控制”3 个角度实现肉类微生物安全。
[1] TAO Feifei, PENG Yankun.A method for nondestructive prediction of pork meat quality and safety attributes by hyperspectral imaging technique[J].Journal of Food Engineering, 2014, 126: 98-106.DOI:10.1016/j.jfoodeng.2013.11.006.
[2] OECD/FAO.OECD-FAO agricultural outlook 2022—2031[EB/OL].(2022-06-29) [2022-10-29].https://www.oecd-ilibrary.org/agricultureand-food/oecd-fao-agricultural-outlook-2022-2031_f1b0b29c-en.
[3] REVELL B.Meat and milk consumption 2050: the potential for demand-side solutions to greenhouse gas emissions reduction[J].EuroChoices, 2015, 14(3): 4-11.DOI:10.1111/1746-692X.12103.
[4] MARVIN H J P, JANSSEN E M, BOUZEMBRAK Y, et al.Big data in food safety: an overview[J].Critical Reviews in Food Science and Nutrition, 2017, 57(11): 2286-2295.DOI:10.1080/10408398.2016.1257481.
[5] JORDAN M I, MITCHELL T M.Machine learning: trends,perspectives, and prospects[J].Science, 2015, 349(6245): 255-260.DOI:10.1126/science.aaa8415.
[6] DENG Xiangyu, CAO Shuhao, HORN A L.Emerging applications of machine learning in food safety[J].Annual Review of Food Science and Technology, 2021, 12(1): 513-538.DOI:10.1146/annurevfood-071720-024112.
[7] HARTIGAN J A, WONG M A.A K-means clustering algorithm[J].Applied Statistics, 1979, 28(1): 100-108.DOI:10.2307/2346830.
[8] CORTES C, VAPNIK V.Support-vector networks[J].Machine Learning, 1995, 20(3): 273-297.DOI:10.1007/BF00994018.
[9] QUINLAN J R.Induction of decision trees[J].Machine Learning,1986, 1(1): 81-106.DOI:10.1023/A:1022643204877.
[10] SCHMIDHUBER J.Deep learning in neural networks: an overview[J].Neural Networks, 2015, 61: 85-117.DOI:10.1016/j.neunet.2014.09.003.
[11] ELLIS D I, GOODACRE R.Rapid and quantitative detection of the microbial spoilage of muscle foods: current status and future trends[J].Trends in Food Science and Technology, 2001, 12(11): 414-424.DOI:10.1016/S0924-2244(02)00019-5.
[12] KADEMI H I, ULUSOY B H, HECER C.Applications of miniaturized and portable near infrared spectroscopy (NIRS) for inspection and control of meat and meat products[J].Food Reviews International,2019, 35(3): 201-220.DOI:10.1080/87559129.2018.1514624.
[13] WU Di, SUN Dawen.Advanced applications of hyperspectral imaging technology for food quality and safety analysis and assessment: a review-Part II: applications[J].Innovative Food Science and Emerging Technologies, 2013, 19: 15-28.DOI:10.1016/j.ifset.2013.04.016.
[14] WANG Kaiqiang, PU Hongbin, SUN Dawen.Emerging spectroscopic and spectral imaging techniques for the rapid detection of microorganisms: an overview[J].Comprehensive Reviews in Food Science and Food Safety, 2018, 17(2): 256-273.DOI:10.1111/1541-4337.12323.
[15] ARGYRI A A, JARVIS R M, WEDGE D, et al.A comparison of Raman and FT-IR spectroscopy for the prediction of meat spoilage[J].Food Control, 2013, 29(2): 461-470.DOI:10.1016/j.foodcont.2012.05.040.
[16] DUAN Cui, CHEN Chunguang, KHAN M N, et al.Non-destructive determination of the total bacteria in flounder fillet by portable near infrared spectrometer[J].Food Control, 2014, 42: 18-22.DOI:10.1016/j.foodcont.2014.01.023.
[17] 董小栋, 郭培源, 徐盼.基于高光谱成像的香肠菌落总数回归预测及数据可视化[J].现代食品科技, 2017, 33(7): 308-314.DOI:10.13982/j.mfst.1673-9078.2017.7.043.
[18] 孙颖颖.表面增强拉曼光谱对肉源性致病菌的快速检测方法研究[D].泰安: 山东农业大学, 2022: 53.DOI:10.27277/d.cnki.gsdnu.2022.000990.
[19] XIE Yunfei, XU Li, WANG Yiqian, et al.Label-free detection of the foodborne pathogens of Enterobacteriaceae by surface-enhanced Raman spectroscopy[J].Analytical Methods, 2013, 5(4): 946-952.DOI:10.1039/c2ay26107c.
[20] WITKOWSKA E, KORSAK D, KOWALSKA A, et al.Surfaceenhanced Raman spectroscopy introduced into the International Standard Organization (ISO) regulations as an alternative method for detection and identification of pathogens in the food industry[J].Analytical and Bioanalytical Chemistry, 2017, 409(6): 1555-1567.DOI:10.1007/s00216-016-0090-z.
[21] FENG Yaoze, ELMASRY G, SUN Dawen, et al.Near-infrared hyperspectral imaging and partial least squares regression for rapid and reagentless determination of Enterobacteriaceae on chicken fillets[J].Food Chemistry, 2013, 138(2/3): 1829-1836.DOI:10.1016/j.foodchem.2012.11.040.
[22] CHENG Junhu, SUN Dawen.Rapid and non-invasive detection of fish microbial spoilage by visible and near infrared hyperspectral imaging and multivariate analysis[J].LWT-Food Science and Technology,2015, 62(2): 1060-1068.DOI:10.1016/j.lwt.2015.01.021.
[23] 郭中华, 郑彩英, 金灵.基于近红外高光谱成像的冷鲜羊肉表面细菌总数检测[J].食品工业科技, 2014, 35(20): 66-81.DOI:10.13386/j.issn1002-0306.2014.20.005.
[24] LIU Shuangshuang, LI Huanhuan, HASSAN M M, et al.Amplification of Raman spectra by gold nanorods combined with chemometrics for rapid classification of four Pseudomonas[J].International Journal of Food Microbiology, 2019, 304: 58-67.DOI:10.1016/j.ijfoodmicro.2019.05.020.
[25] PANAGOU E Z, PAPADOPOULOU O, CARSTENSEN J M, et al.Potential of multispectral imaging technology for rapid and nondestructive determination of the microbiological quality of beef filets during aerobic storage[J].International Journal of Food Microbiology,2014, 174: 1-11.DOI:10.1016/j.ijfoodmicro.2013.12.026.
[26] PANAGOU E Z, MOHAREB F R, ARGYRI A A, et al.A comparison of artificial neural networks and partial least squares modelling for the rapid detection of the microbial spoilage of beef fillets based on Fourier transform infrared spectral fingerprints[J].Food Microbiology,2011, 28(4): 782-790.DOI:10.1016/j.fm.2010.05.014.
[27] 段翠, 陈春光, 刘永志, 等.基于手持式近红外光谱仪的三文鱼菌落总数检测技术[J].食品安全质量检测学报, 2014, 5(3): 889-893.DOI:10.19812/j.cnki.jfsq11-5956/ts.2014.03.044.
[28] BONAH E, HUANG X, AHETO J H, et al.Comparison of variable selection algorithms on vis-NIR hyperspectral imaging spectra for quantitative monitoring and visualization of bacterial foodborne pathogens in fresh pork muscles[J].Infrared Physics and Technology,2020, 107: 103327.DOI:10.1016/j.infrared.2020.103327.
[29] SPYRELLI E D, OZCAN O, MOHAREB F, et al.Spoilage assessment of chicken breast fillets by means of Fourier transform infrared spectroscopy and multispectral image analysis[J].Current Research in Food Science, 2021, 4: 121-131.DOI:10.1016/j.crfs.2021.02.007.
[30] FENGOU L C, MPORAS I, SPYRELLI E, et al.Estimation of the microbiological quality of meat using rapid and non-invasive spectroscopic sensors[J].IEEE Access, 2020, 8: 106614-106628.DOI:10.1109/ACCESS.2020.3000690.
[31] DOUROU D, GROUNTA A, ARGYRI A A, et al.Rapid microbial quality assessment of chicken liver inoculated or not with Salmonella using FTIR spectroscopy and machine learning[J].Frontiers in Microbiology, 2021, 11: 623788.DOI:10.3389/fmicb.2020.623788.
[32] ROPODI A I, PANAGOU E Z, NYCHAS G J E.Data mining derived from food analyses using non-invasive/non-destructive analytical techniques; determination of food authenticity, quality and safety in tandem with computer science disciplines[J].Trends in Food Science and Technology, 2016, 50: 11-25.DOI:10.1016/j.tifs.2016.01.011.
[33] 方太松, 吴瑜凡, 石宁馨, 等.食源性致病菌生长延滞期的研究进展[J].食品科学, 2021, 42(11): 253-260.DOI:10.7506/spkx1002-6630-20200504-024.
[34] LI Weiwei, PIRES S M, LIU Zhitao, et al.Surveillance of foodborne disease outbreaks in China, 2003—2017[J].Food Control, 2020, 118:107359.DOI:10.1016/j.foodcont.2020.107359.
[35] 李红秋, 贾华云, 赵帅, 等.2021年中国大陆食源性疾病暴发监测资料分析[J].中国食品卫生杂志, 2022, 34(4): 816-821.DOI:10.13590/j.cjfh.2022.04.029.
[36] LI Hongqiu, LI Weiwei, DAI Yue, et al.Characteristics of settings and etiologic agents of foodborne disease outbreaks: China, 2020[J].China CDC Weekly, 2021, 3(42): 889-893.DOI:10.46234/ccdcw2021.219.
[37] 董庆利, 王海梅, MALAKAR P K, 等.我国食品微生物定量风险评估的研究进展[J].食品科学, 2015, 36(11): 221-229.DOI:10.7506/spkx1002-6630-201511042.
[38] MCDONALD K, SUN D W.Predictive food microbiology for the meat industry: a review[J].International Journal of Food Microbiology,1999, 52(1/2): 1-27.DOI:10.1016/S0168-1605(99)00126-9.
[39] ROSS T, MCMEEKIN T A.Predictive microbiology[J].International Journal of Food Microbiology, 1994, 23(3/4): 241-264.DOI:10.1016/0168-1605(94)90155-4.
[40] KOUTSOUMANIS K P, ASPRIDOU Z.Individual cell heterogeneity in predictive food microbiology: challenges in predicting a “noisy”world[J].International Journal of Food Microbiology, 2017, 240: 3-10.DOI:10.1016/j.ijfoodmicro.2016.06.021.
[41] POSCHET F, GEERAERD A H, SCHEERLINCK N, et al.Monte carlo analysis as a tool to incorporate variation on experimental data in predictive microbiology[J].Food Microbiology, 2003, 20(3): 285-295.DOI:10.1016/S0740-0020(02)00156-9.
[42] HIURA S, ABE H, KOYAMA K, et al.Transforming kinetic model into a stochastic inactivation model: statistical evaluation of stochastic inactivation of individual cells in a bacterial population[J].Food Microbiology, 2020, 91: 103508.DOI:10.1016/j.fm.2020.103508.
[43] KOYAMA K, HIURA S, ABE H, et al.Application of growth rate from kinetic model to calculate stochastic growth of a bacteria population at low contamination level[J].Journal of Theoretical Biology, 2021,525(4): 110758.DOI:10.1016/j.jtbi.2021.110758.
[44] KOSEKI S, KOYAMA K, ABE H.Recent advances in predictive microbiology: theory and application of conversion from population dynamics to individual cell heterogeneity during inactivation process[J].Current Opinion in Food Science, 2021, 39: 60-67.DOI:10.1016/j.cofs.2020.12.019.
[45] KOUTSOUMANIS K P, LIANOU A, GOUGOULI M.Latest developments in foodborne pathogens modeling[J].Current Opinion in Food Science, 2016, 8: 89-98.DOI:10.1016/j.cofs.2016.04.006.
[46] PIN C, BARANYI J.Kinetics of single cells: observation and modeling of a stochastic process[J].Applied and Environmental Microbiology, 2006, 72(3): 2163-2169.DOI:10.1128/AEM.72.3.2163-2169.2006.
[47] ASPRIDOU Z, KOUTSOUMANIS K P.Individual cell heterogeneity as variability source in population dynamics of microbial inactivation[J].Food Microbiology, 2015, 45: 216-221.DOI:10.1016/j.fm.2014.04.008.
[48] 范志文, 光翠娥, 干建平.基于反向传播人工神经网络的酱牛肉中金黄色葡萄球菌的生长模型[J].食品与生物技术学报, 2020, 39(7):83-90.DOI:10.3969/j.issn.1673-1689.2020.07.012.
[49] OSCAR T P.Neural network model for survival and growth of Salmonella enterica serotype 8,20:-:z(6) in ground chicken thigh meat during cold storage: extrapolation to other serotypes[J].Journal of Food Protection, 2015, 78(10): 1819-1827.DOI:10.4315/0362-028X.JFP-15-093.
[50] OSCAR T P.General regression neural network model for behavior of Salmonella on chicken meat during cold storage[J].Journal of Food Science, 2014, 79(5): M978-M987.DOI:10.1111/1750-3841.12435.
[51] OSCAR T P.Extrapolation of a predictive model for growth of a low inoculum size of Salmonella typhimurium DT104 on chicken skin to higher inoculum sizes[J].Journal of Food Protection, 2011, 74(10):1630-1638.DOI:10.4315/0362-028X.JFP-11-127.
[52] OSCAR T P.General regression neural network and monte carlo simulation model for survival and growth of Salmonella on raw chicken skin as a function of serotype, temperature, and time for use in risk assessment[J].Journal of Food Protection, 2009, 72(10): 2078-2087.DOI:10.4315/0362-028X-72.10.2078.
[53] HAJMEER M, BASHEER I, CLIVER D O.Survival curves of Listeria monocytogenes in chorizos modeled with artificial neural networks[J].Food Microbiology, 2006, 23(6): 561-570.DOI:10.1016/j.fm.2005.09.011.
[54] GOSUKONDA R, MAHAPATRA A K, LIU X, et al.Application of artificial neural network to predict Escherichia coli O157:H7 inactivation on beef surfaces[J].Food Control, 2015, 47: 606-614.DOI:10.1016/j.foodcont.2014.08.002.