肉品腐败变质给人类食品供应带来极大危害。生鲜肉营养丰富、含水量高、极易腐败。以往研究认为肉类微生物的污染主要来自胴体污染,但近年研究发现,最主要来源是接触表面污染[1]。生猪屠宰、分割、运输等阶段均可能通过直接接触使畜禽胴体和分割肉污染大肠杆菌[2],而微生物在生产加工表面形成的生物被膜被认为是主要的交叉污染源头[3]。
大肠杆菌在自然环境中广泛存在,极易污染肉、鱼、乳、蔬菜、水果等,引起食品腐败[4]。致病性大肠杆菌还可引起人类肠道或肠道外感染,甚至会导致死亡[5]。在许多国家,大肠杆菌和大肠菌群通常被认为是食品卫生质量的指示菌[6]。大肠杆菌可以在不锈钢、玻璃、聚苯乙烯等食品加工接触表面及食品表面黏附、定植、形成生物被膜[7]。生物被膜是细菌为了更好地适应生存环境而黏附于接触表面,通过菌体增殖生长、分泌大量胞外聚合物(extracellular polymeric substances,EPS)而形成的具有一定空间结构的细菌聚集体[8]。EPS是生物被膜中细胞周围的厚基质,其多层性质是保护生物被膜内细菌细胞的主要机制[9]。EPS的主要成分是占比高达97%的水,此外还包括胞外多糖、聚合物、蛋白质、核酸、脂质、吸收的营养素及代谢物[10-11]。EPS的存在可以提供细菌物理屏障,减缓抗菌剂向生物被膜内细胞的扩散,从而使酶有时间降解某些毒性分子,使得生物被膜的抗药性增强,难以消除,形成持续性污染[12-13]。
大肠杆菌生物被膜形成的相关基因,如csgA、fimH和papC是附着和感染宿主的重要因子,并且与抗菌素耐药性有关[14]。csgA基因与大肠杆菌在接触面的初始黏附有关,而fimH基因编码F1菌毛的黏附亚基,促进定植、侵入和形成生物被膜。在一些犬类和人体中,papC基因能够使大肠杆菌在内脏中黏附和存活[15]。
为此,本研究首先从生猪屠宰线、猪胴体表面及生鲜猪肉中分离鉴定大肠杆菌,进而采用微孔板结晶紫染色法评价猪肉源大肠杆菌分离株在屠宰加工常见温度(15 ℃)下的生物被膜形成能力,进一步选取代表性菌株探讨生物被膜形成过程中被膜量、EPS组成及大肠杆菌成膜基因表达的变化。
1 材料与方法
1.1 材料与试剂
生鲜猪肉,购于南京农贸市场某品牌专卖柜。
食品级304不锈钢片(75 mm×25 mm×1 mm,2B光洁面)、玻璃容器 南通顺丰医疗器械有限公司;96 孔透明细胞培养板 美国Corning公司;大肠杆菌标准菌株ATCC43895 美国菌种保藏中心;胰蛋白胨大豆琼脂(tryptone soya agar,TSA)、胰蛋白胨大豆肉汤(tryptic soy broth,TSB)、酵母提取物(yeast extract,YE)、大肠杆菌和大肠菌群显色培养基(E.coli and coliform chromogenic agar plates,ECCA)、麦康凯琼脂(MacConkey agar,MAC) 北京陆桥技术有限公司;结晶紫、乙醇、磷酸盐缓冲液(phosphate buffered saline,PBS)、丙酮(均为分析纯) 南京藤春生物科技有限公司;苯酚(分析纯) 南京峰昌生物科技有限公司;浓硫酸(分析纯) 南京化学试剂有限公司;BCA蛋白含量测定试剂盒 南京建成生物科技有限公司;总RNA提取试剂盒、PrimeSTAR® Max DNA聚合酶、荧光定量试剂盒日本TaKaRa公司;DNA Marker(DL 2000)、Gel Red核酸染料 上海生物工程有限公司;Premix Taq酶 苏州近岸蛋白科技有限公司。
1.2 仪器与设备
M2多功能酶标仪 美国Molecular Devices公司;Thermal Cycler普通聚合酶链式反应(polymerase chain reaction,PCR)仪 美国Molecular Devices公司;Universal Hood II凝胶成像仪 美国Bio-Rad公司;2-16KL高速冷冻离心机 美国Sigma公司;Nano-30微量紫外分光光度计 杭州奥盛仪器有限公司;DYCP-32B琼脂糖水平电泳仪 北京六一生物科技有限公司;SW-CJ-2F超净工作台 苏州安泰空气技术有限公司;HVE-50立式压力蒸汽灭菌锅日本Hirayama公司;HX-4拍打式无菌均质器 上海泸析实业有限公司;QL-200微型涡旋混合仪 海门其林贝尔仪器有限公司;ZQTY-70台式振荡培养箱 上海知楚仪器有限公司;DHP-9052电热恒温培养箱 上海一恒科技有限公司。
1.3 方法
1.3.1 肉源大肠杆菌的分离鉴定
1.3.1.1 肉源大肠杆菌的分离纯化
生鲜猪肉样品于超净工作台剪取25 g,放入加有80 mL无菌生理盐水的无菌均质袋中,均质机充分拍打2 min(8 次/s)。某生猪屠宰企业的屠宰车间(传送带表面、操作台表面以及车间工人刀具、手套表面)及猪酮体表面采用棉签涂抹擦拭法取样,取样后用无菌剪刀将棉签头剪于装有10 mL无菌生理盐水的离心管中,用涡旋混匀器充分涡旋。
取一定体积的上述样液进行梯度稀释,选择合适稀释度涂布于ECCA平板,37 ℃培养24 h。培养结束后挑取蓝绿色并呈现圆形凸起的可疑菌落至MAC平板上反复进行平板划线分离,分离纯化3 次[16]。纯化后的可疑菌株于含体积分数25%甘油的TSB中-80 ℃冻存。
1.3.1.2 大肠杆菌属特异性PCR鉴定
将上述冻存的可疑菌株在TSA-YE平板上划线活化2 次,37 ℃倒置培养24 h,挑取单菌落于加入150 μL灭菌蒸馏水的1.5 mL离心管,涡旋,99 ℃水浴10 min,冰浴2 min,12 000×g离心2 min,吸取上清液至另一离心管中即为细菌基因组DNA,-20 ℃冻存备用。
采用PCR扩增大肠杆菌属特异基因phoA,其扩增序列引物:F:5’-CGATGCCTTACCGCTTAC-3’;R:5’-TTTCCCTGCCATTCACC-3’[17]。反应体系(25 μL):模板DNA 1 μL,上、下游引物(10 μmol/L)各0.5 μL,Premix Taq酶12.5 μL,ddH2O 10.5 μL。反应程序为95 ℃预变性5 min,95 ℃变性30 s、55 ℃退火30 s、72 ℃延伸30 s,共30 个循环,72 ℃延伸10 min。扩增完成后进行琼脂糖凝胶电泳(120 V、25 min),凝胶成像仪成像观察。
1.3.2 肉源大肠杆菌生物被膜形成能力比较
1.3.2.1 菌液制备
将冻存的肉源大肠杆菌在TSA-YE平板上划线活化2 次,37 ℃培养24 h。挑取单菌落分别接种于3 mL TSB中,37 ℃、220 r/min振荡培养18 h,连续活化2 次,取生长达到稳定期的菌液1 mL于4 ℃、8 000×g离心5 min,弃去上清,用PBS(pH 7.2)洗涤3 次,调整菌悬液浓度为108 CFU/mL左右,备用。
1.3.2.2 微孔板结晶紫染色法分析大肠杆菌生物被膜形成能力
将上述菌悬液用TSB梯度稀释至102 CFU/mL,吸取180 μL至96 孔细胞培养板中,封口膜封口。置于15 ℃培养72 h,以无菌TSB作为对照。培养结束后,弃去微孔板中培养液,用无菌蒸馏水漂洗3 次,室温下干燥30 min后,每孔加入200 μL 0.25 g/100 mL结晶紫染色30 min,弃去染液后用无菌蒸馏水漂洗3 次,之后用200 μL体积分数95%乙醇溶液洗脱(30 min)黏附于微孔表面的菌体,最后将微孔板置于酶标仪中,在570 nm波长处测定光密度OD570 nm,每个菌株重复测定6 次,结果取平均值。
菌株根据所得OD570 nm进一步作如下分类[18]:无生物被膜形成能力:OD570 nm≤ODc;较弱生物被膜形成能力:ODc<OD570 nm≤2×ODc;中等生物被膜形成能力:2×ODc<OD570 nm≤4×ODc;较强生物被膜形成能力:4×ODc<OD570 nm。其中ODc为临界值,按式(1)计算。
1.3.3 大肠杆菌生物被膜形成曲线
根据1.3.2.2节结果,选取1 株代表菌株按照上述方法进行微孔板操作,分别在培养24、72、120、168 h取样,以无菌TSB作为对照组。经结晶紫染液染色、乙醇洗脱,测定OD570 nm,去除对照组OD570 nm后按照培养时间绘制生物被膜形成曲线。每个时间点均重复测定6 次。
1.3.4 大肠杆菌生物被膜形成时EPS含量分析
1.3.4.1 大肠杆菌生物被膜制备与处理
选用食品级304不锈钢片作为生物被膜形成的接触载体。为防止不锈钢片表面附着的化学杂质及油污影响生物被膜的形成,使用前对不锈钢片进行彻底清洗。具体方法如下:将洗净的钢片放入丙酮溶液中浸泡3 h,拭净后用乙醇溶液浸泡12 h,最后用蒸馏水冲洗干净并晾干[19]。实验前,经121 ℃灭菌20 min备用。
将1.3.2.1节制备的大肠杆菌菌悬液接种至含新鲜TSB和不锈钢片的无菌玻璃容器中,菌液浓度约为106 CFU/mL。无菌保鲜膜封口,15 ℃下静置培养72 h和168 h。培养结束后取出不锈钢片并用无菌生理盐水漂洗3 次以去除表面未黏附菌体,采用棉签擦拭法收集不锈钢片表面的生物被膜。将拭子置于装有10 mL无菌生理盐水和一定量玻璃珠的离心管中,充分涡旋3 min使拭子上的菌体充分脱落,得到的菌悬液用于EPS含量测定。实验重复3 次。
1.3.4.2 EPS中蛋白质和多糖含量测定
参考Cao Bin等[20]的方法制备1.3.4.1节菌悬液中的松散型EPS(loose EPS,L-EPS)和紧密型EPS(bound EPS,B-EPS)。L-EPS和B-EPS的蛋白含量采用BCA蛋白含量检测试剂盒测定。蛋白质-吸光度的标准曲线方程为y=0.001 8x+0.151 4(R2=0.997 8),其中x为蛋白质质量浓度/(μg/mL),y为吸光度,根据标准曲线计算EPS中蛋白质含量。总糖含量采用苯酚硫酸法[21]测定,多糖-吸光度的标准曲线方程为y=0.195 6x+0.081 8(R2=0.997 3),其中x为葡萄糖质量浓度/(μg/mL),y为吸光度,根据标准曲线计算EPS中多糖含量。
1.3.5 生物被膜中相关成膜基因表达量测定
1.3.5.1 生物被膜的收集
以食品级304不锈钢片作为接触载体,15 ℃下静置培养72 h和168 h后收集生物被膜,方法同1.3.4.1节。将菌悬液4 ℃、12 000×g离心15 min,沉淀用于核酸提取。
1.3.5.2 总RNA提取及cDNA合成
按照总RNA提取试剂盒说明书进行,用核酸蛋白微量测定仪测定提取的总RNA浓度和纯度,确定RNA浓度和纯度合格(OD260 nm/OD280 nm为1.8~2.0),调整RNA浓度后,根据Prime ScriptTM RT Master Mix反转录试剂盒进行反转录。反转录体系(10 μL):2 μL 5×Prime Script RT Master Mix混合液、2 μL RNA、6 μL无RNA酶水;反应程序:37 ℃、15 min,85 ℃反转录酶失活反应5 s,保持4 ℃。将反转录得到的cDNA于-20 ℃保存进行后续实验。
1.3.5.3 标准质粒构建及标准曲线建立
大肠杆菌csgA、papC、fimH基因及引物情况见表1,引物由上海生物工程有限公司合成。
表1 实验所用PCR引物
Table 1 PCR primers used in this study
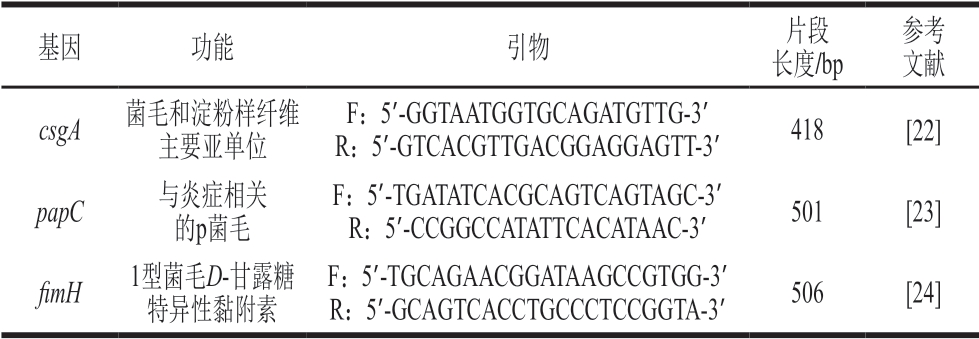
基因功能引物片段长度/bp参考文献csgA菌毛和淀粉样纤维主要亚单位F:5’-GGTAATGGTGCAGATGTTG-3’R:5’-GTCACGTTGACGGAGGAGTT-3’418[22]papC与炎症相关的p菌毛F:5’-TGATATCACGCAGTCAGTAGC-3’R:5’-CCGGCCATATTCACATAAC-3’501[23]fimH1型菌毛D-甘露糖特异性黏附素F:5’-TGCAGAACGGATAAGCCGTGG-3’R:5’-GCAGTCACCTGCCCTCCGGTA-3’506[24]
标准质粒由南京擎科公司构建合成。取测序结果正确的质粒作为标准质粒,采用微量分光光度计测定纯度和质量浓度,按式(2)计算质粒DNA拷贝数。
使用无RNA酶水梯度稀释,使标准检测质粒的质量浓度为100~10-6 ng/μL,作为模板。PCR反应体系(20 μL):SYBR Green Master(2×)(ROX)10 μL,PCR正、反向引物(10 μmol/L)各0.25 μL,DNA模板2 μL,双蒸水7.5 μL。反应条件:95 ℃预变性30 s后,95 ℃5 s、60 ℃ 30 s反应40 个循环,95 ℃ 15 s、60 ℃ 1 min、95 ℃保持15 s。以拷贝数的对数值(lg(copies/μL))为横坐标,循环阈(cycle threshold,Ct)值为纵坐标,构建一元线性方程,即为标准曲线。
1.3.5.4 相关成膜基因荧光定量PCR分析
以1.3.5.2节制备的cDNA为模板进行荧光定量PCR,用所得样品的Ct值通过标准曲线计算目标基因表达量(lg(copies/μL)),进一步计算不锈钢片生物被膜中成膜基因表达量(copies/cm2)。
1.4 数据处理
利用GraphPad Prism 8软件(GraphPad Software公司)处理相应数据及图表分析,采用SPSS 17.0(IBM公司)对数据进行单因素方差分析和Duncan’s多重比较检验,2 组之间显著性差异分析采用t检验。结果以平均值±标准差表示,P<0.05时差异显著。
2 结果与分析
2.1 肉源大肠杆菌的分离与鉴定
由图1可知,ECCA大肠杆菌显色平板中共分离得到蓝绿色可疑菌落350 个,可疑菌落进一步采用MAC平板划线分离纯化,得到粉红色可疑菌株90 株。采用大肠杆菌菌属特异性PCR扩增phoR基因,由图2可知,扩增出阳性条带(444 bp),共鉴定到阳性菌株31 株,其中4 株来源于生猪胴体表面,11 株来源于生猪屠宰线,16 株来源于生鲜猪肉,菌株信息见表2。
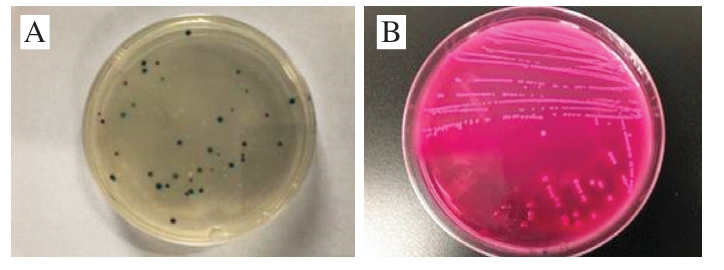
图1 大肠杆菌在ECCA平板(A)和MAC平板(B)上的生长情况
Fig.1 Growth of E.coli on ECCA (A) and MAC plates (B)
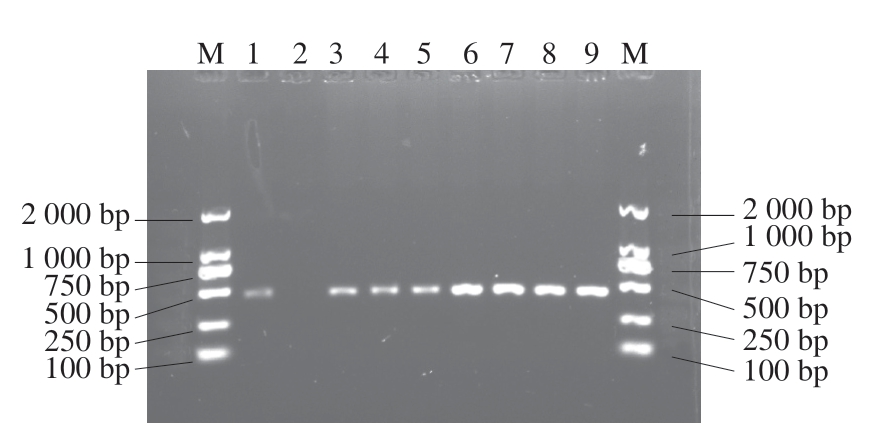
图2 部分菌株的属特异性PCR产物电泳图
Fig.2 Electrophoresis profile of genus-specific PCR products of selected isolates
泳道M.DNA Marker;泳道1.阳性对照大肠杆菌ATCC43895;泳道2.空白对照;泳道3~9.部分被测菌株。
表2 大肠杆菌鉴定结果
Table 2 Results of identification of E.coli

菌株编号来源鉴定结果D-21、C-15、C-3、C-12、A-9、A-15、A-13、D-15、B-7、D-6、C-13生猪屠宰线S1-4、S2-4、S2-6、S1-14猪胴体表面D4-11、D3-11、D4-27、D4-18、D4-25、D4-9、D4-13、D4-12、D4-5、D4-22、D2-18、D3-12、D4-26、D2-17、D4-10、D4-14大肠杆菌生鲜猪肉
大肠杆菌是人和动物肠道共生菌群的组成部分,部分致病性大肠杆菌能引起人体感染而致病[25]。受屠宰加工方式及运输条件等因素影响,猪肉从屠宰场到餐桌的过程中,会受到不同程度的微生物污染[26]。国内有关学者对不同环境中大肠杆菌的污染情况进行了许多调查。吴萱[27]、于庆华[28]等的研究均从猪肉流通的不同环节中分离到大肠杆菌,可能来源于畜禽肠道及体表、屠宰、贮存、运输、销售环境,也有可能来源于屠宰或销售人员。本研究从生猪屠宰线、猪胴体表面及生鲜猪肉表面分离到大肠杆菌,证实了大肠杆菌在猪肉生产环节中普遍存在,表明生产加工环境中加强卫生监控对控制猪肉微生物污染和降低食品安全隐患十分必要。
2.2 大肠杆菌生物被膜形成能力分析
采用微孔板结晶紫染色法评估31 株肉源大肠杆菌的生物被膜形成能力,大多菌株均可在微孔板底部见到一层淡紫色薄膜,加入乙醇溶解后,不同菌株孔板中颜色深浅不同。进一步测定OD570 nm,由图3可知,不同菌株间生物被膜形成能力存在显著差异。31 株肉源大肠杆菌中,27 株菌株表现出生物被膜形成能力。按照临界值ODc的生物被膜形成能力进行分类,强、中、弱、无4 种生物被膜表型的菌株数量分别为3、4、20、4 株,分别占菌株总数的9.68%、12.90%、64.52%和12.90%。
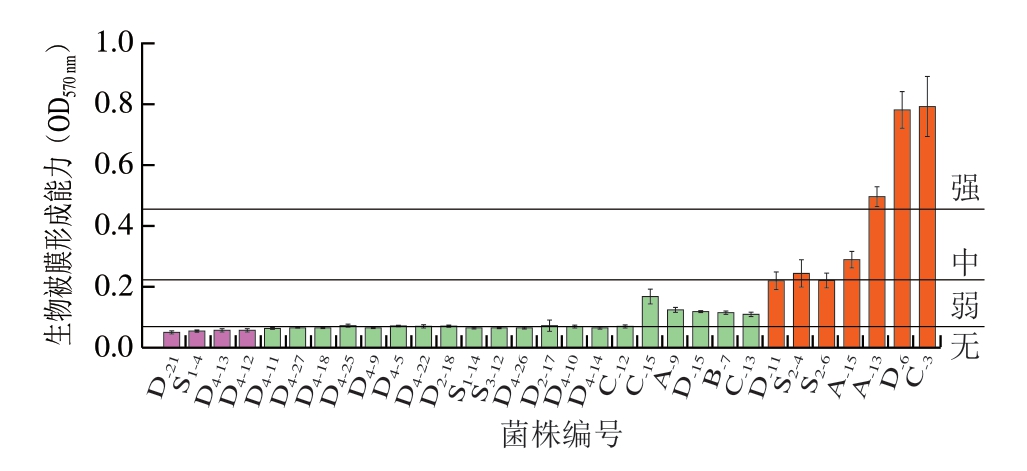
图3 31 株大肠杆菌微孔板表面生物被膜形成能力
Fig.3 Biofilm formation ability of 31 isolates of E.coli on polystyrene microplate surfaces
在实际生产中,生猪屠宰车间的温度为13.28~16.70 ℃,平均(15.85±0.99) ℃[29]。因此本研究结合生产实际,探讨15 ℃条件下肉源大肠杆菌生物被膜形成情况。研究表明,大多数食源性腐败菌,如假单胞菌[30]、希瓦氏菌[31]等均具有一定的生物被膜形成能力且存在明显菌株差异。本研究亦发现,肉源大肠杆菌的生物被膜形成能力存在明显菌株差异,且大多数大肠杆菌生物被膜形成能力较弱。这与前人的一些报道类似,王娴静等[32]用微孔板结晶紫法评价14 株大肠杆菌O157:H7的生物被膜形成能力,发现所有菌株均具有一定的生物被膜形成能力,其中85.7%菌株成膜能力较弱且存在明显的菌株差异性。即使大肠杆菌的成膜能力较弱,一旦形成生物被膜,对清洁剂、消毒剂等的抗性增强,不易清除,则可能成为持续的污染源头。因此探讨肉类屠宰加工环境及肉中大肠杆菌的生物被膜形成对生鲜肉安全生产和污染控制具有科学指导意义。
本研究中肉源大肠杆菌中60%以上菌株的生物被膜形成能力较弱,从这些菌株中随机选取1 株大肠杆菌D4-18进行后续实验。
2.3 结晶紫染色法检测大肠杆菌D4-18生物被膜的生长过程
采用结晶紫染色法研究大肠杆菌D4-18生物被膜的形成过程。由图4可知,大肠杆菌D4-18生物被膜在24 h时形成量较少,随着时间延长,OD570 nm逐渐增大,即生物被膜形成量逐渐增多,168 h时生物被膜量达到最大值。研究表明,生物被膜形成过程是一个动态的过程,分为起始黏附期、集聚增殖期、成熟期及脱落散播期4 个阶段[33]。李安琪等[34]发现,副溶血弧菌在微孔板中37 ℃培养48~72 h时达到生物被膜的成熟阶段,72~144 h时达到生物被膜的解离阶段。邓小玲等[35]报道了大肠杆菌在微孔板中37 ℃条件下形成生物被膜的动态过程,发现有部分大肠杆菌在72 h后进入脱落期。王娴静等[32]研究大肠杆菌O157:H7在微孔板中25 ℃条件下生物被膜形成过程,发现成膜能力较强的菌株J29培养48 h后生物被膜量缓慢增多,成膜能力中等的菌株ATCC43895在60 h后生物被膜量增长较快,168 h时达到最多,其他成膜能力弱的菌株在168 h内生物被膜量都维持在较低的相对稳定状态。本研究中,大肠杆菌D4-18在微孔板中15 ℃条件下培养168 h生物被膜量持续增长。上述研究表明,生物被膜形成过程与菌种、菌株的生物被膜形成能力、培养温度等成膜条件有关。
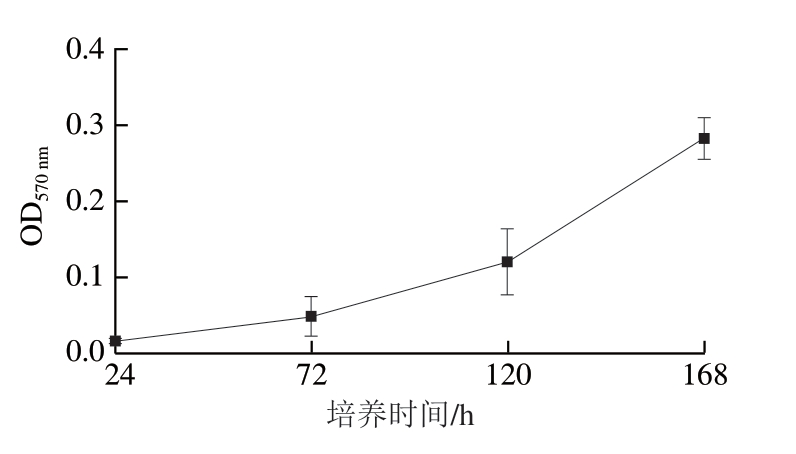
图4 大肠杆菌D4-18生物被膜形成曲线
Fig.4 Curve of biofilm formation of E.coli D4-18
2.4 大肠杆菌D4-18生物被膜形成时EPS中蛋白质和多糖含量变化
由图5可知,成膜72 h时大肠杆菌D4-18产生了一定量胞外蛋白质和胞外多糖,随着培养时间延长,EPS含量增加,其中L-EPS中蛋白质及多糖的含量均极显著增加(P<0.01),B-EPS中蛋白质含量极显著增加(P<0.01)。EPS基质在生物被膜发育、发展和成熟阶段发挥重要作用,与菌体的黏附、支架、机械稳定性和保护作用密切相关[36]。EPS中蛋白质与多糖的含量可以在一定程度上反映生物被膜发育程度。蛋白质中含有的氨基酸及部分疏水性氨基酸增加了细菌细胞表面疏水性[37-38],而多糖中含有的羧基、羟基等亲水官能团增加了细胞的亲水性。使得EPS同时具有疏水和亲水的特性。多糖的黏性可以更好包裹细菌,使得菌体更加稳定地黏附在非生物表面。蛋白的疏水性增强了菌体在疏水性的不锈钢表面定植、聚集、形成生物被膜和获取营养的能力,从而增强了细菌对外界恶劣环境的抵抗力[39]。
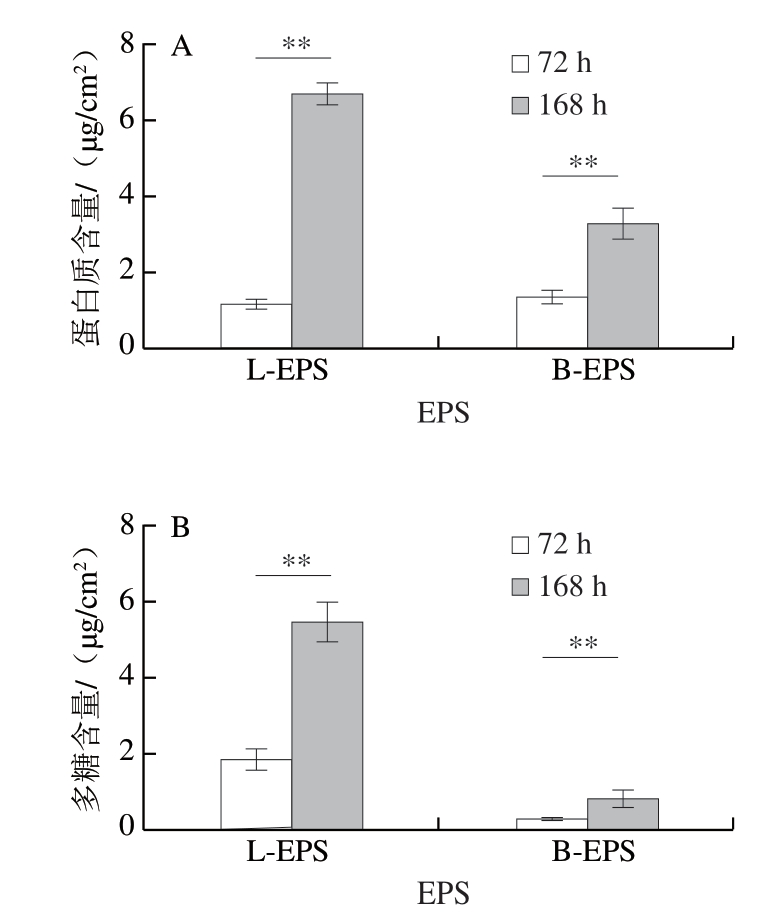
图5 大肠杆菌D4-18生物被膜中L-EPS和B-EPS蛋白质(A)及多糖(B)含量
Fig.5 Content of protein (A) and polysaccharide (B) in L-EPS and B-EPS in the biofilm of E.coli D4-18
**.差异极显著(P<0.01)。图6同。
2.5 生物被膜中相关成膜基因的表达变化
大肠杆菌D4-18的3 种成膜基因papC、fimH、csgA标准曲线如表3所示,相关系数R2均大于0.99,Ct值满足标准曲线要求。
表3 papC、fimH、csgA基因标准曲线
Table 3 Standard curves for detection of papC, fimH, and csgA genes

成膜基因标准曲线方程R2 papCy=-3.342x+36.5830.993 fimHy=-3.359x+35.8670.997 csgAy=-3.089x+34.6950.996
由图6可知,大肠杆菌在15 ℃条件下培养72 h时,papC、fimH、csgA基因的表达量分别为0.095、0.933、0.435 copies/cm2,培养168 h时papC基因表达量显著增多(P<0.05),fimH基因表达量极显著增多(P<0.01),csgA基因表达量无显著变化。关于大肠杆菌生物被膜形成时相关基因表达方面有较多报道,朱琳等[40]研究发现,fimA基因和fliC基因编码菌毛属细菌的黏附结构,在生物被膜的初始黏附阶段起重要作用,flu基因编码的Ag43能促进细菌的自聚集,有利于细菌生物被膜团聚结构的形成。Surgers等[41]研究发现,papC基因的存在与生物被膜形成有很强的相关性。Ren等[42]采用DNA微阵列研究大肠杆菌生物被膜的基因表达,发现fimH基因在生物被膜形成时表达上调。Bhardwaj等[43]采用荧光定量PCR相对定量发现,csgA基因在大肠杆菌生物被膜形成时表达上调。本研究采用荧光定量PCR绝对定量同样发现,在不锈钢接触表面形成的生物被膜中,papC、fimH、csgA基因进行了一定表达,且在生物被膜不同成膜阶段,接触面上相关基因表达量发生变化,表明这些基因在大肠杆菌生物被膜形成过程中发挥了不同程度的调控作用。
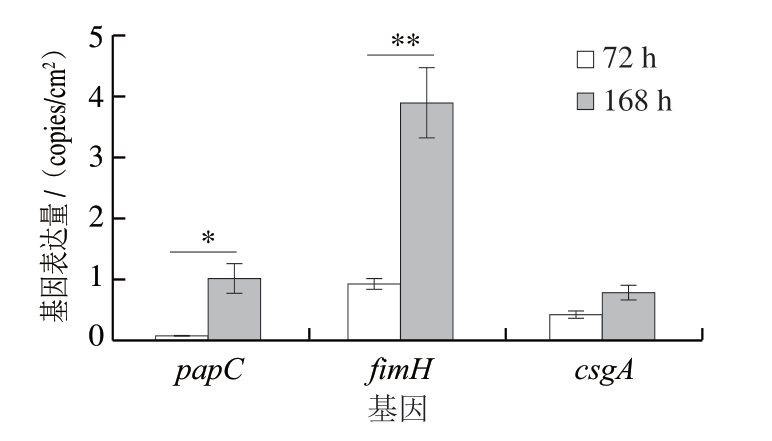
图6 大肠杆菌生物被膜相关基因表达的变化
Fig.6 Changes in the expression of biofilm related-genes of E.coli
*.差异显著(P<0.05)。
需要指出的是,在实际生产环境中,多种微生物共存、多菌种混合生物被膜是更常见的存在状态,混合生物被膜内部不同菌种之间相互作用将导致基因表达水平发生变化,形成更加紧密、复杂的微系统,生物被膜的结构和特性更复杂,抗性可能更强[44-45]。因此混合生物被膜的形成和控制值得进一步关注和重视。
3 结 论
从生猪屠宰线、猪胴体表面及生鲜猪肉表面均分离到大肠杆菌,证实了大肠杆菌在猪肉生产环节中普遍存在。大部分大肠杆菌可以形成生物被膜,虽然它们的被膜形成能力大多较弱,但生物被膜抗性强,增加了食品安全隐患,所以在生猪屠宰、猪肉加工、销售各环节应加强卫生管理,加大清洁消毒,减少细菌及其生物被膜造成的污染。本研究发现,在大肠杆菌生物被膜形成过程中发生EPS的分泌,并受不同基因调控,这为大肠杆菌生物被膜的靶向控制提供了科学思路。本研究可为揭示肉类生产加工中大肠杆菌生物被膜的形成规律提供理论基础。
[1] WANG Rong.Biofilms and meat safety: a mini-review[J].Journal of Food Protection, 2019, 82(1): 120-127.DOI:10.4315/0362-028x.jfp-18-311.
[2] BACON R T, SOFOS J N, SCHMIDT R H, et al.Characteristics of biological hazards in foods[M].New York: John Wiley & Sons, 2005:157-195.DOI:10.1002/047172159X.ch10.
[3] GALIE S, GARCIA-GUTIERREZ C, MIGUELEZ E M, et al.Biofilms in the food industry: health aspects and control methods[J].Frontiers in Microbiology, 2018, 9: 898.DOI:10.3389/fmicb.2018.00898.
[4] VAZQUEZ-SANCHEZ D, GALVAO J A, OETTERER M.Contamination sources, biofilm-forming ability and biocide resistance of Shiga toxin-producing Escherichia coli O157:H7 and non-O157 isolated from tilapia-processing facilities[J].Journal of Food Safety,2018, 38(3): e12446.DOI:10.1111/jfs.12446.
[5] CLEMENTS A, YOUNG J C, CONSTANTINOU N, et al.Infection strategies of enteric pathogenic Escherichia coli[J].Gut Microbes,2012, 3(2): 71-87.DOI:10.4161/gmic.19182.
[6] METZ M, SHEEHAN J, FENG P C H.Use of indicator bacteria for monitoring sanitary quality of raw milk cheeses: a literature review[J].Food Microbiology, 2020, 85: 103283.1-103283.11.DOI:10.1016/j.fm.2019.103283.
[7] 程传松, 闫晓彤, 李长城, 等.食品接触表面食源性致病菌生物膜形成及控制研究进展[J].包装工程, 2021, 42(13): 1-8.DOI:10.19554/j.cnki.1001-3563.2021.13.001.
[8] FLEMMING H C, WINGENDER J.The biofilm matrix[J].Nature Reviews Microbiology, 2010, 8(9): 623-633.DOI:10.1038/nrmicro2415.
[9] BUCK L D, PALADINO M M, NAGASHIMA K, et al.Temperaturedependent influence of FliA overexpression on PHL628 E.coli biofilm growth and composition[J].Frontiers in Cellular and Infection Microbiology, 2021, 11: 775270.DOI:10.3389/fcimb.2021.775270.
[10] BELOIN C, ROUX A, GHIGO J M.Escherichia coli biofilms[J].Current Topics in Microbiology and Immunology, 2008, 322: 249-289.DOI:10.1007/978-3-540-75418-3_12.
[11] GUILHEN C, MIQUEL S, CHARBONNEL N, et al.Colonization and immune modulation properties of Klebsiella pneumoniae biofilm dispersed cells[J].NPJ Biofilms and Microbiomes, 2019, 5(1): 1-11.DOI:10.1038/s41522-019-0098-1.
[12] LEWIS K.Riddle of biofilm resistance[J].Antimicrobial Agents and Chemotherapy, 2001, 45(4): 999-1007.DOI:10.1128/AAC.45.4.999-1007.2001.
[13] PENESYAN A, NAGY S S, KJELLEBERG S, et al.Rapid microevolution of biofilm cells in response to antibiotics[J].NPJ Biofilms and Microbiomes, 2019, 5(1): 1-14.DOI:10.1038/s41522-019-0108-3.
[14] WANG Xiangru, WEI Liuya, WANG Bin, et al.Complete genome sequence and characterization of avian pathogenic Escherichia coli field isolate ACN001[J].Standards in Genomic Sciences, 2016, 11(1):1-7.DOI:10.1186/s40793-015-0126-6.
[15] GRAKH K, MITTAL D, PRAKASH A, et al.Characterization and antimicrobial susceptibility of biofilm-producing avian pathogenic Escherichia coli from broiler chickens and their environment in India[J].Veterinary Research Communications, 2022, 46(2): 537-548.DOI:10.21203/rs.3.rs-665570/v1.
[16] YANG Xianqin, WANG Hui, HE A, et al.Microbial efficacy and impact on the population of Escherichia coli of a routine sanitation process for the fabrication facility of a beef packing plant[J].Food Control, 2017, 71: 353-357.DOI:10.1016/j.foodcont.2016.07.016.
[17] 张亚楠, 焦仁刚, 赵贵, 等.牛奶中3 种致病菌多重PCR的建立及初步应用[J].中国乳品工业, 2020, 48(3): 47-51.DOI:10.19827/j.issn1001-2230.2020.03.10.
[18] PATEL H, GAJJAR D.Cell adhesion and twitching motility influence strong biofilm formation in Pseudomonas aeruginosa[J].Biofouling,2022, 38(3): 235-249.DOI:10.1080/08927014.2022.2054703.
[19] BELESSI C E A, GOUNADAKI A S, PSOMAS A N, et al.Efficiency of different sanitation methods on Listeria monocytogenes biofilms formed under various environmental conditions[J].International Journal of Food Microbiology, 2011, 145: S46-S52.DOI:10.1016/j.ijfoodmicro.2010.10.020.
[20] CAO Bin, SHI Liang, BROWN R N, et al.Extracellular polymeric substances from Shewanella sp HRCR-1 biofilms: characterization by infrared spectroscopy and proteomics[J].Environmental Microbiology,2011, 13(4): 1018-1031.DOI:10.1111/j.1462-2920.2010.02407.x.
[21] LOUSTAU E, ROLS J L, LEFLAIVE J, et al.Comparison of extraction methods for the characterization of extracellular polymeric substances from aggregates of three biofilm-forming phototrophic microorganisms[J].Canadian Journal of Microbiology, 2018, 64(11):887-899.DOI:10.1139/cjm-2018-0182.
[22] LEE J H, KIM Y G, RYU S Y, et al.Resveratrol oligomers inhibit biofilm formation of Escherichia coli O157:H7 and Pseudomonas aeruginosa[J].Journal of Natural Products, 2014, 77(1): 168-172.DOI:10.1021/np400756g.
[23] SCHIEBEL J, BOHM A, NITSCHKE J, et al.Genotypic and phenotypic characteristics associated with biofilm formation by human clinical Escherichia coli isolates of different pathotypes[J].Applied and Environmental Microbiology, 2017, 83(24): e01660-17.DOI:10.1128/aem.01660-17.
[24] CHEN Tianpeng, LIU Na, REN Peifang, et al.Efficient biofilm-based fermentation strategies for L-threonine production by Escherichia coli[J].Frontiers in Microbiology, 2019, 10: 1773.DOI:10.3389/fmicb.2019.01773.
[25] 唐云云, 韩瑞, 张文波, 等.波尔山羊肠外致病性大肠杆菌的分离鉴定及药敏试验[J].今日畜牧兽医, 2022, 38(7): 1-3; 7.
[26] 邱燕, 温贵兰, 张升波.定点屠宰场(点)猪肉中三种主要食源性致病菌污染情况调查[J].中国动物检疫, 2018, 35(8): 23-28.DOI:10.3969/j.issn.1005-944X.2018.08.006.
[27] 吴萱, 杨璐, 刘艳超, 等.北京市售鸡肉和猪肉中大肠杆菌污染情况及耐药特征分析[J].中国食品卫生杂志, 2022, 34(2): 211-216.DOI:10.13590/j.cjfh.2022.02.004.
[28] 于庆华.不同动物源食品中大肠杆菌分离鉴定与耐药性分析[J].饲料研究, 2019, 42(2): 50-52.DOI:10.13557/j.cnki.issn1002-2813.2019.02.016.
[29] 刘寿春, 赵春江, 杨信廷, 等.猪肉冷链加工与物流微生物危害分析与控制[J].食品科技, 2012, 37(5): 103-109.DOI:10.13684/j.cnki.spkj.2012.05.041.
[30] YUAN L, BURMOLLE M, SADIQ F A, et al.Interspecies variation in biofilm-forming capacity of psychrotrophic bacterial isolates from Chinese raw milk[J].Food Control, 2018, 91: 47-57.DOI:10.1016/j.foodcont.2018.03.026.
[31] 张雯, 卞丹, 阮成旭, 等.大黄鱼源腐败菌的黏附特性与生物膜特性分析[J].食品科学, 2019, 40(14): 84-90.DOI:10.7506/spkx1002-6630-20180730-369.
[32] 王娴静, 董晨, 禹金龙, 等.酸应激对大肠杆菌O157:H7生物菌膜形成的影响[J].食品科学, 2019, 40(19): 1-7.DOI:10.7506/spkx1002-6630-20181018-193.
[33] OTTO M.Virulence factors of the coagulase-negative staphylococci[J].Frontiers in Bioscience, 2004, 9: 841-863.DOI:10.2741/1295.
[34] 李安琪, 石成龙, 钱森和, 等.副溶血性弧菌生物被膜动态形成机制的转录组分析[J/OL].食品科学: 1-16[2022-08-22].http://kns.cnki.net/kcms/detail/11.2206.TS.20220612.1523.002.html.
[35] 邓小玲, 孙影, 尤向峰, 等.禽致病性大肠杆菌生物被膜形成能力及相关特性分析[J].南京农业大学学报, 2021, 44(5): 918-926.DOI:10.7685/jnau.202011020.
[36] 刘静聪, 方金玉, 朱军莉.细菌生物被膜基质的研究进展[J].微生物学报, 2022, 62(1): 47-56.DOI:10.13343/j.cnki.wsxb.20210168.
[37] 廖青, 李小明, 杨麒, 等.好氧颗粒污泥的快速培养以及胞外多聚物对颗粒化的影响研究[J].工业用水与废水, 2008(4): 13-19.DOI:10.3969/j.issn.1009-2455.2008.04.004.
[38] 季思彤, 李进, 吴芳芳, 等.再生水中SS304表面生物膜的活性与结构[J].环境工程学报, 2017, 11(3): 1920-1926.DOI:10.12030/j.cjee.201511196.
[39] 曹月, 郭鹏, 朱莹, 等.高效柴油降解菌细胞表面疏水性及降解特性[J].环境与健康杂志, 2018, 35(12): 1078-1084.DOI:10.16241/j.cnki.1001-5914.2018.12.013.
[40] 朱琳, 王亚楠, 闫运发, 等.犊牛腹泻源大肠杆菌生物被膜形成相关基因检测及菌株致病力测定[J].畜牧与兽医, 2015, 47(7): 31-36.
[41] SURGERS L, BOYD A, GIRARD P M, et al.Biofilm formation by ESBL-producing strains of Escherichia coli and Klebsiella pneumoniae[J].International Journal of Medical Microbiology, 2019,309(1): 13-18.DOI:10.1016/j.ijmm.2018.10.008.
[42] REN D, BEDZYK L A, THOMAS S M, et al.Gene expression in Escherichia coli biofilms[J].Applied Microbiology and Biotechnology,2004, 64(4): 515-524.DOI:10.1007/s00253-003-1517-y.
[43] BHARDWAJ D K, TANEJA N K, SHIVAPRASAD D P, et al.Phenotypic and genotypic characterization of biofilm forming,antimicrobial resistant, pathogenic Escherichia coli isolated from Indian dairy and meat products[J].International Journal of Food Microbiology, 2021, 336: 108899.DOI:10.1016/j.ijfoodmicro.2020.108899.
[44] VAZQUEZ-SANCHEZ D, CABO M L, IBUSQUIZA P S, et al.Biofilm-forming ability and resistance to industrial disinfectants of Staphylococcus aureus isolated from fishery products[J].Food Control,2014, 39: 8-16.DOI:10.1016/j.foodcont.2013.09.029.
[45] 陈忠军, 张钰皎, 胡炜东, 等.食源性混合病原菌生物被膜行为对金黄色葡萄球菌毒素因子转录水平的影响[J].中国食品学报, 2021,21(6): 287-295.DOI:10.16429/j.1009-7848.2021.06.034.