宣威火腿是我国云南省的传统名特优肉制品[1],以营养丰富、滋味鲜美、油而不腻、香气浓郁著称于世,它的主要特点是:形似琵琶、只大骨小、皮薄肉厚、肥瘦适中,切开断面香气浓郁、色泽鲜艳,瘦肉呈鲜红色或玫瑰色,肥肉呈乳白色,骨头略显桃红,给人以自然协调的美感[2]。宣威独特的地理气候造就了火腿腌制、发酵的天然环境。宣威火腿盐含量一般为9.1%~11.2%[3],口感偏咸,属于高盐肉制品,然而,已有大量研究表明,过量摄入食盐是目前导致高血压和心血管疾病等的重要因素[4-6]。因此,严重影响消费者健康及火腿产业的发展,成为导致宣威火腿优势不优、名牌不名、产业不强、效益不大的因素之一。
在火腿加工生产过程中,食盐能使肉制品脱水,起到抑制肉制品中微生物增殖的作用[7],还能使火腿中蛋白质发生变性与重组,从而形成稳定的凝胶结构,在一定范围内改善火腿制品的嫩度与多汁性[8]。因此在低钠肉制品中,会影响产品的品质,丧失原有风味,并且更容易造成腐败变质。在肉制品加工过程中使用NaCl的替代品已成为必然的趋势。NaCl的替代品主要有氯化钾(KCl)、氯化镁、氯化钙、乳酸钾、抗坏血酸钙和甘氨酸等[9],因KCl与NaCl性质相似,目前为止仍然是最常见的钠盐替代品之一,虽然KCl被广泛用于各种肉制品的研究中,但肉制品种类繁多,工艺条件各异,不同替代盐对产品的作用可能存在一定差异。一项新的建模研究表明,在中国使用钾盐替代钠盐每年可防止约50 万人死于心血管疾病[10]。因此,以钾盐替代钠盐应用于肉制品中有益于人体健康,但KCl的替代量与产品品质之间的关系仍需进一步研究。
以往的研究表明,火腿的特殊风味是由火腿发酵过程中经过一系列复杂的生化反应产生的,包括脂质氧化、美拉德反应和蛋白质降解等[11-12]。随着火腿的不断发酵,火腿的理化性质发生变化,从而影响最终产品的品质,这些变化主要依赖于微生物内源酶的催化作用[13-14]。 近年来,火腿中微生物群落的组成和优势种的鉴定得到了广泛研究。李平兰等[15]通过分析宣威火腿成熟产品中主要微生物菌相构成,发现宣威火腿中的优势菌群主要为葡萄球菌、微球菌和霉菌,耐盐性的葡萄球菌和微球菌的代谢活动以及火腿表面大量霉菌的生长是宣威火腿独特风味形成的基础。Wang Xinghong等[16]对宣威火腿天然微生物的研究发现,酵母是对宣威火腿品质起重要作用的主要微生物。此外,邹颖玲等[17]基于聚合酶链式反应(polymerase chain reaction,PCR)-变性梯度凝胶电泳技术分析宣威火腿中真菌群落结构,从不同加工年份的宣威火腿中共检测到27 种真菌,其中假灰绿曲霉(Aspergillus pseudoglaucus)是宣威火腿的优势菌种,加工3 年宣威火腿表面的真菌群落多样性最高,加工2 年宣威火腿表面最低;而加工2 年宣威火腿内部真菌群落多样性最高,加工3 年宣威火腿内部最低。此外,高通量测序技术是表征微生物群落多样性的有效分子工具,已成功用于测定许多肉制品生产或贮藏过程中微生物种群的变化[18-19],也具有独特的优势,如能在较短时间内识别不可培养微生物[20]。
使用KCl替代NaCl腌制宣威火腿是否影响宣威火腿中的微生物具有重要的研究意义。基于此,本研究以30%~60% KCl替代NaCl对宣威火腿进行腌制,对不同KCl替代量的宣威火腿细菌多样性和群落结构进行研究,旨在为低钠盐宣威火腿的开发提供理论依据,为全面提升产品品质提供科学指导。
1 材料与方法
1.1 材料与试剂
选用同批次饲养10 月龄的含乌金猪血统的宣和猪15 头,统一屠宰后取猪后腿30 条作为原料腿,平均腿质量为(14.77±0.72) kg,于云南省宣威市浦记火腿食品有限公司进行腌制、发酵、成熟。
食盐(食品级) 云南省盐业有限公司;KCl(食品级) 连云港科德化工有限公司;DNA提取试剂盒 美国Bio Tek公司;AxyPrepDNA凝胶回收试剂盒 美国Axygen公司。
1.2 仪器与设备
GeneAmp® 9700 PCR仪 美国ABI公司;QuantiFluorTM-ST蓝色荧光定量系统 美国Promega公司;移液器 德国Eppendorf公司;DYY-6C电泳仪 北京六一生物科技有限公司;Gel-Doc2000凝胶成像仪 美国Bio-Rad公司;MiSeq测序仪 美国Illumina公司。
1.3 方法
1.3.1 实验设计
采用单因素试验设计,将30 条猪后腿随机分成5 组,每组6 条。根据国内外干腌肉制品KCl替代NaCl比例的相关研究,最大可接受替代比例 为50%~60%[21-22],结合已有研究结果设计KCl的替代比例。本研究以100% NaCl为对照,使用KCl分别替代30%、40%、50%和60% NaCl制备宣威火腿。
1.3.2 宣威火腿的加工
火腿的加工按照传统宣威火腿的加工工艺进行。各组KCl的替代量分别与对应的食盐混合,腌制时食盐和钾盐的添加量为鲜腿质量的6.5%。宣威火腿的加工参照包媛媛等[23]的方法进行,其工艺流程主要包括修割定型、上盐腌制、堆码翻压、洗晒整形、上挂风干和发酵管理等基本步骤。火腿的加工及发酵均于云南省宣威市浦记火腿食品有限公司进行。
发酵成熟的宣威火腿经洗霉修割,取各处理组火腿股二头肌样品为实验材料,样品取出后真空封口,于-80 ℃冰箱保存待分析。宣威火腿的品质测定于云南省畜产品加工工程技术研究中心进行。
1.3.3 宣威火腿微生物多样性的测定
1.3.3.1 样品制备
按照KCl替代比例0%、30%、40%、50%、60%的顺序,将每组火腿样品依次对应编号为A、B、C、D、E组,同时按表面(S)和内部(N)进行编号。
表面取样:在每只火腿三签点(上、中、下签)表面均匀选取,将无菌的脱脂棉签用无菌生理盐水浸湿后在选取区域擦拭,每个点擦拭30 s,擦拭面积4 cm2,取样后将棉拭子头(3 个)剪下立即放入无菌冻存管,液氮速冻,-80 ℃保存。
内部取样:在每只火腿三签点,在火腿表面切除0.5 cm厚、边长4 cm的四方形弃置,然后在此处切割厚度为2 cm的肉块,取出后在酒精灯上进行灼烧消毒,立即放入无菌冻存管,液氮速冻,-80 ℃保存。
1.3.3.2 微生物多样性测序
测序流程:样品DNA提取→设计合成引物接 头→PCR扩增与产物纯化→PCR产物定量与均一化→ 构建MiSeq PE文库→Illumina测序
1)DNA提取及检测:使用基因组DNA提取试剂盒对宣威火腿微生物基因组DNA进行提取,利用1%琼脂糖凝胶电泳检测抽提的基因组DNA。
2)PCR扩增:以样品的基因组DNA为模板进行PCR扩增,细菌用16S rRNA的V3~V4区扩增,其引物为338F(5’-ACTCCTACGGGAGGCAGCAG-3’)和805R(5’-GG-ACTACHVGGGTWTCTAAT-3’);PCR反应体系和扩增程序为:5×Fast Pfu缓冲液4 μL、2.5 mmol/L脱氧核糖核苷酸2 μL、上游引物(5 μmol/L)0.8 μL、下游引物(5 μmol/L)0.8 μL、Fast Pfu聚合酶0.4 μL、牛血清白蛋白0.2 μL、模板DNA 10 ng,补ddH2O至20 μL。反应条件:95 ℃预变性3 min;95 ℃变性30 s,50 ℃退火60 s,72 ℃退火45 s,扩增循环35 次;72 ℃延伸10 min。每个样本3 个重复,将同一样本的PCR产物混合后,用2%琼脂糖凝胶电泳检测,使用AxyPrepDNA凝胶回收试剂盒切胶回收PCR产物,Tris-HCl洗脱,用2%琼脂糖凝胶电泳检测。
3)荧光定量:参照电泳初步定量结果,将PCR产物用QuantiFluor™-ST蓝色荧光定量系统进行检测定量,之后按照每个样本的测序量要求,进行相应比例的混合。
4)MiSeq PE文库构建:通过PCR将Illumina测序接头序列添加至目标区域外端;使用凝胶回收试剂盒切胶回收PCR产物;Tris-HCl缓冲液洗脱,2%琼脂糖凝胶电泳检测;氢氧化钠变性,产生单链DNA片段。
5)MiSeq测序:DNA片段的一端与引物碱基互补,固定在芯片上;以DNA片段为模板,芯片上固定的碱基序列为引物进行PCR合成,在芯片上合成目标待测DNA片段;变性、退火后,芯片上DNA片段的另一端随机与附近的另外一个引物互补,也被固定住,形成“桥(bridge)”;PCR扩增,产生DNA簇;DNA扩增子线性化成为单链。加入改造过的DNA聚合酶和带有4 种荧光标记的dNTP,每次循环只合成1 个碱基;用激光扫描反应板表面,读取每条模板序列第1轮反应所聚合上去的核苷酸种类;将“荧光基团”和“终止基团”化学切割,恢复3’端黏性,继续聚合第2个核苷酸;统计每轮收集到的荧光信号结果,获知模板DNA片段的序列。
1.4 数据处理
微生物多样性数据分析:MiSeq测序得到的PE Reads首先根据Overlap关系采用Flash软件进行拼接,同时利用QIIME软件对序列质量进行质控和过滤[24],区分样本后进行操作分类单元(operational taxonomic units,OTU) 聚类分析和物种分类学分析,基于OTU进行多种多样性指数分析,利用QIIME软件进行α多样性分析[25]。OTU的主坐标分析(principal co-ordinates analysis,PCoA)、样本聚类分析、箱线图、热图及韦恩图等使用R软件绘制。
2 结果与分析
2.1 不同KCl替代组火腿细菌序列分析
通过MiSeq测序,获得用于分析不同样品的序列。运用高通量测序技术检测不同KCl替代组火腿样品细菌群落,由表1可知:在不同KCl替代组火腿表面共得到257777 条16S rRNA序列,其中最大序列数为59288 条,最小序列数为45782 条;火腿内部共得到248102 条16S rRNA序列,其中最大序列数为54066 条,最小序列数为44320 条。序列长度大部分分布在400~500 bp,平均长度420 bp,各样本序列数均在500 条以上,满足分析要求。
表1 不同KCl替代组火腿中细菌序列统计
Table 1 Statistics of bacterial gene sequences from ham with different levels of KCl substitution
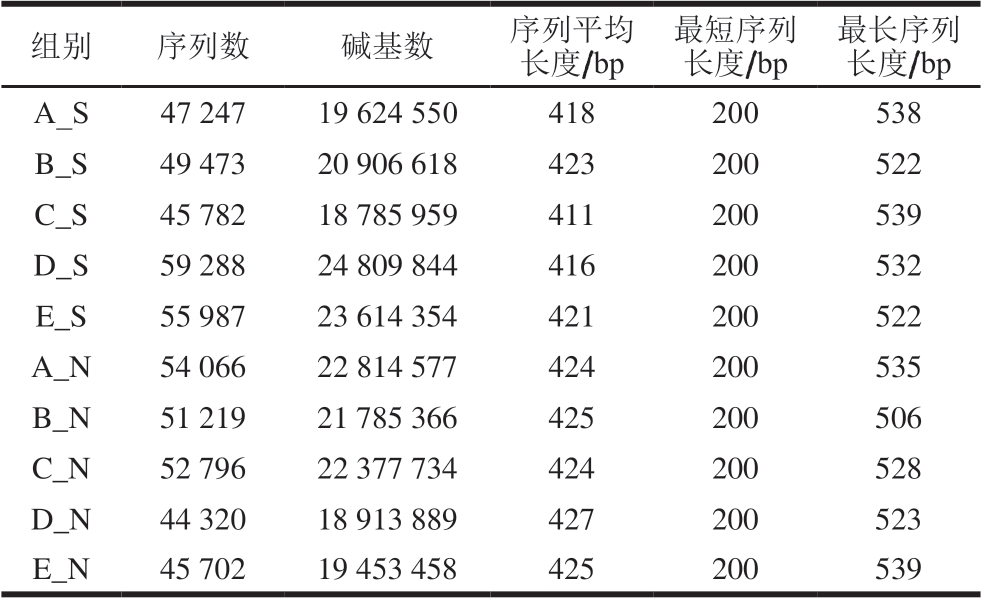
组别 序列数 碱基数 序列平均长度/bp最短序列长度/bp最长序列长度/bp A_S 4724719624550418200538 B_S 4947320906618423200522 C_S 4578218785959411200539 D_S 5928824809844416200532 E_S 5598723614354421200522 A_N 5406622814577424200535 B_N 5121921785366425200506 C_N 5279622377734424200528 D_N 4432018913889427200523 E_N 4570219453458425200539
2.2 不同KCl替代组火腿OTU分析
OTU是在系统发生学或群体遗传学研究中,为了便于进行分析,人为给某一个分类单元(品系、属、种、分组等)设置的统一标志。根据不同的相似度水平,对所有序列进行OTU划分,对97%相似水平下的OTU进行生物信息统计分析。
由表2可知:火腿表面样品在序列相似度97%基础上分类出6153 个OTU,门水平47 个、纲水平117 个、目水平323 个、科水平562 个、属水平1168 个、种水平2259 个;火腿样品内部分类出5704 个OTU,门水平48 个、纲水平114 个、目水平310 个、科水平546 个、属水平1140 个、种水平2165 个。除门水平内部略高于表面外,其余分类水平OTU数表面均高于内部。利用韦恩图研究不同KCl替代组火腿表面和内部细菌群落结构关系,可以直观表现出不同KCl替代组火腿表面和内部细菌OTU数组成的相似性及其重叠情况。由图1A可知,A_S、B_S、C_S、D_S和E_S组样品共有OTU数量171 个,占总序列数的2.78%,其中对照组样品独有的OTU数量最高,为2871 个,其次是50% KCl替代组,为947 个,40% KCl替代组771 个,30% KCl替代组190 个,而60% KCl替代组火腿样品独有的OTU数量最少,为126 个,说明本研究中对照组火腿样品表面细菌群落结构最丰富,而60% KCl替代组火腿样品表面细菌群落结构多样性最小。对于火腿内部,由图1B可知,不同处理组火腿样品共有OTU数量214 个,占总序列数的3.75%,其中,30% KCl替代组火腿样品独有的OTU数量最高,为2659 个,50% KCl替代组火腿样品独有的OTU数量最少,为176 个,说明30% KCl替代组火腿样品内部细菌群落结构最丰富,50% KCl替代组火腿样品内部细菌群落结构多样性最小。
表2 OTU的分类学信息统计
Table 2 Statistics of taxonomic information of OTU

分类 表面(S) 内部(N)门4748纲117114目323310科562546属11681140种22592165 OUT 61535704
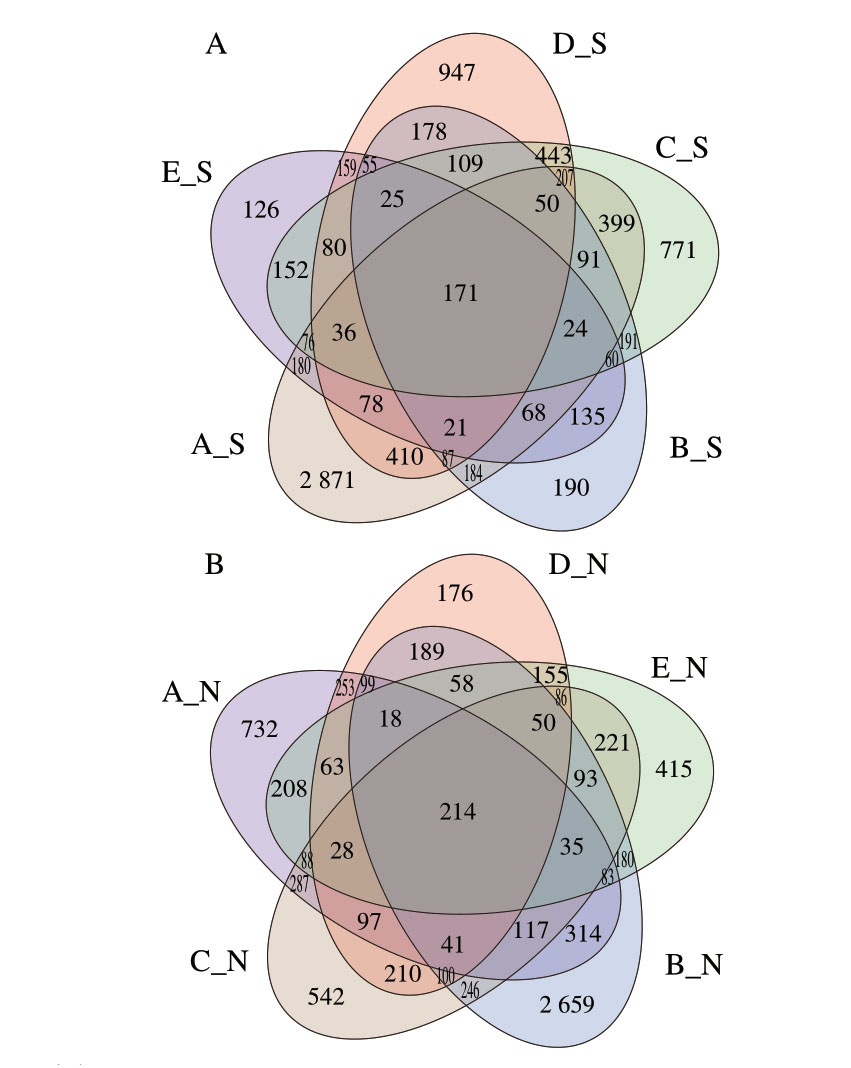
图1 火腿表面(A)和内部(B)细菌OTU分布韦恩图
Fig. 1 Venn diagrams for bacterial OTU distribution on the surface (A) and inside (B) of ham
2.3 稀释曲线分析
稀释曲线是利用各样本在不同测序深度时的微生物α多样性指数构建的曲线,以此反映各样本在不同测序数量时的微生物多样性,它可以用来比较测序数据量不同的样本中物种的丰富度、均一性或多样性,也可以用来说明样本的测序数据量是否合理。由图2可知,随着测序量的扩大,多样性指数增加的趋势逐渐变慢,并且最终均达到饱和状态,说明测序数据量合理,更多的数据量检测只会产生少量新的OTU。并且每个样本的覆盖率均大于99%,这进一步说明实验的测序量足够大,可以覆盖所有样品中的大多数微生物。
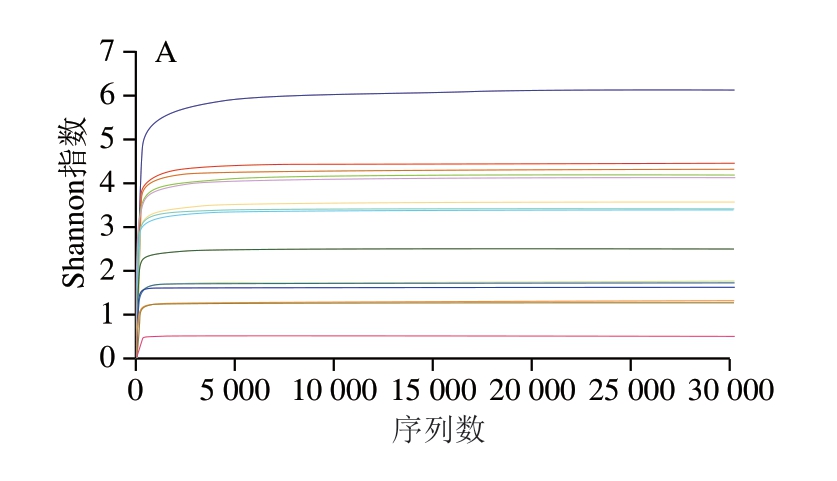
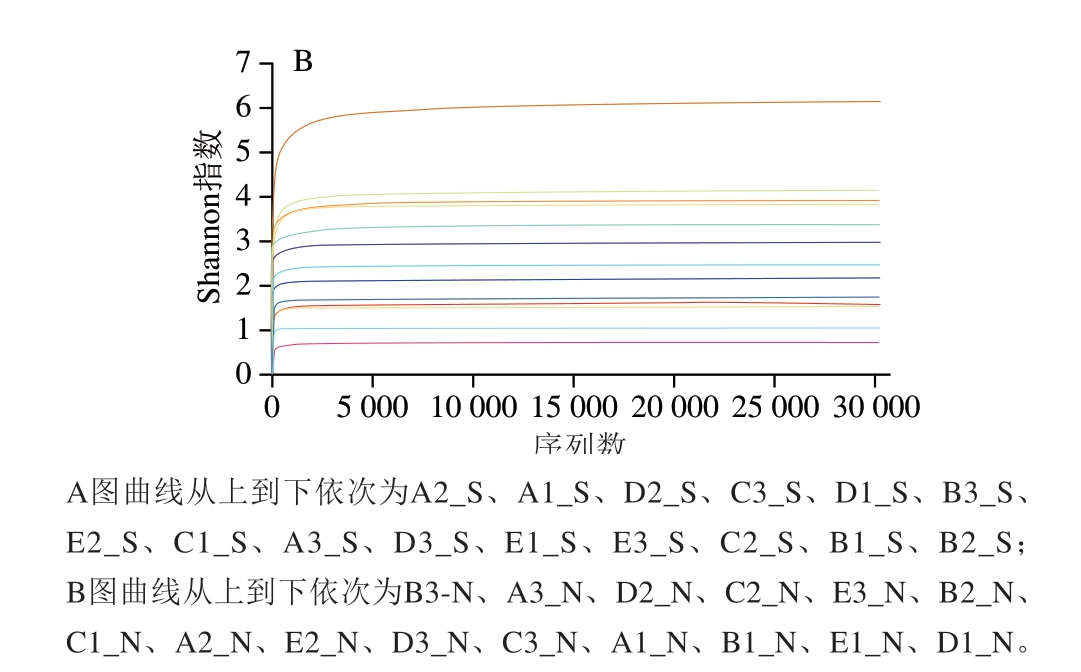
图2 火腿表面(A)和内部(B)细菌的Shannon-Wiener曲线
Fig. 2 Shannon-Wiener curves for bacterial communities on the surface (A) and inside (B) of ham
2.4 基于OTU的α多样性分析
α多样性分析能反映微生物群落中物种的丰富度、覆盖度和多样性等信息。常用的度量标准有Chao 1指数、Shannon指数、Ace指数、Simpson指数、覆盖率等。Ace指数、Chao 1指数用于反映群落丰富度,二者越大则群落丰富度越高。Simpson指数和Shannon指数反映菌群多样性,Shannon指数越大,群落多样性越高,Simpson指数越大,群落多样性越低。覆盖率用于分析样品的测序覆盖率,该指数越高越能反映样本的真实情况。
所有实验样品的覆盖率均为99%,表明本次实验建立的文库能够较准确地反映待测样品的微生物多样性情况。由图3可知,表面样本中,A组火腿样本Ace和Chao 1指数均高于其他4 个KCl替代组,说明KCl部分替代后宣威火腿表层的菌群丰度有所降低。C组与B组和E组的Ace指数存在显著差异(P<0.05),D组与B组和E组的Chao 1指数存在显著差异(P<0.05)。从Shannon指数与Simpson指数均可以看出,A组表面的细菌群落多样性最高,KCl的替代在一定程度上降低了火腿表面群落的多样性,但各组间群落多样性无显著差异。对于火腿内部样品,B组的Ace指数、Chao 1指数与Shannon指数均为组间最高,且Simpson指数最低,说明B组样品内部的菌群丰富度与多样性较好。同时由图3B可知,5 组样本的Ace指数、Chao 1指数、Shannon指数与Simpson指数组间无显著性差异。
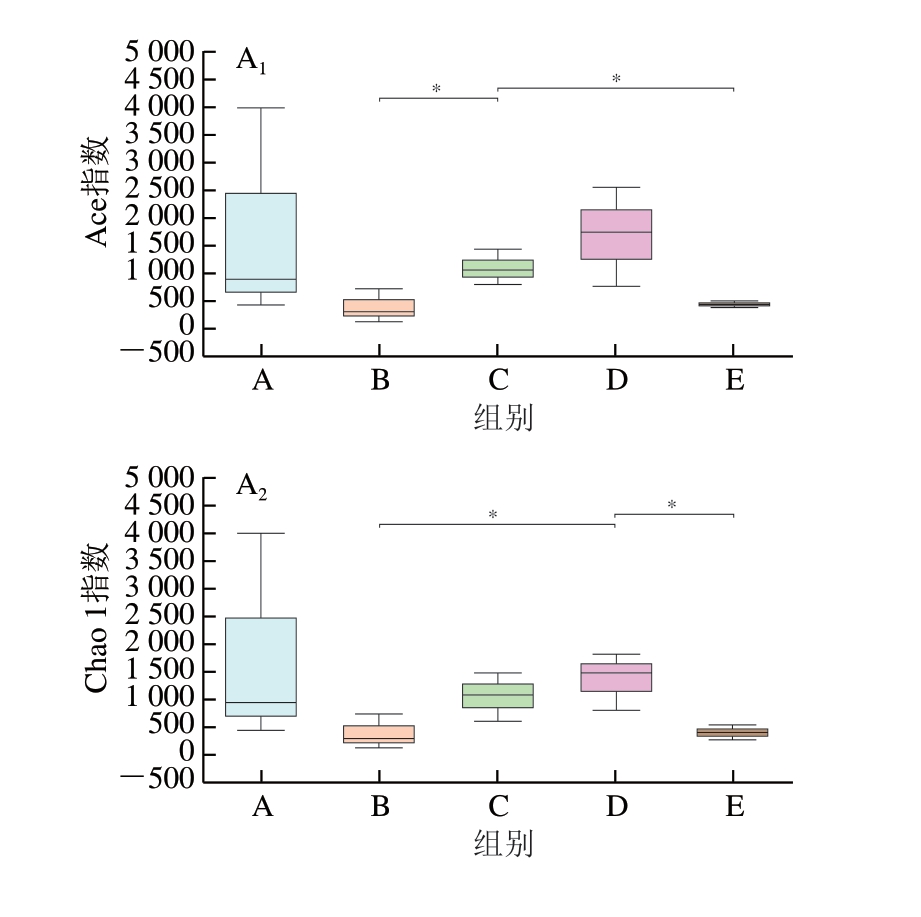
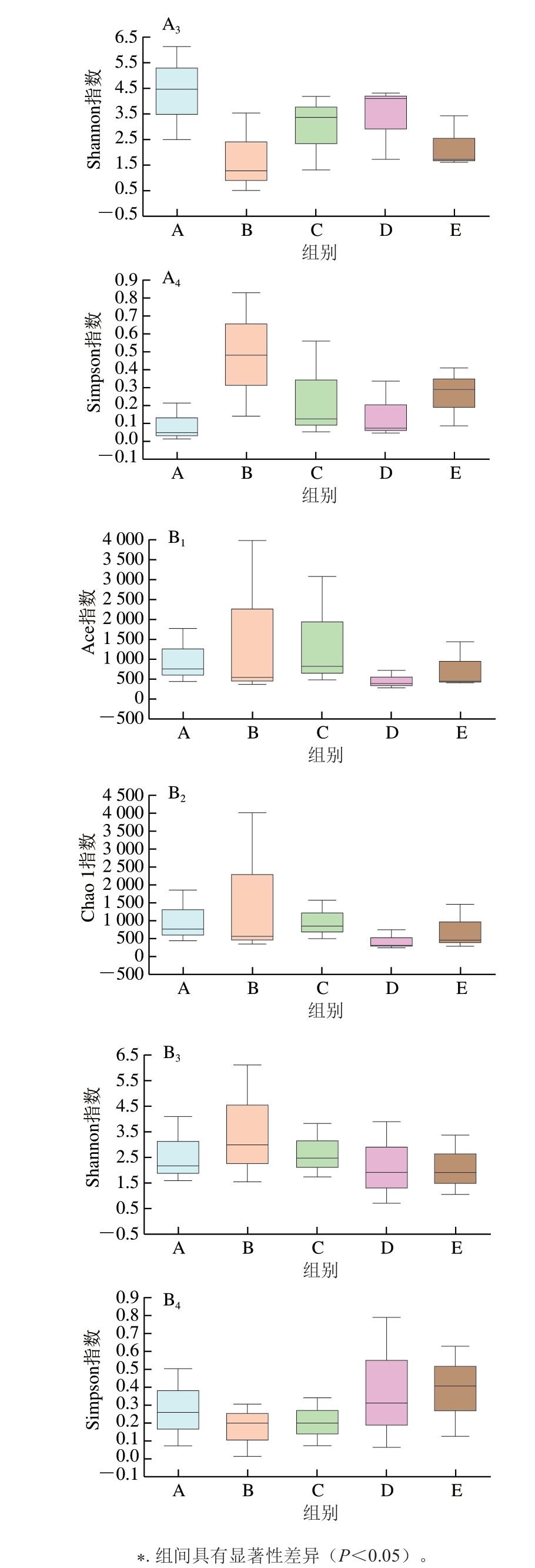
图3 火腿表面(A)与内部(B)样本α多样性指数组间差异检验直方图
Fig. 3 Analysis of inter-group differences in α diversity indexes of surface (A) and internal (B) bacterial communities
2.5 不同KCl替代组火腿群落结构及丰度差异性分析
2.5.1 样本在门水平上的物种分布
根据OTU结果对不同处理组火腿表面样品中的细菌OTU进行整理,进而研究不同KCl替代量火腿样本中的细菌菌落组成。由图4可知,不同处理组火腿表面样品被检出的细菌门主要有变形菌门(Proteobacteria)、厚壁菌门(Firmicutes)、放线菌门(Actinobacteria)、拟杆菌门(Bacteroidetes)、蓝藻菌门(Cyanobacteria)、酸杆菌门(Acidobacteria)、绿弯菌门(Chloroflexi)和迷踪菌门(Elusimicrobia)等。其中,变形菌门和厚壁菌门在不同处理组的样本中占多数,是优势菌门,但是二者在不同处理组的相对丰度不同。A(对照组)、B(30%)、C(40%)、D(50%)和E(60%)组样品中变形菌门相对丰度分别为44.06%、45.45%、72.57%、56.69%和56.54%,而厚壁菌门相对丰度分别为28.28%、37.54%、9.06%、15.44%和24.64%。KCl的替代使变形菌门的相对丰度增加,在40% KCl替代组中最高,对其他处理组影响较小,而厚壁菌门则在40% KCl替代组中最低。由图5可知,不同处理组火腿内部样品中被检出的细菌门主要有变形菌门、厚壁菌门、拟杆菌门、放线菌门、酸杆菌门、绿弯菌门等。相比于表面样本,内部样本门水平菌群种类减少,但变形菌门和厚壁菌门仍然是不同处理组样本中相对丰度较高的优势菌门。变形菌门在A(对照组)、B(30%)、C(40%)、D(50%)和E组(60%)样品中的相对丰度分别为34.67%、49.97%、55.75%、39.43%和39.04%,与表面菌群结构相似,KCl的替代使变形菌门的相对丰度增加,均在40% KCl替代组中最高。而厚壁菌门的相对丰度分别为42.91%、21.42%、31.36%、52.16%和51.05%。当KCl替代量低于40%,厚壁菌门的相对丰度低于对照组,50%~60% KCl替代组间厚壁菌门无明显差别。
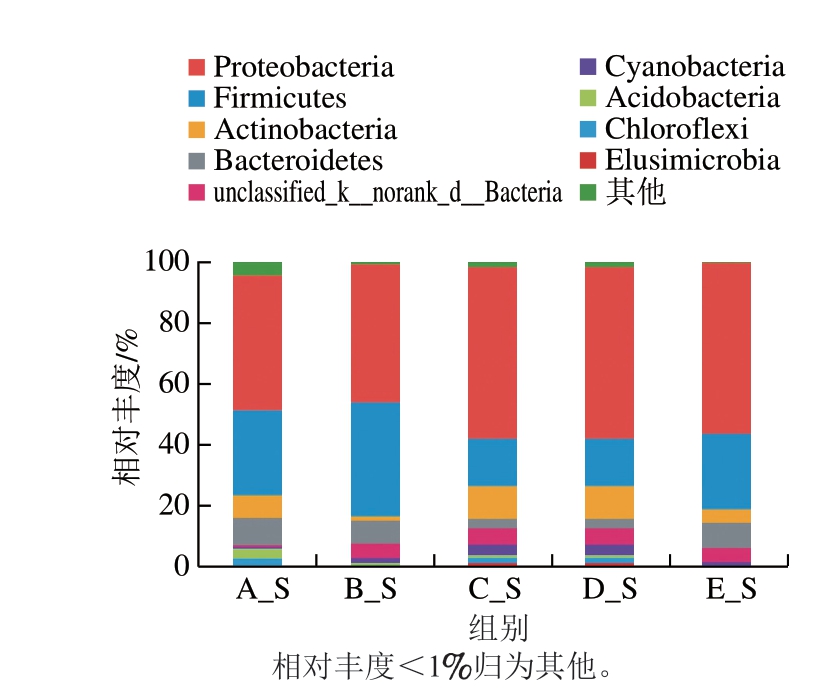
图4 各组火腿表面样本在门水平的菌群分布
Fig. 4 Phylum-level distribution of surface bacterial community
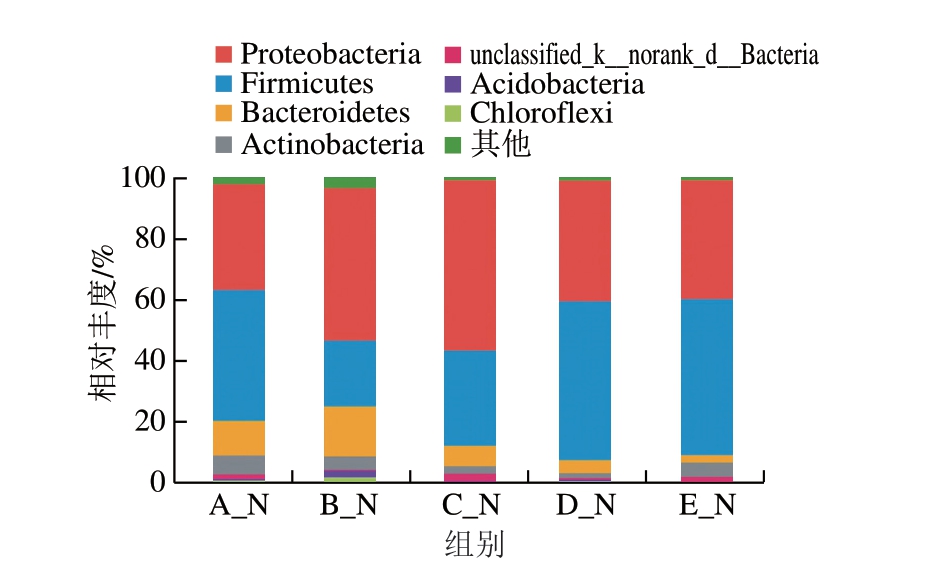
图5 各组火腿内部样本在门水平的菌群分布
Fig. 5 Phylum-level distribution of internal bacterial community
2.5.2 样本在属水平上的物种分布
热图可以通过颜色的相似性及差异性直观比较分析不同样本在各种菌群上的差异。在属水平上,选择丰度前20的物种进行热图分析,从物种和样品2 个层面进行聚类。由图6可知,葡萄球菌属(Staphylococcus)是火腿中属水平占比较多的菌群,其次分别为盐弧菌属(Salinivibrio)和青枯菌属(Ralstonia)。但KCl的替代对葡萄球菌属、盐弧菌属和青枯菌属相对丰度的影响较小。其中,葡萄球菌属在B组(30%)中占比最高,为58.07%。此外,KCl的替代使盐弧菌属的丰度增加,但影响较小。由图7可知,葡萄球菌属是火腿内部属水平占比较多的菌群,葡萄球菌属在A(对照组)、B(30%)、 C(40%)、D(50%)和E组(60%)样品中相对丰度分别为28.37%、18.03%、37.09%、36.25%和31.10%。KCl的替代使盐弧菌属相对丰度明显增加。此外,火腿内部其他占比较大的微生物还有沙雷氏菌属(Serratia)、青枯菌属等,但KCl部分替代NaCl对沙雷氏菌属、青枯菌属无明显影响。
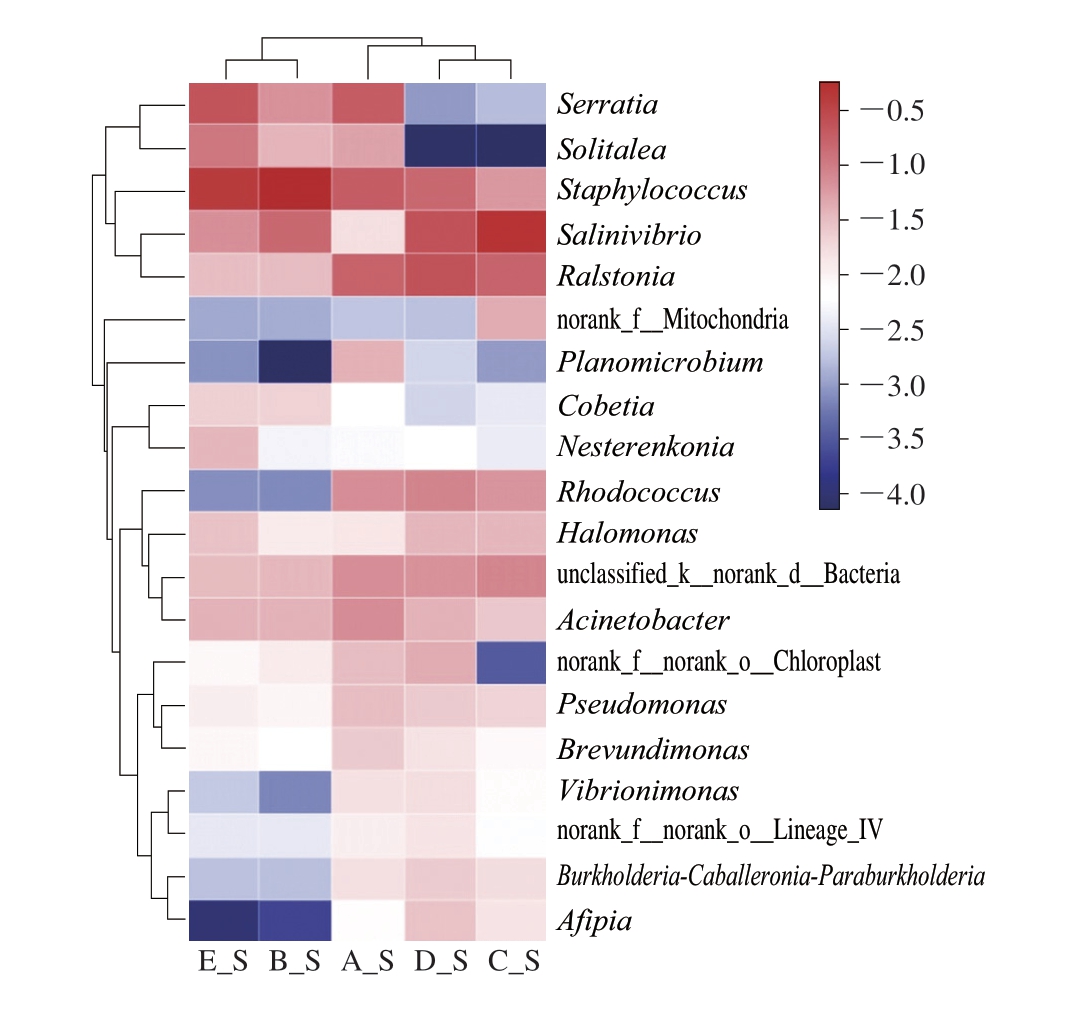
图6 火腿表面细菌属水平群落结构热图
Fig. 6 Heatmap of bacterial community structure at the genus level on the surface of ham
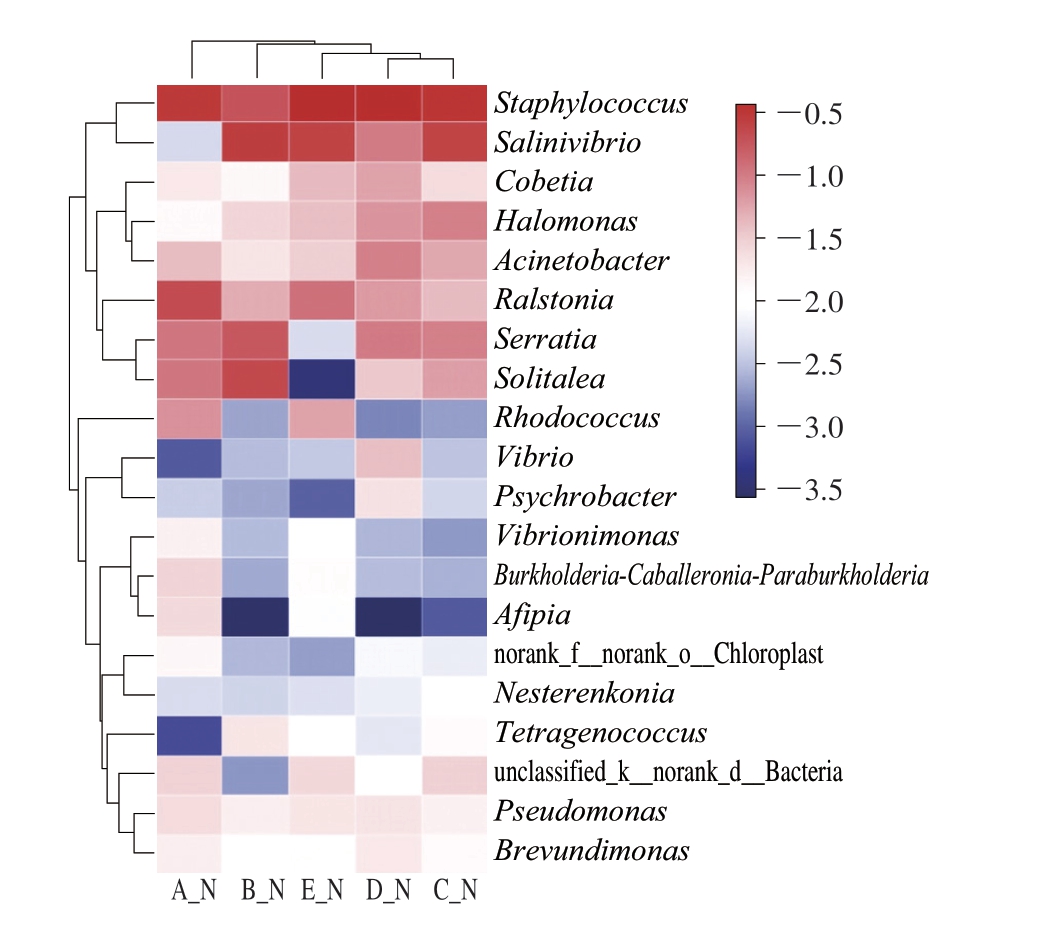
图7 火腿内部细菌属水平群落结构热图
Fig. 7 Heatmap of bacterial community structure at the genus level inside ham
2.6 样本组间PCoA
由图8可知,不同KCl替代组火腿表面PC1和PC2分别解释了细菌群落46.71%和28.70%的信息,不同替代组火腿内部PC1和PC2分别解释了细菌群落51.16%和19.67%的信息,无论是火腿表面还是内部,2 个主成分之和均大于70%,表明2 个主成分较好代表了样品中细菌群落信息。火腿表面和内部各处理组样品有交叉,说明不同KCl替代组细菌存在较大相似性。
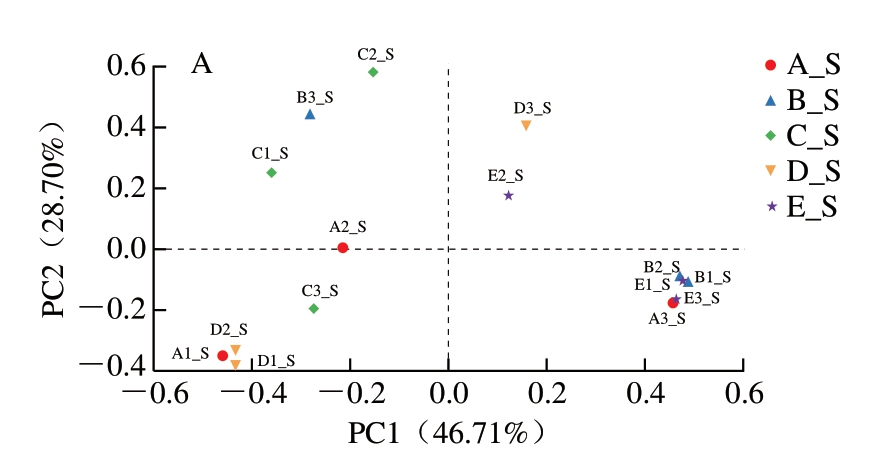
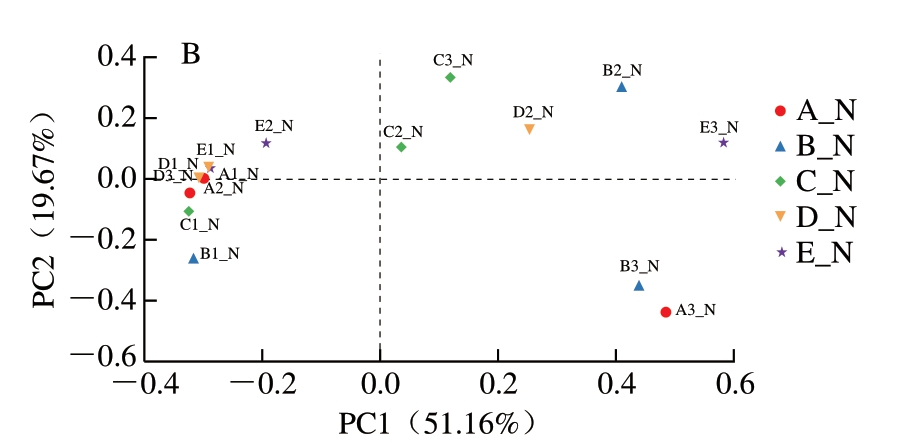
图8 不同KCl替代组火腿表面(A)和内部(B)细菌PCoA图
Fig. 8 PCoA plots for bacteria communities on the surface (A) and inside (B) of ham with different levels of KCl substitution
3 讨论
宣威火腿属于发酵肉制品,火腿成熟过程中微生物在风味的形成和安全方面发挥着重要作用。火腿中微生物种类丰富多样,不同微生物对宣威火腿品质的影响各异。宣威火腿中主要有细菌、酵母菌、霉菌等多种微生物,这些微生物可能来源于猪肉本身、食盐等发酵剂,或者来源于工具、设备、加工环境[26],因此不同的盐分和水分活度等会影响火腿中微生物的种类和含量[13]。本研究结果表明,宣威火腿微生物组成丰富,不同KCl替代组火腿的微生物群落存在一定差异,但葡萄球菌属仍然是不同KCl替代组火腿的优势菌群,这与邹颖玲等[27]的研究结果相一致。葡萄球菌可以促进脂肪和蛋白质降解,从而有助于挥发性风味化合物的生物合成。同时在发酵过程中,葡萄球菌具有硝酸盐还原酶活性,可将硝酸根还原成亚硝酸根,亚硝酸根会分解产生一氧化氮,再与肌红蛋白结合生成亚硝基肌红蛋白,对火腿的颜色也发挥了重要作用[28-30]。
李平兰等[15]通过采用选择性培养基对宣威火腿成熟产品中的主要微生物菌相进行分离和计数,结果表明,葡萄球菌、微球菌和霉菌为宣威火腿的优势菌群。黄艾祥[31]对云南干腌火腿品质特征形成与微生物的作用进行研究,发现葡萄球菌为云腿加工过程中的优势菌,这与本研究结果较为一致,葡萄球菌均是宣威火腿的优势菌,且在不同KCl替代组火腿中葡萄球菌属的相对丰度无显著差异,说明KCl替代对宣威火腿优势菌群影响较小,可能是由于KCl与NaCl的化学性质较为相似。KCl的替代使宣威火腿表层和内部变形菌门的相对丰度增加,均在40% KCl替代组中最高,可能是由于KCl代替NaCl可以增加耐盐菌群的数量[32]。
李欣蔚等[33]对剑门火腿的研究表明,火腿内部的主要微生物为乳酸菌,这与本研究存在一定的差异,可能是由于不同的发酵条件及不同的发酵阶段会有不同的优势菌群。除了优势菌群外,还含有少量丰度较低的微生物群落,在不同的KCl替代组中均检测出部分不可分类微生物,这是由于测序结果与数据库中的序列不匹配或相似度低于阈值,因此这些序列被归为不可分类的微生物,这些微生物对火腿的作用机理尚不明确,有待进一步研究。
4 结论
KCl部分替代NaCl在一定程度上降低了宣威火腿表层菌群丰度和群落多样性,对火腿内部菌群丰度和群落多样性则无显著影响。各KCl替代组宣威火腿表层和内部样本在门水平上的优势菌群为变形菌门和厚壁菌门,KCl的替代使宣威火腿表层和内部变形菌门的相对丰度增加。而在属水平上,葡萄球菌属是各替代组宣威火腿表层及内部占比最大的菌群,属于宣威火腿的优势菌属,不同比例KCl替代组宣威火腿葡萄球菌属的相对丰度无显著差异。不同组之间微生物群落结构并无明显区分,因此不同KCl替代组宣威火腿表面和内部细菌存在较大相似性。
[1] 廖国周, 王桂瑛, 程志斌, 等. 双向电泳分析宣威火腿蛋白质组方法的建立[J]. 食品科学, 2012, 33(23): 125-128.
[2] 陈明, 王正祥, 徐宝才. 宣威火腿生产气候变化的研究[J]. 肉类工业, 2009(1): 21-24.
[3] 乔发东, 马长伟. 传统宣威火腿品质特征及成因分析[J]. 食品科学, 2004, 25(8): 55-61.
[4] 郭秀云, 张雅玮, 彭增起. 食盐减控研究进展[J]. 食品科学, 2012, 33(21): 374-378.
[5] KLOSS L, MEYER J D, GRAEVE L, et al. Sodium intake and its reduction by food reformulation in the European Union: a review[J]. NFS Journal, 2015, 1(30): 9-19. DOI:10.1016/j.nfs.2015.03.001.
[6] COSTA-CORREDOR A, SERRA X, ARNAU J, et al. Reduction of NaCl content in restructured dry-cured hams: post-resting temperature and drying level effects on physicochemical and sensory parameters[J]. Meat Science, 2009, 83(3): 390-397. DOI:10.1016/j.meatsci.2009.06.011.
[7] 张东, 李洪军, 吴练军, 等. 减少肉制品中氯化钠含量的研究进展[J]. 食品与发酵工业, 2017, 43(11): 238-243. DOI:10.13995/j.cnki.11-1802/ts.014561.
[8] 周光宏, 李春保, 徐幸莲. 肉类食用品质评价方法研究进展[J]. 中国科技论文在线, 2007, 2(2): 75-82.
[9] 孔庆新, 张径舟, 黄业传, 等. KCl部分替代NaCl对猪肉脂肪氧化的动力学影响[J]. 食品工业科技, 2022, 43(5): 98-104. DOI:10.13386/j.issn1002-0306.2021060033.
[10] MARKLUND M, SINGH G, GREER R, et al. Estimated population wide benefits and risks in China of lowering sodium through potassium enriched salt substitution: modelling study[J]. The British Medical Journal, 2020, 369: 1-14. DOI:10.1136/bmj.m824.
[11] ZHAO Fan, ZHOU Guanghong, YE Keping, et al. Microbial changes in vacuum-packed chilled pork during storage[J]. Meat Science, 2015, 100: 145-149. DOI:10.1016/j.meatsci.2014.10.004.
[12] ZHOU Guanghong, ZHAO Gaiming. Biochemical changes during processing of traditional Jinhua ham[J]. Meat Science, 2007, 77: 114-120. DOI:10.1016/j.meatsci.2007.03.028.
[13] 王桥美, 杨瑞娟, 严亮. 微生物多样性与宣威火腿品质关系的研究进展[J]. 食品安全导刊, 2016(33): 137-139. DOI:10.16043/j.cnki.cfs.2016.33.102.
[14] PETROVA I, AASEN I M, RUSTAD T, et al. Manufacture of drycured ham: a review. Part 1. Biochemical changes during the technological process[J]. European Food Research and Technology, 2015, 241: 587-599. DOI:10.1007/s00217-015-2490-2.
[15] 李平兰, 沈清武, 吕燕妮, 等. 宣威火腿成熟产品中主要微生物菌相构成分析[J]. 中国微生态学杂志, 2003(5): 12-13. DOI:10.13381/j.cnki.cjm.2003.05.007.
[16] WANG Xinghong, PING Ma, JIANG Dongfu, et al. The natural microflora of Xuanwei ham and the no-mouldy ham production[J]. Journal of Food Engineering, 2006, 77(1): 103-111. DOI:10.1016/j.jfoodeng.2005.06.047.
[17] 邹颖玲, 刘姝韵, 王桂瑛, 等. 基于PCR-DGGE技术分析宣威火腿中真菌群落结构[J]. 食品与发酵工业, 2020, 46(6): 269-274. DOI:10.13995/j.cnki.11-1802/ts.022394.
[18] WANG Xinhui, WANG Songhu, ZHAO Hai. Unraveling microbial community diversity and succession of Chinese Sichuan sausages during spontaneous fermentation by high-throughput sequencing[J]. Journal of Food Science and Technology, 2019, 56: 3254-3263. DOI:10.1007/s13197-019-03781-y.
[19] LIN Fengke, CAI Fei, LUO Binsheng, et al. Variation of microbiological and biochemical profiles of Laowo dry-cured ham, an indigenous fermented food, during ripening by GC-TOF-MS and UPLC-QTOF-MS[J]. Journal of Agricultural and Food Chemistry, 2020, 68(33): 8925-8935. DOI:10.1021/acs.jafc.0c03254.
[20] PHEWPAN A, PHUWAPRISIRISAN P, TAKAHASHI H, et al. Investigation of kokumi substances and bacteria in Thai fermented freshwater fish (Pla-ra)[J]. Journal of Agricultural and Food Chemistry, 2020, 68(38): 10345-10351. DOI:10.1021/acs.jafc.9b06107.
[21] DICKINSON B D, HAVAS S. Reducing the population burden of cardiovascular disease by reducing sodium intake: a report of the Council on Science and Public[J]. HealthArchives of Internal Medicine, 2007, 167(14): 1460-1468. DOI:10.1001/archinte.167.14.1460.
[22] 吴海舟, 张迎阳, 黎良浩, 等. KCl部分替代NaCl腌制对干腌肉制品蛋白质水解和感官品质的影响[J]. 食品科学, 2014, 35(1): 39-43.
[23] 包媛媛, 林奇, 黄艾祥. 传统宣威火腿的加工工艺[J]. 肉类工业, 2007(7): 16-18.
[24] MAGOČ T, SALZBERG S L. FLASH: fast length adjustment of short reads to improve genome assemblies[J]. Bioinformatics, 2011, 27(21): 2957-2963. DOI:10.1093/bioinformatics/btr507.
[25] CAPORASO J G, KUCZYNSKI J, STOMBAUGH J, et al. QIIME allows analysis of high-throughput community sequencing data[J]. Nature Methods, 2010, 7(5): 335-336. DOI:10.1038/nmeth.f.303.
[26] 鞠铭, 刘鑫, 李官浩, 等. 干腌火腿微生物研究进展[J]. 食品与机械, 2020, 36(9): 217-222. DOI:10.13652/j.issn.1003-5788.2020.09.039.
[27] 邹颖玲, 刘姝韵, 王桂瑛, 等. 基于PCR-DGGE分析宣威火腿的细菌群落结构[J]. 食品研究与开发, 2020, 41(23): 192-198.
[28] STAVROPOULOU D A, VAN RECKEM E, DE SMET S, et al. The narrowing down of inoculated communities of coagulase-negative staphylococci in fermented meat models is modulated by temperature and pH[J]. International Journal of Food Microbiology, 2018, 274: 52-59. DOI:10.1016/j.ijfoodmicro.2018.03.008.
[29] BOSSE R, GIBIS M, SCHMIDT H, et al. Nitrate reductase activity of Staphylococcus carnosus affecting the color formation in cured raw ham[J]. Food Research International, 2016, 85: 113-120. DOI:10.1016/j.foodres.2016.04.021.
[30] 郭明亮. 火腿菌群分析及优良菌株对发酵香肠中菌群构成的影响[D]. 扬州: 扬州大学, 2015: 10-15.
[31] 黄艾祥. 云南干腌火腿品质特征形成与微生物作用研究[D]. 重庆: 西南大学, 2006: 72-75.
[32] LORENZO J M, BERMÚDEZ R, DOMÍNGUEZ R, et al. Physicochemical and microbial changes during the manufacturing process of dry-cured lacón salted with potassium, calcium and magnesium chloride as a partialreplacement for sodium chloride[J]. Food Control, 2015, 50: 763-769. DOI:10.1016/j.foodcont.2014.10.019.
[33] 李欣蔚, 迟原龙, 贾冬英, 等. 剑门火腿菌相构成及主要腐败菌特性分析[J]. 中国酿造, 2012, 31(11): 132-134.