狐、貂与狗同属犬型总科动物,携带多种病毒,贸然食用易对食用者身体健康造成潜在威胁[1-2]。新型冠状病毒肺炎的全球性肆虐,也在警醒食用野生动物的危险性[3-5]。为更好避免潜在的病毒威胁,我国对此作出了一系列举措。2020年2月24日,全国人大委员会通过了关于革除滥食野生动物陋习的相关决定。同年3月31日,深圳市正式立法,狗肉缺席“白名单”[6],狐、貂仅作为药用。因此,亟需建立一种快速准确检测狗、狐、貂源性成分的方法,为立法后的监管提供便利。
肉质成分检测多以形态学[7]、代谢学[8-9]、蛋白质学[10-11]和基因学差异[12-14]作为切入点。目前,分子生物学技术是肉及制品检测的常用技术[15]。近年来,频频爆出肉类掺假的新闻,肉类质量安全监管变得愈加迫切。史莹莹等[1 6]应用实时荧光定量聚合酶链式反应(polymerase chain reaction,PCR)法鉴定肉制品中牛源性成分。Li Tingting等[17]建立了一种基于对照引物的实时荧光定量PCR方法,用于猪肉中掺假山羊肉的定量检测。张秀平等[18]应用微滴数字PCR定量检测鸭源性成分。以上方法可以完成定量检测,但对于快速定性检测无疑增加了成本。多重PCR方法可以同时检测多个核酸片段,适合肉制品中动物源性成分鉴定[19],利于市场监管,有更广泛的市场应用价值。Li Xinnan等[20]应用多重PCR技术建立三重、四重PCR体系,检测牛羊肉中的狐、貂及浣熊源性成分。Qin Panzhu等[21]基于侧向流动条带平台及PCR准确鉴别掺假鸭肉。Liu Wanwan等[22]建立基于通用引物的多重PCR检测系统,确定羊肉中是否含有来自大鼠、狐狸和鸭子的非突变成分。Xu Rusu等[23]建立多重TaqMan锁定核酸实时PCR检测方法,用于同时检测鸭肉、猪肉、牛肉和鸡肉。任易婕等[24]基于多重PCR与膜芯片技术实现对牛、羊、牦牛及驴源性肉类食品的鉴别及检测。本研究从监管角度出发,基于多重PCR技术,开发3 种常见犬型总科动物(狗、狐、貂)DNA成分检测试剂盒,旨在为市场监管提供有利技术支持。
1 材料与方法
1.1 材料与试剂
标准品肉样(狗肉、狐肉、貂肉)和其他检测肉样(猪肉、牛肉、羊肉、马肉、驴肉、鸡肉、鸭肉)由长春食品药品检测中心提供,模拟混合肉样由标准品肉样按质量比混合而成,如表1所示。市售肉样分别购自农贸市场、狗肉馆及线上销售。
表1 样品模拟混合方式
Table 1 Composition of binary and ternary meat mixtures
组织/细胞/血液基因组DNA提取试剂盒、大肠杆菌DH5α感受态细胞、普通琼脂糖凝胶DNA回收试剂盒、pGM-T连接试剂盒、质粒小提试剂盒、100 bp DNA Marker、2×Taq PCR MasterMix 北京天根生化科技有限公司;其他试剂均为国产分析纯。
1.2 仪器与设备
TC9600-G多功能梯度PCR仪 美国Labnet公司;JY300E通用型电泳仪 北京君意东方电泳设备有限公司;UV WHITE-2020D紫外凝胶成像分析仪 美国Bio-Rad公司;NanoDrop One微量核酸蛋白测定仪 美国Thermo公司。
1.3 方法
1.3.1 肉样DNA提取
称取待测样品约0.05 g,分别置于洁净EP管中,0.9 g/100 mL NaCl溶液浸泡、冲洗,去血水;每管加入150 μL 0.9 g/100 mL NaCl,匀浆器处理为细胞悬液,12 000 r/min离心1 min;弃上清,依次加入P1溶液500 μL、P2溶液30 μL、P3溶液15 μL,颠倒混匀,56 ℃水浴振荡1 h(每15 min颠倒混匀1 次,至溶液变清亮即可);加入P4溶液500 μL,充分混匀,12 000 r/min离心5 min;取上清液转移至另一洁净EP管,加入等体积P5溶液,-20 ℃放置5 min,12 000 r/min离心5 min;弃上清液,加入体积分数70%冰冻乙醇溶液500 μL,混匀,12 000 r/min离心5 min,弃上清液(重复该步骤1 次);室温晾干后(约15 min即可),加入P6溶液50 μL溶解DNA(可二次洗脱),即得肉样DNA溶液。
P1溶液:1 mol/L Tris-HCl、0.5 mol/L乙二胺四乙酸、0.9 g/100 mL NaCl;P2溶液:10 g/100 mL十二烷基硫酸钠;P3溶液:蛋白酶K;P4溶液:饱和乙酸钠;P5溶液:异丙醇;P6溶液:ddH2O。下同。
1.3.2 引物设计
靶基因序列选取:狗种属基因(序列I D:KU291093.1)、狐狸种属基因(序列ID:LT560065.1)、貂种属基因(序列ID:KU146454.1);采用Primer 5.0软件设计并选取理想种属特异性引物(表2),由生工生物工程(上海)股份有限公司合成。所有引物经NCBI-Nucleotide BLAST预分析PCR产物片段特异性。
表2 引物序列
Table 2 Primer sequences used in this study
1.3.3 多重PCR反应体系的建立
1.3.3.1 多重PCR反应体系
反应体系(30 μL):2×Taq PCR Master Mix 15 μL、Can-481F、R(浓度均为1.55 µmol/L)、Vul-393F、R(2.0 µmol/L)、Mar-289F、R(10.0 µmol/L)、各物种DNA模板(5 ng/μL)0.5 μL,共计1.5 μL,无菌ddH2O补足。
反应参数:94 ℃预变性5 min;94 ℃变性30 s,58 ℃退火45 s,72 ℃延伸55 s,30 个循环;72 ℃延伸10 min,4 ℃保存。
PCR产物检测:1.5%琼脂糖凝胶,85 V/cm电泳85 min。紫外凝胶成像分析仪观察,狗肉、狐肉、貂肉应分别在481、393、289 bp处有单一明亮条带。
1.3.3.2 克隆与测序
紫外灯下,刀片切取特异性目的条带(取自1.3.3.1节电泳凝胶),普通琼脂糖凝胶DNA回收试剂盒回收、纯化;pGM-T连接试剂盒连接产物DNA于pGM-T载体;转化至大肠杆菌DH5α感受态细胞,SOC培养基增菌培养,LB固体培养基(添加5-溴-4-氯-3-吲哚-β-D-半乳糖苷(X-gal)、异丙基-β-D-硫代半乳糖苷(IPTG))蓝白斑筛选阳性克隆;LB液体培养基增菌,质粒小提试剂盒提取质粒DNA;微量核酸蛋白测定仪测定质粒DNA质量浓度。
取1.5 μL质粒DNA原液,分别配制PCR反应体系。每个反应体系含1.5 μL质粒DNA原液、1.5 μL引物混合液(Can-481F、R(1.55 µmol/L)、Vul-393F、R(2.0 µmol/L)、Mar-289F、R(10.0 µmol/L))、15 μL 2×Taq PCR Master Mix、12 μL无菌ddH2O。以最佳反应参数扩增,进行琼脂糖凝胶电泳,紫外凝胶成像分析仪观察结果。
质粒D N A原液经分装密封,送至生工生物工程(上海)股份有限公司测序,NCBI-Nucleotide BLAST分析比对。
1.3.4 犬型总科动物(狗、狐、貂)DNA检测试剂盒组装与评价
1.3.4.1 DNA检测试剂盒组装
DNA提取体系:加入P1、P2、P3溶液破坏细胞膜、核膜,去除蛋白质;加入P4、P5溶液沉淀核酸;最后加入P6溶液溶解DNA。
PCR反应体系:1.5 μL引物混合液(Can-481F、R(1.55 µmol/L)、Vul-393F、R(2.0 µmol/L)、Mar-289F、R(10.0 µmol/L))、15 μL 2×Taq PCR Master Mix、12 μL无菌ddH2O。
标准质粒作为阳性对照,ddH2O作为阴性对照。
1.3.4.2 试剂盒特异性评价
按肉样DNA提取方法分别提取标准样品(狗肉、狐肉、貂肉、猪肉、牛肉、羊肉、马肉、驴肉、鸡肉、鸭肉)DNA,微量核酸蛋白测定仪测定DNA质量浓度及纯度,并稀释至5 ng/μL。制备DNA混合液(含各物种DNA各1 μL),彻底混匀。使用1.3.3.1节反应条件进行PCR扩增,进行1.5%琼脂糖凝胶电泳,紫外凝胶成像分析仪观察结果。
1.3.4.3 试剂盒灵敏度评价
标准品DNA模板(狗/狐/貂)经无菌ddH2O稀释,制备梯度混悬液,质量浓度102~10-5 ng/μL。引物混合液:含1.55 µmol/L Can-481F、R,2.0 µmol/L Vul-393F、R,10.0 µmol/L Mar-289F、R。参照1.3.3.1节配制反应体系,每个反应体系含1.5 μL DNA混合液(102~10-5 ng/μL)、1.5 μL引物混合液、15 μL 2×Taq PCR Master Mix、12 μL无菌ddH2O。使用1.3.3.1节反应条件进行PCR扩增,进行1.5%琼脂糖凝胶电泳,紫外凝胶成像分析仪观察结果。
1.3.5 模拟混合样品检测
考虑实际生活中各掺假情况出现的可能性,制备模拟混合肉样,每种模拟混合肉样制备2 份,与标准品肉样共计30 份样品。随机抽样10 份,编号分别为A1/A3(狗、狐、貂质量比1∶6∶3)、A2(狗、貂质量比9∶1)、A4(狗、狐质量比9∶1)、A5(狗)、B1(狗、狐质量比9∶1)、B2(狗、貂质量比9∶1)、B3(狗)、B4(狗、狐、貂质量比3∶6∶1)、B5(狗、狐质量比1∶9)。提取肉样DNA,微量核酸蛋白测定仪测定D N A质量浓度,稀释至5 ng/μL。以标准品DNA模板(狗/狐/貂)为阳性对照,根据1.3.3.1节多重PCR反应体系、反应参数进行扩增,进行1.5%琼脂糖凝胶电泳,紫外凝胶成像分析仪观察。
1.3.6 市售样品检测
选取农贸市场、狗肉馆及线上销售3 种购买来源,获取50 份市售样品。检测方法同1.3.5节。
1.4 数据处理
采用Primer 5.0软件设计并选取种属特异性引物,所有引物经NCBI-Nucleotide BLAST预分析PCR产物片段特异性。克隆后的序列结果经NCBI-Nucleotide BLAST分析,与GenBank中已登记的序列进行比对验证。
2 结果与分析
2.1 狗、狐、貂DNA检测试剂盒特异性评价
由图1可知,在多种DNA模板(狗肉、狐肉、貂肉、猪肉、牛肉、羊肉、马肉、驴肉、鸡肉、鸭肉)存在情况下,泳道1按1.3.4.1节引物比例加入狗、狐、貂的引物混合液,泳道2、3、4分别加入狗、狐、貂的引物,电泳条带出现情况与相应检测体系中目的条带相符,无非特异性干扰,无交叉干扰,背景干净,条带清晰,狗、狐、貂的引物特异性较好。初步表明本研究所制备的试剂盒特异性良好,常见食用肉猪肉、牛肉、羊肉、马肉、驴肉、鸡肉、鸭肉的DNA成分于本检测体系无非特异性扩增,3 种特异性引物检测肉质成分于本检测体系无交叉干扰。
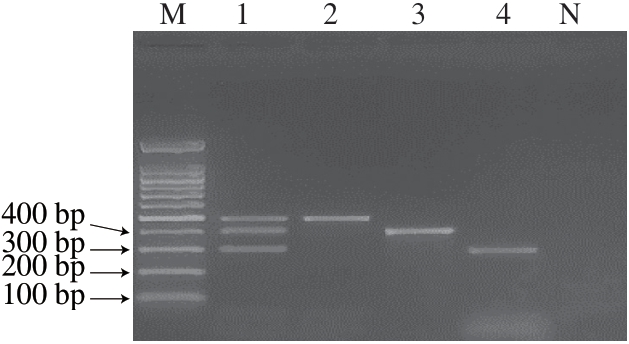
图1 试剂盒特异性评价的琼脂糖凝胶电泳图谱
Fig. 1 Agarose gel electrophoresis patterns showing the kit’s specificity
泳道M. 100 bp DNA Ladder;泳道1. 狗、狐、貂混合物;泳道2. 狗;泳道3. 狐;泳道4. 貂;泳道N. 空白对照。
2.2 狗、狐、貂DNA检测试剂盒灵敏度评价
由图2可知:标准品DNA模板质量浓度处于102~10-4 ng/μL时,可见3 条特异性条带(狗:481 bp,狐:393 bp,貂:289 bp),条带清晰无拖尾,背景干净;标准品DNA模板质量浓度降至10-5 ng/μL时,条带消失;空白对照无非特异性条带。表明本研究所制备的试剂盒灵敏度高,最低检测限为10-4 ng/μL。
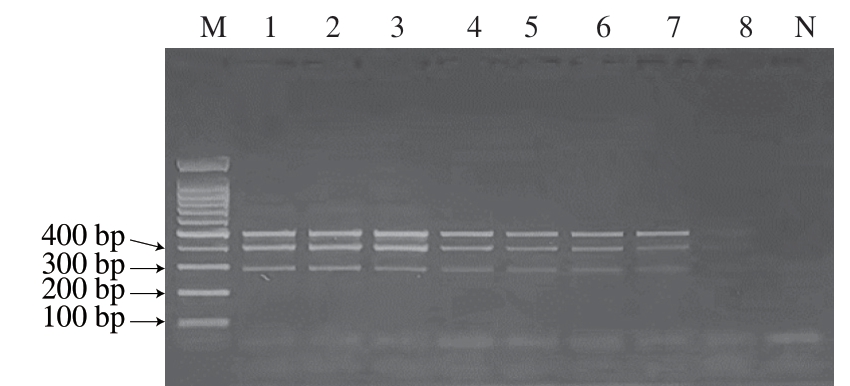
图2 试剂盒灵敏度评价的琼脂糖凝胶电泳图谱
Fig. 2 Agarose gel electrophoresis patterns showing the kit’s sensitivity
泳道M. 100 bp DNA Ladder;泳道1~8. DNA模板质量浓度102、101、100、10-1、10-2、10-3、10-4、10-5 ng/µL;泳道N. 空白对照。
2.3 狗、狐、貂PCR产物克隆与测序

特异性目的条带经切胶回收,连接转化并提取质粒DNA后,进行测序比对。由图3可知,序列经NCBINucleotide BLAST分析,与GenBank中已登记的狗/狐/貂序列相似性为100%,证明本研究所选取靶基因序列可作为种属特异性检测序列,间接证明多重PCR检测体系特异性实验结果的准确性。

图3 狗(A)、狐(B)、貂(C)测序序列比对分析结果
Fig. 3 Results of alignment of target gene sequences with dog (A),fox (B), and mink (C) sequences deposited in GenBank
质粒DNA原液经PCR扩增后,进行琼脂糖凝胶电泳。由图4可知,泳道1~6皆见明亮特异性条带,条带大小与原切取条带大小相符(泳道1~2:481 bp,泳道3~4:393 bp,泳道5~6:289 bp),背景干净无杂带,空白对照无非特异性条带,为测序结果作进一步佐证。
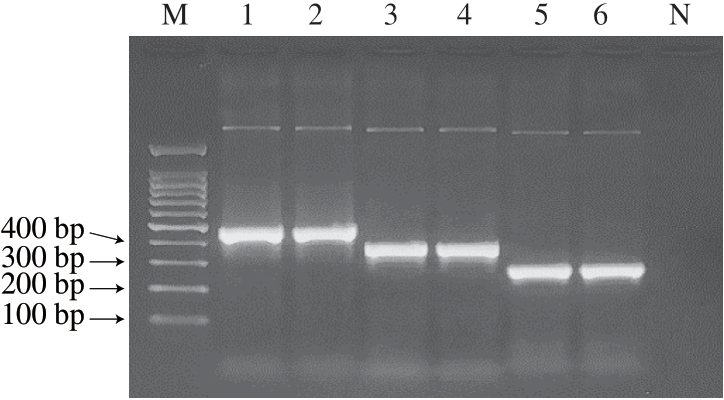
图4 质粒DNA原液PCR产物琼脂糖凝胶电泳图谱
Fig. 4 Agarose gel electrophoresis pattern of PCR products from plasmid DNA stock solution
泳道M. 100 bp DNA Ladder;泳道1~2. 狗;泳道3~4. 狐;泳道5~6. 貂;泳道N. 空白对照。
2.4 狗、狐、貂模拟混合样品检测
10 份随机抽取的模拟混合样品经DNA提取、多重PCR检测体系扩增后,得到琼脂糖凝胶电泳图谱。由图5可知,条带有无及明暗情况与各样品原模拟混合方式大致相符。阳性对照组条带清晰且亮度均一,阴性对照组无非特异性扩增,表明该多重PCR检测体系特异性良好,3 种特异性成分比例变化不影响检测准确性,可满足掺假肉的日常检测。
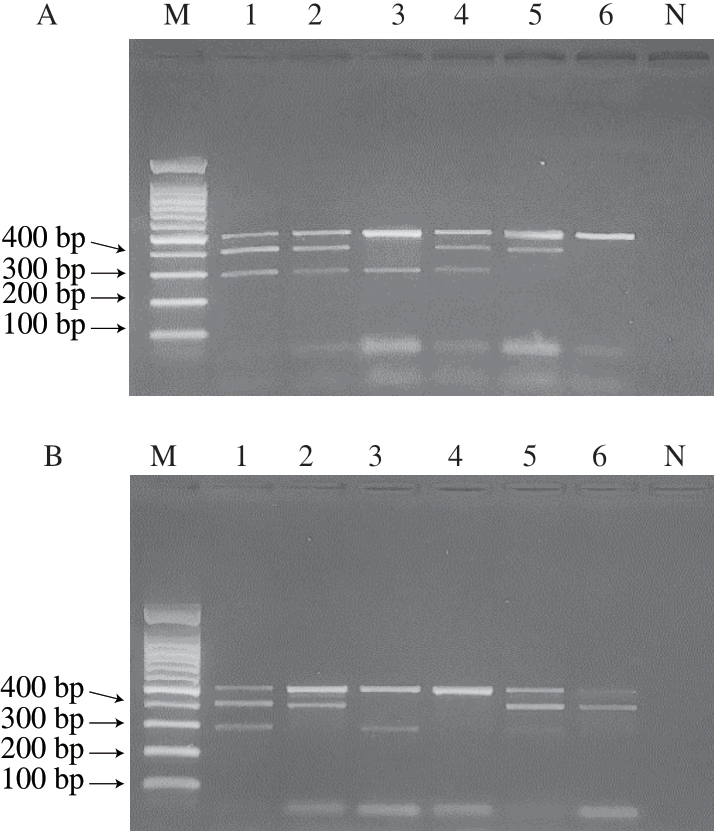
图5 随机抽取混合肉样琼脂糖凝胶电泳图谱
Fig. 5 Agarose gel electrophoresis patterns for detection of random mixed meat sample
泳道M. 100 bp DNA Ladder;泳道1. 阳性对照;泳道N. 空白对照;图A泳道2~6. 样品A1~A5;图B泳道2~6. 样品B1~B5。
2.5 狗、狐、貂市售样品检测
对50 份市售样品,包括猪肉、牛肉、羊肉、马肉、驴肉、鸡肉、鸭肉各3 份、狗肉29 份进行检测,结果发现:21 份非狗肉样品中均未检测到狗/狐/貂源性成分;29 份狗肉样品中,2 份出现貂特异性条带,1 份出现狐特异性条带,其余皆为狗肉。
3 讨 论
近年来多重PCR技术迅速发展,具有简单、快速、灵敏度和准确性高等优点[25],可以同时分析多个样品,应用前景非常广阔[26-29]。本研究根据狗、狐、貂动物基因序列的位点差异设计狗、狐、貂特异性引物,进行多重PCR检测。结果表明,所设计的引物只有在相应狗、狐、貂物种模板DNA存在的情况下才会发生反应,产生特异性目的条带,而且3 种物种之间DNA模板均无交叉反应,狗、狐、貂引物特异性好,而且其灵敏度可以达到10-4 ng/μL;经克隆测序分析证明所选取靶基因序列可作为种属特异性检测序列,用于犬型总科动物(狗、狐、貂)DNA成分检测。该方法的建立与应用为打击肉类产品市场违法违规行为提供了技术保障,对于政府部门的监督管理工作具有极其重要的意义。
4 结 论
本研究建立的多重PCR检测试剂盒可精准鉴别肉制品中狗、狐、貂源性成分,最低检测限可达10-4 ng/µL,特异性、灵敏度良好,具有较显著的现实意义和应用价值。对50 份市售样品进行检测,结果皆与长春食品药品检测中心鉴定结果相符,进一步证实本研究所建立多重PCR检测体系准确性良好。相较于荧光定量PCR检测技术[30],该试剂盒不受标准曲线及背景荧光的影响,无需额外设计探针及购买昂贵的检测仪器,更经济便捷,节省实验材料,为操作者提供便利,保证检测效率的同时维持检测的低成本,可广泛应用于常见犬型总科动物(狗、狐、貂)DNA成分检测。
[1] 秦天, 阮向东, 段招军, 等. 开展野生动物微生物研究应对未来新发传染病[J]. 疾病监测, 2021, 36(3): 209-213. DOI:10.3784/jbjc.202101260044.
[2] OH M, JACKSON J. Animal rights vs. Cultural rights: exploring the dog meat debate in south Korea from a world polity perspective[J].Journal of Intercultural Studies, 2011, 32(1): 31-56. DOI:10.1080/072 56868.2010.491272.
[3] 张知彬. 野生动物疫病暴发成因及其防控对策[J]. 中国科学院院刊,2021, 36(2): 188-198. DOI:10.16418/j.issn.1000-3045.20210126001.
[4] 周立, 李刚, 孔雪, 等. 新冠疫情下野生动物资源保护与利用的生物安全问题思考[J]. 生物资源, 2020, 42(4): 461-469. DOI:10.14188/j.ajsh.2020.04.013.
[5] 彭鹏, 初冬, 耿海东, 等. 我国陆生野生动物疫源疫病监测防控体系建设[J]. 南京林业大学学报(自然科学版), 2020, 44(6): 20-26.DOI:10.3969/j.issn.1000-2006.202004023.
[6] 江德斌. 深圳立法“禁食猫狗”具有破冰意义[N]. 民主与法制时报,2020-04-11(002). DOI:10.28579/n.cnki.nmzfz.2020.000586.
[7] 线洪臣, 阮瀛光. 几种动物肉的正常形态特征及性状异常肉的鉴别[J].现代畜牧兽医, 2006(3): 29-30.
[8] 袁凯, 张志勇, 席前, 等. 3 步混合变量选择策略在鸡肉近红外水分检测中的应用[J]. 食品与机械, 2020, 36(9): 72-76; 81.DOI:10.13652/j.issn.1003-5788.2020.09.012.
[9] 徐记各, 韩莹, 忻欣, 等. 广域照射拉曼光谱技术结合簇类独立软模式法快速鉴别原料肉及掺假肉[J]. 肉类研究, 2020, 34(5): 70-75.DOI:10.7506/rlyj1001-8123-20191211-303.
[10] 赵维章, 崔乃元, 张汉青, 等. 时间分辨荧光免疫层析定量检测牦牛肉中喹诺酮类药物[J]. 食品工业科技, 2020, 41(18): 239-243; 251.DOI:10.13386/j.issn1002-0306.2020.18.038.
[11] 乌恩其. 基于IgG物种特异性的家畜鲜肉真实性鉴定可行性研究[D]. 呼和浩特: 内蒙古农业大学, 2019: 1-32.
[12] MARIA M, VASILIKI M M, THEODORE K C, et al. Lateral flow test for meat authentication with visual detection[J]. Food Chemistry,2019, 274: 803-807. DOI:10.1016/j.foodchem.2018.09.063.
[13] 杨梦婕, 徐玲丽, 马学军, 等. 肉及肉制品中6 种致病菌的GeXP多重PCR检测[J]. 食品研究与开发, 2021, 42(13): 168-173.DOI:10.12161/j.issn.1005-6521.2021.13.025.
[14] 励炯, 江海, 吴琼. 基于COI序列的DNA微条形码技术鉴别熟肉制品中11 种肉掺假的研究[J]. 食品工业科技, 2021, 42(2): 99-104.DOI:10.13386/j.issn1002-0306.2020040288.
[15] 施姿鹤, JOSEF V, 刘丽. 肉及其加工制品的掺假鉴别技术研究进展[J].食品科学, 2019, 40(23): 319-326. DOI:10.7506/spkx1002-6630-20181229-354.
[16] 史莹莹, 康雨薇, 邵俊锋, 等. 实时荧光定量PCR鉴定肉制品中牛源性成分及其含量[J]. 食品研究与开发, 2020, 41(6): 158-163.DOI:10.12161/j.issn.1005-6521.2020.06.027.
[17] LI Tingting, JALBANI Y M, ZHANG Guilan, et al. Detection of goat meat adulteration by real-time PCR based on a reference primer[J]. Food Chemistry, 2019, 277: 554-557. DOI:10.1016/j.foodchem.2018.11.009.
[18] 张秀平, 苗丽, 黄世英, 等. 食品中鸭源性成分的微滴数字PCR定量检测方法的建立[J]. 中国兽医杂志, 2020, 56(6): 29-34.
[19] ALIKORD M, KERAMAT J, KADIVAR M, et al. Multiplex-PCR as a rapid and sensitive method for identification of meat species in halalmeat products[J]. Recent Patents on Food, Nutrition and Agriculture,2017, 8(3): 175-182. DOI:10.2174/2212798409666170113151213.
[20] LI Xinnan, GUAN Yifu. Specific identification of the adulterated components in beef or mutton meats using multiplex PCR[J]. Journal of AOAC International, 2019, 102(4): 1181-1185. DOI:10.5740/jaoacint.18-0338.
[21] QIN Panzhu, QIAO Dongqing, XU Jianguo, et al. Rapid visual sensing and quantitative identification of duck meat in adulterated beef with a lateral flow strip platform[J]. Food Chemistry, 2019, 294: 224-230.DOI:10.1016/j.foodchem.2019.05.030.
[22] LIU Wanwan, WANG Xiaonan, TAO Jingxi, et al. A multiplex PCR assay mediated by universal primers for the detection of adulterated meat in mutton[J]. Journal of Food Protection, 2019, 82(2): 325-330.DOI:10.4315/0362-028X.JFP-18-302.
[23] XU Rusu, WEI Shuang, ZHOU Guangbiao, et al. Multiplex TaqMan locked nucleic acid real-time PCR for the differential identification of various meat and meat products[J]. Meat Science, 2018, 137: 41-46.DOI:10.1016/j.meatsci.2017.11.003.
[24] 任易婕, 霍胜楠, 翟清燕, 等. 膜芯片技术对牛、羊、牦牛、驴肉源食品的掺伪鉴别[J]. 肉类研究, 2019, 33(6): 33-38. DOI:10.7506/rlyj1001-8123-20190417-081.
[25] 陈振中. 食品检验中多重PCR技术的应用[J]. 现代食品, 2020(23):147-149. DOI:10.16736/j.cnki.cn41-1434/ts.2020.23.043.
[26] JOANA C, FILIPA F, RICARDO A, et al. Multiplex PCR based strategy for detection of fungal pathogen DNA in patients with suspected invasive fungal infections[J]. Journal of Fungi, 2020, 6(4):308. DOI:10.3390/jof6040308.
[27] FRIEDER S, NEELE F, ROBIN K. A comparison of two multiplex-PCR assays for the diagnosis of traveller’s diarrhoea[J]. BMC Infectious Diseases, 2021, 21(1): 181. DOI:10.1186/S12879-021-05885-3.
[28] RIHNE T, NAMITA, SINGH K P, et al. Improvement in molecular detection of phytoplasma associated with rose by selection of suitable primers and development of a multiplex PCR assay[J]. 3 Biotech,2021, 11(4): 1-7. DOI:10.1007/S13205-021-02713-Y.
[29] 李明骏, 廖萌, 蒋开蓉, 等. 五种玉米病毒多重PCR检测体系的建立[J]. 植物保护学报, 2021, 48(2): 465-466. DOI:10.13802/j.cnki.zwbhxb.2021.2020281.
[30] 范梦晨, 韩爱云. 肉类掺假检测技术的研究进展[J]. 食品安全质量检测学报, 2021, 12(1): 236-241.