香肠作为一种即食类肉制品,在我国有着悠久的历史,香肠中不仅含有人体所需的蛋白质、维生素等物质,还具有口感好、吸收率高、饱腹性强等优点,是人们日常生活中经常食用的肉制品之一。随着人们对香肠肉制品需求日益增大,确保香肠在食用过程中的品质显得尤为重要[1]。但香肠制品中原材料标注信息与实际不符的情况越来越多,如使用低价低质的鸡肉、猪肉来替代营养价值较高的牛肉等[2]。这不仅扰乱了香肠制品的正常市场秩序,也侵害了消费者的权益和健康[3-5]。为保护消费者权益,相关监管部门不仅要采用相应的法律条款对生产厂家进行约束,同时还要采取有效的检测手段对肉制品进行品质监管[4-7]。
目前,主要采用聚合酶链式反应(polymerase chain reaction,PCR)和荧光PCR方法对肉制品中动物源性成分进行定性检测[8-11]。但是,随着肉制品掺假方式的变化和市场监管部门的需求变化,目前定性检测技术已不能满足掺假成分检测的需求,而定量检测肉制品中的动物源性成分含量已成为新的需求和研究方向。实时荧光PCR方法可以根据不同物种特异性基因的循环阈值(cycle threshold,Ct),建立标准曲线,从而推导其基因拷贝数,实现对多种混合肉样中不同成分的相对定量检测[12-13]。但该方法受PCR扩增效率的影响,导致检测值与真实值之间存在较大误差,因此在定量检测的准确度方面有所欠缺。
微滴式数字PCR(droplet digital PCR,ddPCR)采用微滴分析仪对PCR扩增后的每个微滴进行检测,实现了对靶序列核酸的绝对定量,在精确性、准确性和灵敏度方面效果更佳[14]。与实时荧光PCR相比,ddPCR技术不依赖标准曲线,不受PCR抑制物及PCR扩增效率的影响,通过单分子计数方式实现外源基因拷贝数绝对定量分析,更适合食品等基质复杂样本中掺假成分的准确定量检测[15]。本研究基于ddPCR技术,以鸡的转化生长因子β-3(transforming growth factor beta-3,TGFB3)基因、猪的朊蛋白(prion protein,PRNP)基因和牛的生长激素(growth hormone,GH)基因为靶基因,分别建立鸡、猪、牛源性成分ddPCR检测方法,有效实现香肠制品中鸡、猪、牛源性成分的定量检测。
牛肉、猪肉、鸡肉、鸭肉、鹅肉、羊肉、马肉、驴肉、狐狸肉、水貂肉和貉子肉,保存于本实验室;64 份香肠样品,均购于石家庄市不同农贸市场或超市。
食品中基因组DNA纯化系统 美国Promega公司;ddPCR预混液(不含脱氧尿嘧啶核苷三磷酸)、ddPCR机油、微滴发生盖、微滴反应板 美国Bio-Rad公司。
QX200微滴生成仪、QX200TM微滴读取仪、PX1热封仪、T100PCR仪 美国Bio-Rad公司;NanoDrop2000C超微量分光光度计 美国Thermo Fisher公司;MM400混合型球磨仪 德国莱驰公司;SL 202电子天平 德国赛多利斯公司;1-14台式离心机 德国Sigma公司;SQ2119N料理机 慈溪市西贝乐电器有限公司。
1.3.1 肉粉样品的制备及基因组DNA提取
称取500 g肉样组织,用洁净的剪刀将其剪成小块,放入料理机内,加入500 mL去离子水,进行搅拌,每次搅拌时间不超过30 s,搅拌成肉泥即可停止,收集肉泥于洁净的容器内备用。将制备好的肉泥尽量薄地平铺到放有烘焙纸的托盘内,65 ℃烘箱烘干16 h。
将烘干的肉泥剪成小块,放入混合型球磨仪的研磨罐内,肉样体积占研磨罐体积的1/3,放入25 粒直径7 mm研磨球,30 Hz/s研磨2 min。根据不同动物肉粉的密度,选择不同目的筛子对研磨后的肉粉进行过滤,收集过筛的肉粉于50 mL离心管中,密封,置于4 ℃保存备用。
使用Wizard Magnetic® DNA Purification System for Food试剂盒,按照说明书进行不同肉样基因组DNA的提取,并使用超微量分光光度计测定DNA质量浓度。
1.3.2 引物和探针设计
参考GeneBank中鸡物种的基因序列,根据单拷贝基因TGFB3设计鸡源性成分的特异性引物和探针;根据GB/T 25165—2010《明胶中牛、羊、猪源性成分的定性检测方法 实时荧光PCR法》[16]合成猪源性和牛源性成分的特异性引物和探针;所有引物探针均由生工生物工程(上海)股份有限公司合成,序列见表1。
表1 引物探针序列表
Table 1 Sequences of primers and probes used in this study

名称 引物探针序列(5′→3′) 扩增片段大小/bp目的基因(序列号) 来源鸡-F TGCTGCACCTATGAAAATGAATG鸡-R AGAATGCAGTCTCAGCACAACAC鸡-P FAM-TGCCCCGGTCTCCCTATGGTGC-Eclipse猪-F TTTGTGCATGACTGCGTCAAC 99 TGFB3(NC_006092.5) 本研究猪-R CTTGGTGGTCGTGGTCACTGT猪-P FAM-CACCGTCAAGCAGC-(NFQ)-MGB牛-F GCCAAATGTCTGGGTGTAGATACC 60 PRNP(NC_010459.5) GB/T 25165—2010牛-R CCGATGGATGTGTTCAGAGCT牛-P FAM-TGGGCTTTAGGGCTTCCGAATGTGAA-Eclipse 76 GH(AC_000176.1) GB/T 25165—2010
1.3.3 ddPCR反应体系及反应条件
ddPCR反应体系为20 μL,其中ddPCR预混液10 μL、上游引物和下游引物(10 μmol/L)各0.8 μL、探针(10 μmol/L)0.4 μL、DNA模板2 μL、ddH2O 6 μL。
ddPCR反应条件:95 ℃预变性5 min;94 ℃变性15 s,60 ℃退火及延伸60 s,40 个循环;98 ℃加热10 min,10 ℃保存,升温速率1 ℃/s。扩增结束后,将反应板置于微滴读取仪中进行微滴荧光读取。采用FAM通道收集微滴信号,读取阳性微滴数和阴性微滴数进行分析。
1.3.4 ddPCR特异性实验
取鸡、猪、牛、鸭、鹅、羊、马、驴、狐狸、水貂和貉子肉粉样品各30 mg,使用Wizard Magnetic® DNA Puri fication System for Food试剂盒提取肉粉基因组DNA,使用超微量分光光度计测定DNA质量浓度,并将其稀释至10~100 ng/μL。根据1.3.3节的反应体系和条件分别进行鸡、猪、牛源性成分ddPCR扩增,验证引物和探针特异性,并重复3 次。
1.3.5 肉粉质量与DNA质量浓度的线性相关性分析
分别称取5、10、20、30、40、50 mg鸡肉粉、猪肉粉和牛肉粉,提取肉粉基因组DNA,测定DNA质量浓度,每个质量浓度梯度设置3 个平行。对不同肉粉质量与DNA质量浓度间的线性相关性进行分析,以肉粉质量(x1,mg)为横坐标,以DNA质量浓度的平均值(y1,ng/μL)为纵坐标,建立肉粉质量-DNA质量浓度的线性关系式,即:y1=ax1+b。计算不同质量3 个平行肉粉样品DNA质量浓度的相对标准偏差(relative standard deviation,RSD),以RSD≤25%和标准曲线相关系数(R2)≥0.98作为成功建立标准曲线的评价标准[17]。
1.3.6 DNA质量浓度与基因片段拷贝数浓度的线性相关性分析
将提取的鸡肉粉、猪肉粉和牛肉粉DNA稀释至质量浓度分别为50、25、12.5、6.25、3.125、1.562 5、0.625 ng/μL,进行ddPCR检测,每个质量浓度设置3 个平行。通过对DNA质量浓度与基因片段拷贝数浓度间的线性相关性进行分析,建立DNA质量浓度-基因片段拷贝数浓度的线性关系。以DNA质量浓度(x2,ng/μL)为横坐标,基因片段拷贝数浓度的平均值(y2,copies/μL)为纵坐标,建立标准曲线,即:y2=cx2+d。计算基因片段拷贝数浓度的RSD,以RSD≤25%和标准曲线相关系数(R2)≥0.98作为成功建立标准曲线的评价标准。
1.3.7 肉粉质量与基因片段拷贝数浓度的线性相关性分析
一般情况下,20 μL ddPCR反应体系中靶基因含量不能超过105拷贝,而在实际样品检测过程中,提取的部分样品核酸质量浓度过高,需要将样品DNA稀释至合适质量浓度再进行ddPCR检测。设样品DNA质量浓度的稀释倍数为n,则y1=nx2。根据1.3.5和1.3.6节得到的肉粉质量与DNA质量浓度、DNA质量浓度与基因片段拷贝数浓度的线性关系,可以推出肉粉质量(x1,mg)、反应体系基因片段拷贝数浓度(y2,copies/μL)和样品稀释倍数(n)之间的关系,即![]() b、c、d为数值常数)。
b、c、d为数值常数)。
1.3.8 ddPCR方法的检测限分析
将鸡肉粉、猪肉粉和牛肉粉含量分别为1%、5%、10%、20%、50%、60%、80%、99%的混合肉粉样品提取DNA,分别检测鸡的TGFB3基因、猪的PRNP基因和牛的GH基因拷贝数,根据1.3.7节确定的肉粉质量与基因拷贝数浓度线性关系推算3 种肉粉的含量,并分别计算各含量样品测量值与实际值的绝对误差、相对误差、变异系数及回收率,确定该方法的检测限。
1.3.9 市售香肠样品的检测
将购买的64 份不同种类香肠样品按照1.3.1节进行处理。取100 mg肉粉,提取基因组DNA,测定DNA质量浓度并稀释至50 ng/μL,以之为模板,通过建立的ddPCR方法进行鸡源性、猪源性和牛源性成分检测,每份样品设置3 个平行。根据得到的相应动物源性成分的平均拷贝数浓度,计算香肠中鸡肉、猪肉和牛肉的含量。
根据测定结果,利用Excel软件绘制线性关系曲线,建立线性关系式。
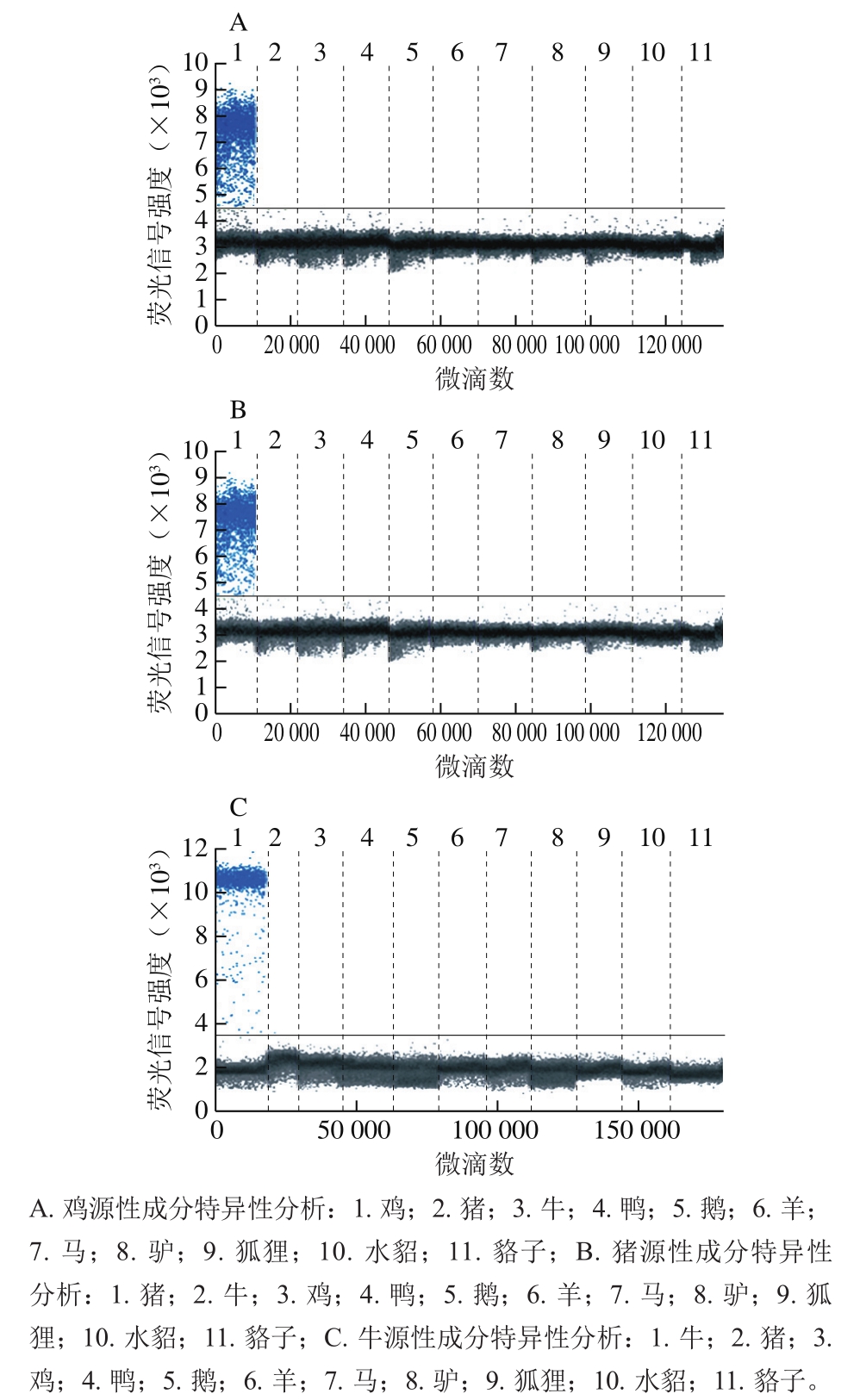
图1 ddPCR方法特异性实验结果
Fig. 1 Specificity of ddPCR
由图1可知,在ddPCR分析过程中,每个样品产生的总微滴数均大于12 000,符合泊松分布的统计要求。鸡肉粉、猪肉粉和牛肉粉DNA分别在鸡源性、猪源性和牛源性成分ddPCR反应中出现特异性微滴扩增,其他异源性肉粉基因组DNA均未出现明显扩增,表明所建立的ddPCR方法均具有良好的特异性。
DNA质量浓度与肉粉质量的线性关系式分别为:鸡:y1=28.350x1-6.124(R2=0.992);猪:y1=5.638x1-1.214(R2=0.996);牛:y1=4.376x1-5.330(R2=0.992)。线性关系式的相关系数均大于0.98,同时DNA质量浓度3 个平行样品间RSD均小于25%,满足评价标准。表明肉粉质量与DNA质量浓度之间存在良好的线性关系。
基因片段拷贝数浓度与DNA质量浓度的线性关系式分别为:鸡:y2=9.663x2+0.309(R2=0.999);猪:y2=11.520x2-1.071(R2=0.999);牛:y2=14.360x2-4.969(R2=0.998)。线性关系式的相关系数均大于0.98,同时基因拷贝数浓度3 个平行样间RSD均小于25%,满足评价标准。表明基因拷贝数浓度与DNA质量浓度之间存在良好的线性关系。
根据2.2、2.3节得到的线性关系式,可推出鸡肉粉、猪肉粉和牛肉粉质量(x1,mg)、反应体系基因片段拷贝数浓度(y2,copies/μL)和样品稀释倍数(n)之间的关系式分别如式(1)~(3)所示。

将各肉粉含量分别为1%、5%、10%、20%、50%、60%、80%、99%的鸡肉、猪肉、牛肉混合样品提取DNA,分别检测肉粉中鸡源性、猪源性和牛源性成分的基因拷贝数,计算鸡肉粉、猪肉粉和牛肉粉的含量。当鸡肉粉、猪肉粉和牛肉粉含量为5%~99%时,根据本实验确定的肉粉质量与基因拷贝数浓度线性关系计算混合样本中各肉粉含量,绝对误差均≤1.28%,相对误差均≤2.84%,变异系数均<6.50%,回收率为99.09%~102.80%,测量值接近于实际值。该方法检测牛肉中掺杂猪肉的最低检测限为5%,最高检测限达99%。
表2 市售香肠样品鸡、猪、牛源性成分定量检测结果
Table 2 Quantitative results of chicken-, porcine- and bovine-derived ingredients in sausage samples
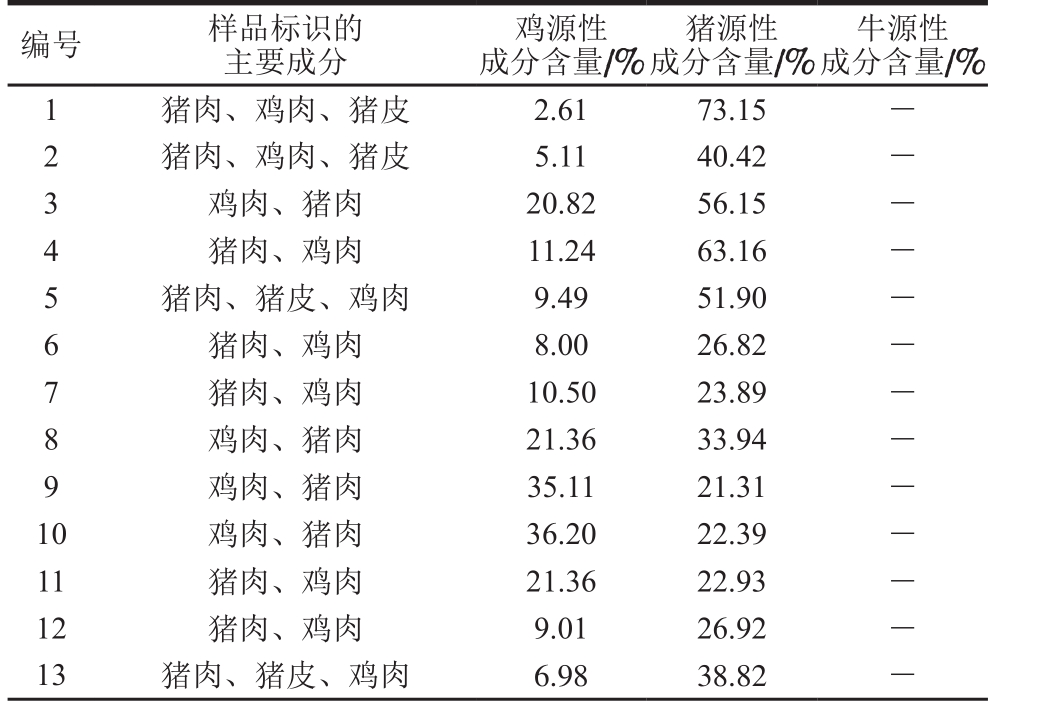
编号 样品标识的主要成分牛源性成分含量/%1 猪肉、鸡肉、猪皮 2.61 73.15 -2 猪肉、鸡肉、猪皮 5.11 40.42 -3鸡肉、猪肉 20.82 56.15 -4猪肉、鸡肉 11.24 63.16 -5 猪肉、猪皮、鸡肉 9.49 51.90 -6猪肉、鸡肉 8.00 26.82 -7猪肉、鸡肉 10.50 23.89 -8鸡肉、猪肉 21.36 33.94 -9鸡肉、猪肉 35.11 21.31 -10 鸡肉、猪肉 36.20 22.39 -11 猪肉、鸡肉 21.36 22.93 -12 猪肉、鸡肉 9.01 26.92 -13 猪肉、猪皮、鸡肉 6.98 38.82 -鸡源性成分含量/%猪源性成分含量/%
续表2

注:-. 未检出。
牛源性成分含量/%14 猪肉、猪皮、鸡肉 8.24 36.83 -15 猪肉、鸡肉、猪皮 16.09 58.69 -16 猪肉、鸡肉 7.59 28.63 -17 猪肉、鸡肉 13.99 68.43 -18 猪肉、鸡肉 5.01 57.93 -19 猪肉、鸡肉 14.32 28.65 -20 鸡肉、猪肉 35.11 33.86 -21 鸡肉、猪肉 21.71 15.62 -22 牛肉、猪肉 - 15.77 25.60 23 鸡肉、猪肉 33.98 43.35 -24 鸡肉、猪肉 21.26 17.26 -25 猪肉 - 89.89 -26 猪肉 - 99.09 -27 猪肉 18.68 69.94 -28 鸡肉、猪肉 28.48 9.24 -29 猪肉 - 39.02 -30 猪肉、鸡肉 11.16 40.95 -31 鸡肉、猪肉 28.48 15.55 -32 猪肉、鸡肉 9.97 73.89 -33 猪肉 - 47.50 -34 鸡肉、猪皮、猪肉 42.15 40.45 -35 鸡肉、猪肉 33.61 20.45 -36 猪肉 - 68.04 -37 猪肉、鸡肉 21.48 56.62 -38 猪肉、鸡肉 20.89 62.25 -39 鸡肉、猪肉 21.78 60.27 -40 鸡肉、猪肉 37.26 15.08 -41 鸡肉、猪肉 62.45 7.00 -42 猪肉、鸡肉 21.84 33.75 -43 猪肉、鸡肉、牛肉 17.70 50.57 18.21 44 猪肉、鸡肉 27.36 47.05 -45 鸡肉、猪肉 37.26 25.31 -46 猪肉 - 71.28 -47 猪肉 - 72.27 -48 牛肉 0.77 - 42.38 49 鸡肉、牛肉 45.64 - 2.48 50 牛肉、鸡肉 20.31 - 34.41 51 牛肉、鸡肉 30.43 - 35.40 52 牛肉 1.23 - 62.10 53 牛肉 - - 71.11 54 鸡肉、牛肉 60.56 - 9.02 55 牛肉 - - 33.11 56 鸡肉、牛肉 50.45 - 10.93 57 牛肉 - - 69.07 58 牛肉、鱼柳食品 - - 12.43 59 鸡肉、牛肉 48.56 - 7.50 60 鸡肉、牛肉、猪肉 27.26 14.32 28.21 61 鸡肉、牛肉 23.33 - 12.63 62 牛肉 - - 44.92 63 牛肉 - - 43.04 64 牛肉 - - 33.66编号 样品标识的主要成分鸡源性成分含量/%猪源性成分含量/%
通过建立的ddPCR方法对随机购买的64 份香肠样品进行鸡、猪、牛源性成分定量检测。由表2可知,所建立的ddPCR方法能够对香肠样品中鸡、猪、牛源性成分进行良好的定量检测,其中有3 份样品检出鸡源性成分,与实际标识的成分不符,含量分别为18.68%(27号样品)、1.23%(52号样品)和0.77%(48号样品);其余61 份样品检测结果与原料成分表标识一致。
香肠制品以次充好的问题越来越受到公众关注,建立有效的动物源性成分定量检测方法是食品安全研究的热点问题[18-19]。肉类制品中不同动物源性成分的含量是衡量掺假程度的有效途径,动物源性成分的定量检测是执法监管部门有效监管的技术保障。市售肉制品种类多样,所含成分复杂,DNA的提取过程往往会受到多种因素(如组织成分、样品处理方式等)的影响,进而影响检测结果,导致不能获得真实值。本研究采用干燥法对肉样进行处理,保证肉样间的细胞密度趋于一致,并以鸡、猪和牛的单拷贝基因作为定量检测的目的基因,检测结果更加准确、可信。
目前,ddPCR技术已被广泛用于食品中转基因成分、肉制品中动物源性成分和食源性致病菌的定量检测,其结果的精确性、准确性和灵敏度等方面较实时荧光PCR方法更优[20-23]。在动物源性成分检测中如何获得各种成分质量分数,一直是研究的难点和热点[24-25]。研究表明,动物源性成分的质量与基因拷贝数之间存在密切的线性关系[12,26-29]。目前线性回归标准曲线法是国内外最常用的定量检测方法[30-31]。苗丽等[27]建立了肉制品中牛源性和猪源性成分的ddPCR检测方法,并对10 份肉制品进行了定量检测,检测结果可以用来衡量肉制品的掺假程度;任君安等[29]根据复制蛋白A1基因建立羊源性和猪源性成分的ddPCR方法,通过二者基因拷贝数固定比值对14 份羊肉制品进行定量检测,方法具有很好的定量准确性。
本研究以鸡、猪和牛的单拷贝基因作为靶基因,建立鸡、猪、牛源性成分的ddPCR检测方法,并通过构建肉粉质量(mg)与DNA质量浓度(ng/μL)、DNA质量浓度(ng/μL)与基因拷贝数浓度(copies/μL)之间的线性关系,确定肉粉质量与基因拷贝数浓度之间的关系式。对64 份香肠样品的定量检测结果表明,本研究建立的ddPCR方法能够实现样品中相应动物源性成分的定量检测,检测限为5%,具有良好的实际应用价值。在3 份原料成分表中仅标示“猪肉”的样品中均检出不同含量的鸡源性成分,其中27号样品中鸡源性成分含量为18.68%,可认为该样品中蓄意添加了鸡肉成分;48号和52号样品中鸡源性成分含量仅为0.77%和1.23%,推断可能是生产过程中无意沾染导致或是产品中添加了微量鸡源性调味品。
本研究建立的鸡源性、猪源性和牛源性ddPCR方法能够有效地用于香肠中鸡、猪、牛源性成分的定量检测,初步区分蓄意添加还是生产过程中无意带入。
以鸡、猪和牛的单拷贝基因作为靶基因,建立了鸡、猪、牛源性成分的ddPCR检测方法,确定了肉粉质量与基因拷贝数浓度之间的关系式分别为:鸡:![]()
![]() ; 牛 :
; 牛 :![]() +1.218,肉粉含量为5%~99%,可以进行有效定量检测。
+1.218,肉粉含量为5%~99%,可以进行有效定量检测。
[1] 李志强, 王济民. 我国畜产品消费及消费市场前景分析[J]. 中国畜牧兽医文摘, 2003, 19(1): 7-8.
[2] 金萍, 丁洪流, 李培, 等. 2013年苏州地区肉及其制品掺假情况调查[J]. 中国食品卫生杂志, 2014, 26(2): 168-172. DOI:10.13590/j.cjfh.2014.02.016.
[3] OMAHONY P J. Finding horse meat in beef products: a global problem[J]. QJM-International Journal of Medicine, 2013, 106(6):595-597. DOI:10.1093/qjmed/hct087.
[4] ALI M E, RAZZAK M A, HAMID S B, et al. Multiplex PCR assay for the detection of five meat species forbidden in Islamic foods[J]. Food Chemistry,2015, 177(15): 214-224. DOI:10.1016/j.foodchem.2014.12.098.
[5] ZUKAL E, KORMENDY L. On calculation of ‘meat content’according to the quantitative ingredient declarations (QUID)[J].Journal of Food Engineering, 2007, 78(2): 614-621. DOI:10.1016/j.jfoodeng.2005.10.033.
[6] 任君安, 黄文胜, 葛毅强, 等. 肉制品真伪鉴别技术研究进展[J]. 食品科学, 2016, 37(1): 247-257. DOI:10.7506/spkx1002-6630-201601043.
[7] 蒋士强. 《食品安全法》的启迪: 对风险评估和标准体系的反思(修订稿)[C]//第十三届北京分析测试学术报告会及展览会: 2009分析仪器应用技术报告会论文集. 北京: 中国分析测试协会, 2009: 1-9.
[8] LIU L, CHEN F C, DORSEY J L, et al. Sensitive monoclonal antibody-based sandwich ELISA for the detection of porcine skeletal muscle in meat and feed products[J]. Journal of Food Science, 2006,71(1): 1-6. DOI:10.1111/j.1365-2621.2006.tb12393.x.
[9] MONTOWSKA M, POSPIECH E. Species-specific expression of various proteins in meat tissue: proteomic analysis of raw and cooked meat and meat products made from beef, pork and selected poultry species[J]. Food Chemistry, 2013, 136(3): 1461-1469. DOI:10.1016/j.foodchem.2012.09.072.
[10] CHOU C C, LIN S P, LEE K M, et al. Fast differentiation of meats from fifteen animal species by liquid chromatography with electrochemical detection using copper nanoparticle plated electrodes[J]. Journal of Chromatography B, 2007, 846(1): 230-239.DOI:10.1016/j.jchromb.2006.09.006.
[11] AMARAL J S, SANTOS C G, MELO V S, et al. Authentication of a traditional game meat sausage (Alheira) by species-specific PCR assays to detect hare, rabbit, red deer, pork and cow meats[J].Food Research International. 2014, 60(6): 140-145. DOI:10.1016/j.foodres.2013.11.003.
[12] IWOBI A, SEBAH D, KRAEMER I, et al. A multiplex real-time PCR method for the quantification of beef and pork fractions in minced meat[J]. Food Chemistry, 2015, 169(15): 305-313. DOI:10.1016/j.foodchem.2014.07.139.
[13] LÓPEZ-ANDREO M, ALDEGUER M, GUILLÉN I, et al. Detection and quantification of meat species by qPCR in heat-processed food containing highly fragmented DNA[J]. Food Chemistry, 2012, 134(1):518-523. DOI:10.1016/j.foodchem.2012.02.111.
[14] WEI Fu, ZHU Pengyu, WANG Chenguang, et al. A highly sensitive and speci fic method for the screening detection of genetically modi fied organisms based on digital PCR without pretreatment[J]. Scientific Reports, 2015, 5(1): 395-402. DOI:10.1038/srep12715.
[15] FLOREN C, WIEDEMANN I, BRENIG B, et al. Species identi fication and quanti fication in meat and meat products using droplet digital PCR(ddPCR)[J]. Food Chemistry, 2015, 173: 1054-1058. DOI:10.1016/j.foodchem.2014.10.138.
[16] 中国检验检疫科学研究院. 明胶中牛、羊、猪源性成分的定性检测方法 实时荧光PCR法: GB/T 25165—2010[S]. 北京: 中国标准出版社, 2010.
[17] Codex Alimentarius Commission. Guidelines on performance criteria and validation of methods for detection, identification and quantification of specific DNA sequences and specific proteins in foods: CAC/GL 74—2010[S].
[18] 李家鹏, 乔晓玲, 田寒友, 等. 食品和饲料中动物源性成分检测技术研究进展[J]. 食品科学, 2011, 32(9): 340-347.
[19] 何伟玲, 黄明, 张弛. 食品中肉类成分种属鉴别技术研究进展[J]. 食品科学, 2012, 33(3): 304-307.
[20] 刘津, 李婷, 冼钰茵, 等. 转基因大豆MON89788双重数字PCR通用定量检测方法的建立[J]. 食品科学, 2018, 39(4): 312-319.DOI:10.7506/spkx1002-6630-201804047.
[21] 张佳玲, 潘广, 章桂明, 等. 微滴式数字PCR定量检测转基因玉米品系VCO-01981-5[J]. 食品科学, 2017, 38(12): 253-259. DOI:10.7506/spkx1002-6630-201712038.
[22] 王珊, 李志娟, 苗丽. 微滴式数字PCR与实时荧光PCR检测羊肉制品中羊源和猪源性成分方法的比较[J]. 肉类工业, 2015(7): 38-41.DOI:10.3969/j.issn.1008-5467.2015.07.012.
[23] REN Junan, DENG Tingting, HUANG Wensheng, et al. A digital PCR method for identifying and quantifying adulteration of meat species in raw and processed food[J]. PLoS One, 2017, 12(3): 1-17.DOI:10.1371/journal.pone.0173567.
[24] 董莲华, 张玲, 姜君, 等. 大肠杆菌O157:H7微滴数字PCR定量方法的建立[J]. 分析化学, 2015, 43(3): 319-324. DOI:10.11895/j.issn.0253-3820.140569.
[25] 杨华, 汪小福, 肖英平, 等. 牛肉及其制品中掺入鸡肉、鸭肉和猪肉的多重数字PCR快速检测方法研究[J]. 浙江农业学报, 2017, 29(6):994-1000. DOI:10.3969/j.issn.1004-1524.2017.06.20.
[26] 刘立兵, 石蕊寒, 项佳林, 等. 应用微滴式数字聚合酶链式反应定量检测牛肉制品中的猪源性成分[J]. 肉类研究, 2018, 32(9): 39-44.DOI:10.7506/rlyj1001-8123-201809006.
[27] 苗丽, 张秀平, 陈静, 等. 微滴数字PCR法对肉制品中牛源和猪源成分的定量分析[J]. 食品科学, 2016, 37(8): 187-191. DOI:10.7506/spkx1002-6630-201608033.
[28] 苗丽, 张秀平, 陈静, 等. 肉制品中羊源性成分微滴数字PCR法定量检测方法的研究[J]. 食品工业科技, 2016, 37(4): 73-76.DOI:10.13386/j.issn1002-0306.2016.04.005.
[29] 任君安, 邓婷婷, 黄文胜, 等. 微滴式数字聚合酶链式反应精准定量检测羊肉中掺杂猪肉[J]. 食品科学, 2017, 38(2): 311-316.DOI:10.7506/spkx1002-6630-201702049.
[30] DRUML B, GRANDITS S, MAYER W, et al. Authenticity control of game meat products: a single method to detect and quantify adulteration of fallow deer (Dama dama), red deer (Cervus elaphus)and sika deer (Cervus nippon) by real-time PCR[J]. Food Chemistry,2015, 170(1): 508-517. DOI:10.1016/j.foodchem.2014.08.048.
[31] IWOBI A, SEBAH D, SPIELMANN G, et al. A multiplex realtime PCR method for the quantitative determination of equine(horse) fractions in meat produces[J]. Food Control, 2017, 74: 89-97.DOI:10.1016/j.foodcont.2016.11.035.
Quantitative Analysis of Chicken-, Porcine- and Bovine-Derived Ingredients in Sausage Products by Droplet Digital Polymerase Chain Reaction