鱿鱼属于软体动物门头足纲(Cephalopoda),是头足类中最重要的经济资源之一,鱿鱼加工也是国内水产品加工业的主要组成部分之一[1-2]。鱿鱼含有高质量的蛋白质、多不饱和脂肪酸以及具有各种生理功能的嘌呤、牛磺酸和胆固醇等,是被消费者广泛接受的营养、健康的水产品来源[3]。市场上的鱿鱼品种主要来源于柔鱼科(Ommastrephidae)和枪乌贼科(Loliginidae)[4]。与柔鱼科相比,枪乌贼科鱿鱼的产量较少,因此鱿鱼的开发种类以柔鱼科为主[5]。新西兰柔鱼(Nototodarus sloanii)属于褶柔鱼亚科双柔鱼属,又称新西兰双柔鱼。新西兰柔鱼仅分布在新西兰海域,由于其每年的捕捞量有配额限制,产量较少,因此价格较高[6]。北太平洋柔鱼(Ommastrephes bartramii)隶属于柔鱼亚科柔鱼属,近年来平均年产量为8~12 万t,是西北太平洋海域重要的经济类头足资源之一[7]。北太平洋柔鱼的肉质较薄,口感发面,价格相对于新西兰柔鱼较低。新西兰柔鱼和北太平洋柔鱼形态相似,市场上2 种柔鱼的产品也多以冻品及鱿鱼筒、鱿鱼圈、鱿鱼片等初加工形式存在,传统的形态学检验方法无法对失去形态特征的鱿鱼来源进行鉴别[8-9],使得2 种鱿鱼品种的混淆或掺假成为可能。因此,开发简单、有效的新西兰柔鱼和北太平洋柔鱼的分子生物学鉴定方法不仅可以保护消费者的合法权益,而且可以规范鱿鱼市场,避免贸易摩擦。
DNA分子标记是以个体间核苷酸序列变异为基础的遗传标记,是直接在DNA分子水平上检测生物间的差异[10]。因此,DNA分子标记在生物体各个组织、不同生长发育阶段均可检测到,不受环境因素、个体生长发育阶段及加工过程的影响[11]。近年来,DNA分子标记技术在物种来源鉴定和检测中呈现明显优势[12]。在动物的基因组序列中,核DNA和线粒体DNA为常用的两大类分子标记基因。与核DNA相比,线粒体DNA是母系遗传的单倍体,具有分子质量小、没有内含子、很少有等位基因重组现象等特点[13]。相对于其他线粒体DNA的编码基因,细胞色素C氧化酶亚基Ⅰ(cytochrome C oxidase subunit Ⅰ,COⅠ)基因包含的遗传信息量大,同时又有一定的进化速率,更适合分析亲缘关系密切的类群,目前已成为动物物种鉴定的公认条形码[14]。本研究对新西兰柔鱼和北太平洋柔鱼的线粒体COⅠ基因进行扩增和序列比对,拟从中挖掘2 种鱿鱼的特异性位点,并建立用于2 种柔鱼品种区分和鉴定的DNA分子标记方法,以期为加强鱿鱼的质量管理及规范鱿鱼市场的标签制度提供技术支持。
1 材料与方法
1.1 材料与试剂
新西兰柔鱼和北太平洋柔鱼的样品材料由烟台同德食品有限公司提供,秘鲁茎柔鱼样品购自市场。鱿鱼样品的代码和产地如表1所示。
表1 2 种鱿鱼样品信息
Table 1 Information about two squid species analyzed in this study

鱿鱼品种 产地 COⅠ基因在GenBank中的序列号 样品数新西兰柔鱼 新西兰 KY771095 10北太平洋柔鱼 中国 KX591660 10
海洋动物基因组DNA提取试剂盒(EE151-01)、2×PCR SuperMix预混液(AS111-11)、凝胶提取回收试剂盒(EG101-01)、琼脂糖凝胶 北京全式金生物技术有限公司;引物由英潍捷基(上海)贸易有限公司合成。
1.2 仪器与设备
TG18W台式高速微量离心机 长沙湘智离心机仪器有限公司;Mastecycler聚合酶链式反应(polymerase chain reaction,PCR)扩增仪 艾本德(中国)有限公司;BIS910凝胶成像系统 东胜创新生物科技有限公司;Mupid-2Plus水平电泳槽 日本Mupid公司。
1.3 方法
1.3.1 鱿鱼基因组DNA的提取
用新西兰柔鱼和北太平洋柔鱼的冷冻样品作为原料,用灭菌手术剪剪下约30 mg鱿鱼组织,剪碎后放入已灭菌的研钵中,加入液氮充分研磨至粉末,利用海洋动物基因组DNA提取试剂盒(EE151-01),严格按照说明书的步骤提取DNA。提取的DNA样品用1%琼脂糖凝胶电泳检测,-20 ℃保存备用。
1.3.2 线粒体COⅠ基因的扩增及测序
选用通用引物扩增新西兰柔鱼和北太平洋柔鱼的线粒体C OⅠ基因。正向引物为C OⅠF:5'-GGTCAACAAATCATAAAGATATTGG-3',反向引物为COⅠR:5'-TAAACTTCAGGGTGACCAAAAAAT CA-3'[15]。PCR反应体系总体积为20 µL,其中包含10 ng模板DNA、0.5 µmol/L正向引物、0.5 µmol/L反向引物、10.0 µL 2×PCR SuperMix,加超纯水补齐至20 µL。PCR反应过程:94 ℃预变性4 min;94 ℃变性30 s,60 ℃退火30 s,72 ℃延伸1 min,共35 个循环;最后72 ℃延伸7 min,4 ℃结束反应。取3 µL扩增产物,用加入染色剂溴化乙锭的1%琼脂糖凝胶进行电泳,然后在紫外凝胶成像系统下检测,观察条带是否为干净、清晰的单一条带。PCR反应后的产物经纯化后由生工生物工程(上海)股份有限公司进行测序。
1.3.3 品种特异性引物的设计
2 种鱿鱼测序后的COⅠ序列用SeqMan软件组装,通过Clustal Omega软件将2 种鱿鱼的COⅠ序列与GenBank中其他鱿鱼品种的COⅠ序列进行比对[16],选择新西兰柔鱼和北太平洋柔鱼品种稳定遗传的单核苷酸多态性(single nueleotide polymorphism,SNP)位点,利用引物设计软件Primer Premier 5分别设计新西兰柔鱼和北太平洋柔鱼的正向特异性引物(NSF和OBF),利用2 种鱿鱼的相同序列设计通用的反向引物(NOR)。引物的熔解温度设置为54~60 ℃,长度18~25 bp。正向品种特异性引物的3'末端通过改变单个碱基的方式人为引入错配碱基,以保证引物对特定鱿鱼品种的绝对特异性[17]。
1.3.4 新西兰柔鱼和北太平洋柔鱼的分子鉴定
利用设计的特异性引物对新西兰柔鱼和北太平洋柔鱼进行多重PCR鉴定。PCR鉴定的反应体系为20 µL,其中包含10 ng鱿鱼基因组DNA或2 种鱿鱼基因组DNA的混合物、0.5 µmol/L引物NSF、0.5 µmol/L引物OBF、1.0 µmol/L引物NOR、10.0 µL 2×Premix DNA polymerase。PCR反应过程:94 ℃预变性4 min;94 ℃变性30 s,56 ℃退火30 s,72 ℃延伸30 s,共35 个循环;最后72 ℃延伸7 min,4 ℃保温结束反应。取3 µL扩增产物混合染料后,用加入染色剂溴化乙锭的1%琼脂糖凝胶进行电泳,然后在紫外成像系统下进行检测。
2 结果与分析
2.1 2 种柔鱼品种特异性位点的选择
将新西兰柔鱼和北太平洋柔鱼的COⅠ序列用SeqMan软件拼接组装,2 种鱿鱼的GenBank序列号分别为KY771095和KX591660(表1)。利用Clustal Omega比对分析新西兰柔鱼和北太平洋柔鱼的品种特异性位点,并将2 种鱿鱼的COⅠ序列与GenBank中已登记的COⅠ序列比对,确定品种特异性位点的遗传稳定性。分别选取新西兰柔鱼和北太平洋柔鱼的特异性位点设计品种特异性引物。如图1所示,根据211~214 bp处的多态性位点设计北太平洋柔鱼的品种特异性引物,选择337~340 bp处的多态性位点设计新西兰柔鱼的品种特异性引物,并选择合适序列设计新西兰柔鱼和北太平洋柔鱼的通用反向引物。
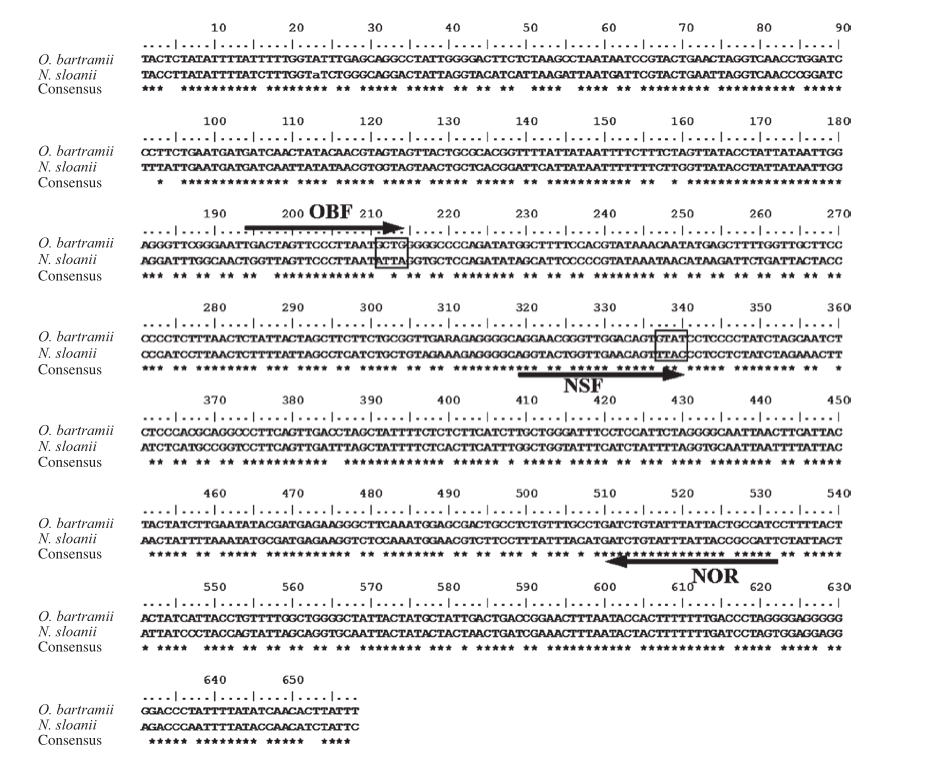
图1 新西兰柔鱼和北太平洋柔鱼的COⅠ序列比对图
Fig. 1 Comparison of COⅠ sequences of Nototodarus sloanii and Ommastrephes bartramii
2.2 引物设计
利用Primer Premier 5设计2 种鱿鱼的品种特异性引物及其反向引物,3 条引物的退火温度尽量保持一致。选择211~214 bp的SNP位点作为3'末端设计北太平洋柔鱼的品种特异性引物OBF(5'-TGACTAGTTCCCTTAATGCTG-3')。在引物OBF的序列中,倒数第1、3、4位碱基与新西兰柔鱼均不相同。根据新西兰柔鱼337~340 bp处的特异性位点,设计新西兰柔鱼的品种特异性引物NSF(5'-AGGTACTGGTTGAACAGTTCAC-3'),将倒数第3位碱基由T改为C,以保证引物NSF对新西兰柔鱼的绝对特异性。利用510~532 bp处的序列设计新西兰柔鱼和北太平洋柔鱼的通用反向引物NOR(5'-AATGGCRGTAATAAATACAGATC-3')。3 条引物的相对位置如图1所示。
2.3 多重PCR鉴定
利用图1所示的3 条引物组合对新西兰柔鱼和北太平洋柔鱼进行多重PCR鉴定,模板分别采用新西兰柔鱼和北太平洋柔鱼的模板DNA及二者的DNA混合样品,混合样品中北太平洋柔鱼的质量分数分别为1%、10%、50%。
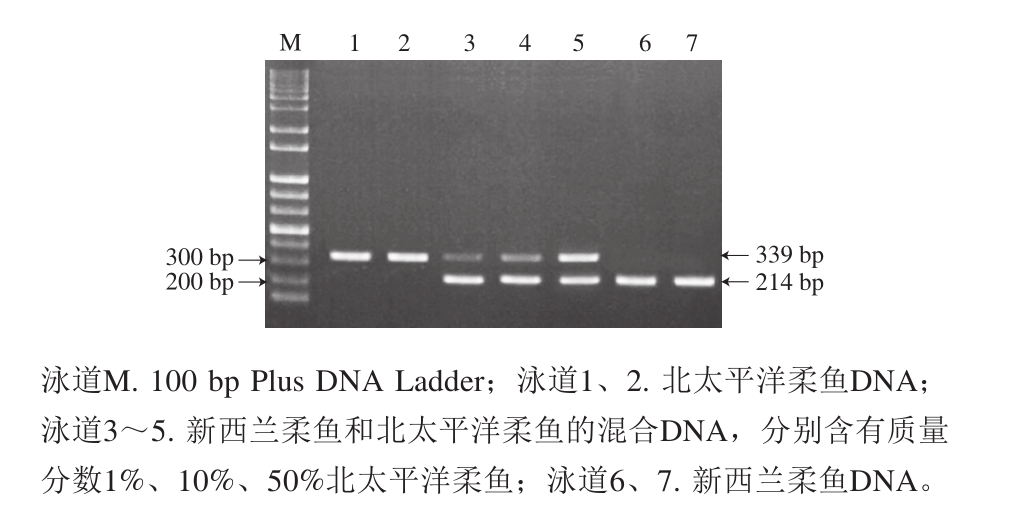
图2 新西兰柔鱼和北太平洋柔鱼特异性鉴定的凝胶电泳图
Fig. 2 Agarose gel electrophoretogram for specific authentication of N. sloanii and O. bartramii
由图2可知,3 条引物组合对不同鱿鱼样品扩增出了不同位置的条带。对不同样品的扩增条带利用胶回收试剂盒进行回收,纯化后进行双向测序,结果表明,新西兰柔鱼的特异性条带大小为214 bp,而北太平洋柔鱼的特异性条带大小为339 bp,且二者的序列和图1中完全一致。由此可见,引物NSF及OBF分别对各自的目标鱿鱼品种新西兰柔鱼和北太平洋柔鱼具有绝对的品种特异性。对于新西兰柔鱼和北太平洋柔鱼的DNA混合样品,3 条引物组合的多重PCR结果同时扩增出214 bp和339 bp的目标条带。在模板总含量为10 ng的条件下,该检测体系可以检测出新西兰柔鱼中1%的北太平洋柔鱼掺入。因此,本研究建立的多重PCR鉴定体系可以有效地对新西兰柔鱼和北太平洋柔鱼进行特异性区分和鉴定,检测限可低至0.1 ng。
2.4 市售样品的检测
为了检验所建立的方法对市售样品来源检测的有效性,利用图1所示的3 条引物组合,以市售新西兰柔鱼、北太平洋柔鱼样品及2 种鱿鱼的复配鱼糜DNA为模板,以超纯水和秘鲁茎柔鱼的DNA为阴性对照进行检测,PCR反应体系及反应条件如1.3.4节所述。
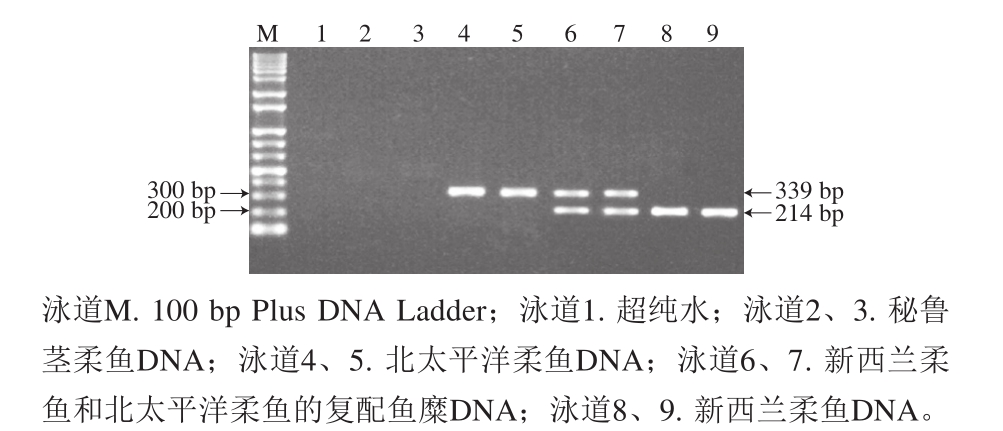
图3 市售样品检测结果的凝胶电泳图
Fig. 3 Agarose gel electrophoretogram for molecular authentication of commercial squid samples
由图3可知,不同鱿鱼样品扩增产物的条带位置和图2一致,2 个新西兰柔鱼样品扩增出214 bp的特异性条带,2 个北太平洋柔鱼样品扩增出339 bp的特异性条带,复配鱼糜则同时扩增出214 bp和339 bp的目标条带,表示样品中同时含有新西兰柔鱼和北太平洋柔鱼,超纯水和秘鲁茎柔鱼作为阴性对照则没有条带产生。因此,本研究建立的多重PCR鉴定体系可以对新西兰柔鱼和北太平洋柔鱼的市售样品及其复配鱼糜的来源进行准确鉴定和检测,可用于市场上新西兰柔鱼和北太平洋柔鱼的快速特异性鉴定。
3 结 论
随着我国远洋捕捞业的飞速发展,鱿鱼的捕捞量和消费量逐年上升,已成为我国水产加工业的主要组成部分之一[18]。鱿鱼的来源接近300 种,但由于不同鱿鱼品种的营养价值和口感差异,只有少量鱿鱼具有商业价值,因此不同品种的鱿鱼价格差异较大[4]。由于传统的形态学方法和蛋白质鉴定方法对鱿鱼初加工产品来源的鉴定存在局限性,因此亟需建立不同鱿鱼品种的鉴定方法,保证产品质量,促进鱿鱼产业的健康发展。近年来,随着分子生物学技术的发展,许多DNA分子标记技术被应用于不同鱿鱼品种的鉴定,如荧光定量PCR[19]、限制性片段长度多态性[20]、法医信息核苷酸测序(forensically informative nucleotide sequencing,FINS)[8]、DNA条形码[21]等。荧光定量PCR技术是近年来研究的热点,既能实现物种鉴定又可以达到定量的目的,具有更高的特异性和灵敏性[22],并且操作简单、重复性好[23],但是由于鱿鱼品种鉴定时需使用荧光定量PCR仪及TaqMan探针,限制了该方法在一般的分子生物学实验室的应用。FINS和DNA条形码技术均是利用未知物种的DNA条形码序列与数据库中的已知物种序列进行比对和聚类分析,从而将个体定位到生物类群中。但由于不同类群可能有不同的种内和种间距离,上述方法也存在一定的争议[24]。SNP分子标记是由于基因组中插入、缺失、置换和颠换单个核苷酸所引起的变异,与上述方法相比,基于SNP分子标记的位点特异性PCR技术检测快速、易实现自动化[25-27],是目前非常有发展潜力的分子标记方法。郝娜等[28]利用SNP分子标记实现了科氏滑柔鱼与剑尖枪乌贼之间的区分和鉴定,田潇然等[29]利用16S rRNA基因序列对秘鲁茎柔鱼和澳洲双柔鱼进行了鉴定。线粒体COⅠ基因具有足够的种内保守性和种间特异性[30],可作为通用的DNA条形码区分大多数动物物种,并且由于线粒体具有更大的基因拷贝数,在加工过程中保持相对完整,因此更适用于鱿鱼初加工及深加工产品原料来源的鉴定。
在线粒体COⅠ基因中,检测到新西兰柔鱼和北太平洋柔鱼的品种特异性位点,通过与GenBank中相应鱿鱼品种的序列进行比对,选取遗传稳定的特异性位点进行品种特异性引物设计,并通过在引物中均引入错配碱基的方式,大大提高了目标片段扩增的特异性。本研究建立的3 条引物的PCR鉴定体系在降低非特异性扩增的同时,大大提高了目标片段扩增的特异性,且该检测体系无需再对样品进行测序分析,可以检测出新西兰柔鱼中1%的北太平洋柔鱼掺入,检测限可低至0.1 ng。与传统需要测序的方法相比,本研究建立的鉴定方法具有简单、准确的优点,可以有效地对新西兰柔鱼、北太平洋柔鱼及市售产品进行特异性区分和鉴定。水产品标签制度的实施必须建立在对源性成分快速、准确鉴定的基础之上,本方法的建立为我国鱿鱼市场的规范管理提供了技术支持,也可为其他水产品源性成分的检测提供借鉴。
[1] 陈新军. 世界头足类资源开发现状及我国远洋鱿钓渔业发展对策[J].上海海洋大学学报, 2019, 28(3): 321-330. DOI:10.12024/jsou.20181102445.
[2] 曲映红, 陈新军, 陈舜胜. 我国鱿鱼加工利用技术研究进展[J].上海海洋大学学报, 2019, 28(3): 357-364. DOI:10.12024/jsou.20190102498.
[3] DENG Yun, LUO Yali, WANG Yuegang, et al. Effect of different drying methods on the myosin structure, amino acid composition, protein digestibility and volatile profile of squid fillets[J]. Food Chemistry, 2015, 171: 168-176. DOI:10.1016/j.foodchem.2014.09.002.
[4] ARKHIPKIN A I, RODHOUSE P G K, PIERCE G J, et al. World squid fisheries[J]. Reviews in Fisheries Science and Aquaculture,2015, 23: 92-252. DOI:10.1080/23308249.2015.1026226.
[5] Food and Agriculture Organization of the United Nations (FAO). The state of world fisheries and aquaculture 2016: contributing to food security and nutrition for all[R/OL]. Rome: Food and Agriculture Organization, 2016. http://www.fao.org/3/a-i5555e.pdf.
[6] 陈新军. 新西兰海域双柔鱼的生物学研究[J]. 中国水产科学, 1999,6(1): 27-32. DOI:10.3321/j.issn:1005-8737.1999.01.007
[7] 陈新军, 刘必林, 王尧耕. 世界头足类[M]. 北京: 海洋出版社, 2009:298-334.
[8] ESPIÑEIRA M, VIEITES J M, SANTACLARA F J. Species authentication of octopus, cuttlefish, bobtail and bottle squids (families Octopodidae, Sepiidae and Sepiolidae) by FINS methodology in seafoods[J]. Food Chemistry, 2010, 121: 527-532. DOI:10.1016/j.foodchem.2009.12.042.
[9] 苏杭. 几何形态测量学法在大洋性头足类种间及种群鉴别中的应用[D]. 上海: 上海海洋大学, 2016: 68-69.
[10] 王嘉鹤, 陈双雅, 陈伟玲, 等. DNA检测方法在鱼类物种鉴定中的应用[J]. 海南大学学报(自然科学版), 2012, 30(3): 293-300.DOI:10.15886/j.cnki.hdxbzkb.2012.03.020.
[11] 施文瑞, 王磊, 陈军平, 等. 鱼类性别特异性DNA分子标记研究进展[J]. 水产科学, 2018, 37(6): 862-866. DOI:10.16378/j.cnki.1003-1111.2018.06.023.
[12] 唐刘秀, 许志强, 葛家春. DNA分子标记技术在水产动物遗传育种中的应用[J]. 水产养殖, 2013, 34(10): 44-48. DOI:10.3969/j.issn.1004-2091.2013.10.011.
[13] 郑小东, 马媛媛, 程汝滨. 线粒体DNA标记在头足纲动物分子系统学中的应用[J]. 水产学报, 2015, 39(2): 294-303. DOI:10.3724/SP.J.1231.2015.59518.
[14] GALIMBERTI A, DE MATTIA F, LOSA A, et al. DNA barcoding as a new tool for food traceability[J]. Food Research International, 2013,50(1): 55-63. DOI:10.1016/j.foodres.2012.09.036.
[15] FOLMER O, BLACK M, HOEH W, et al. DNA primers for amplification of mitochondrial cytochrome coxidase subunit Ⅰ from diverse metazoan invertebrates[J]. Molecular Marine Biology and Biotechnology, 1994, 39(5): 294-299.
[16] SIEVERS F, WILM A, DINEEN D, et al. Fast, scalable generation of high-quality protein multiple sequence alignments using Clustal Omega[J]. Molecular Systems Biology, 2011, 7: 539. DOI:10.1038/msb.2011.75.
[17] KWOK S, CHANG S Y, SNINSKY J J, et al. A guide to the design and use of mismatched and degenerate primers[J]. Genome Research,1994, 3: S39-S47.
[18] 吴少杰, 张俊杰, 姚兴存, 等. 我国鱿鱼的综合加工利用现状与展望[J]. 食品研究与开发, 2011, 32(1): 154-156. DOI:10.3969/j.issn.1005-6521.2011.01.044.
[19] YE Jian, FENG Junli, LIU Shasha, et al. Identification of four squid species by quantitative real-time polymerase chain reaction[J].Molecular and Cell Probes, 2016, 30: 22-29. DOI:10.1016/j.mcp.2016.01.001.
[20] MCKEOWN N J, ROBIN J P, SHAW P W. Species-specific PCR-RFLP for identification of early life history stages of squid and other applications to fisheries research[J]. Fish Research, 2015, 167:207-209. DOI:10.1016/j.fishres.2015.02.007.
[21] 徐春燕, 沈长春, 蔡建堤, 等. 基于COⅠ基因的福建近海部分仔稚鱼DNA条形码分析[J]. 中国水产科学, 2017, 24(6): 1176-1183.DOI:10.3724/SP.J.1118.2017.17068.
[22] KANE D E, HELLBERG R S. Identification of species in ground meat products sold on the U.S. commercial market using DNA-based methods[J]. Food Control, 2016, 59: 158-163. DOI:10.1016/j.foodcont.2015.05.020.
[23] ESPIÑEIRA M, VIEITES J M. Rapid method for controlling the correct labeling of products containing common octopus (Octopus vulgaris) and main substitute species (Eledone cirrhosa and Dosidicus gigas) by fast real-time PCR[J]. Food Chemistry, 2012, 135:2439-2444. DOI:10.1016/j.foodchem.2012.07.056.
[24] UNDHEIM E A B, NORMAN J A, THOEN H H, et al. Genetic identification of Southern Ocean octopod samples using mtCOI[J].Comptes Rendus Biologies, 2010, 333: 395-404. DOI:10.1016/j.crvi.2010.02.002.
[25] LIAO P Y, LEE K H. From SNPs to functional polymorphism: the insight into biotechnology applications[J]. Biochemical Engineering Journal, 2010, 49: 149-158. DOI:10.1016/j.bej.2009.12.021.
[26] 唐立群, 肖层林, 王伟平. SNP分子标记的研究及其应用进展[J].中国农学通报, 2012, 28(12): 154-158. DOI:10.3969/j.issn.1000-6850.2012.12.028.
[27] QIAN Liang, DING Ge, ZHOU Qi, et al. Molecular authentication of Dendrobium loddigesii Rolfe by amplification refractory mutation system (ARMS)[J]. Planta Medica, 2008, 74: 470-473. DOI:10.1055/s-2008-1034360.
[28] 郝娜, 田惠丽, 计旭, 等. 科氏滑柔鱼和剑尖枪乌贼的多重PCR鉴定[J].食品科技, 2018, 43(1): 306-309; 315.
[29] 田潇然, 吕世鑫, 陈玉春, 等. 基于16S rRNA基因序列鉴定秘鲁茎柔鱼和澳洲双柔鱼[J]. 肉类研究, 2020, 34(1): 64-68. DOI:10.7506/rlyj1001-8123-20190904-209.
[30] HEBERT P D N, CYWINSKA A, BALL S L, et al. Biological identifications through DNA barcodes[J]. Proceedings Biological Sciences, 2003, 270: 313-321. DOI:10.1098/rspb.2002.2218.