斑节对虾(Penaeus monodon )俗称虎虾,与中华对虾和南美白对虾并称为世界三大水产养殖虾[1]。斑节对虾具有生长快、抗病力和对环境适应性强、养殖成本低、肉质鲜美、营养丰富等特点,深受养殖户和消费者喜爱,是东南亚和我国重要的水产养殖经济物种[2]。在工厂化养殖过程中,密度会影响水生动物的生存和生长、福利和健康、生理代谢等。提高养殖密度有利于增加产量并降低生产成本,但是当养殖密度过高时,水生动物又会产生应激生理反应和种内竞争,其生长、生存和生理代谢会受到影响。随着水产养殖工业化的快速发展,一些养殖者盲目提高养殖密度以追求高产量,结果会加剧种内竞争、疾病恶化和水环境污染。因此,养殖密度是影响水产品质量的重要因素之一,研究养殖密度对水产品肌肉品质的影响具有重要意义。
养殖密度对水产品肉质和风味影响的报道较少,主要集中在对大菱鲆、鲳鲹鱼、克氏原螯虾、罗氏沼虾等生理指标方面的研究。目前利用转录组测序技术研究肉品质的形成主要集中在金茅黑鸡胸肌的品质性状[3]、南方黄牛肌肉嫩度相关信号通路及其关键基因[4]等方面,鲜见关于水产动物肉品质转录组测序方面的研究。在分子层面上,决定肉质性状的大多数肌内脂肪和肌纤维受基因调控[5],具有稳定的高度遗传性[6],已经确定影响肉品质的主要基因为Halothane(Hal)和Rendement Napole(RN)基因[7]。目前通过转录组测序技术研究肉品质形成的分子机制已有一些研究。张凤[8]通过Illumina Solexa测序技术获得与猪脂肪发育相关的基因是WISP2和KLF6。邢凯等[9]用转录组测序筛选到长白猪、松辽黑猪中调控脂肪沉积的多个重要基因和通路。Li Guoxi等[10]采用miRNA-seq研究不同日龄荣昌猪背部皮下脂肪中的差异miRNA。彭兴[11]构建不同发育时间点的猪骨骼肌miRNA文库,并结合Solexa高通量测序初步确定影响肌肉增殖分化、参与骨骼肌生长调控的miRNA及其靶基因。Hou Xinhua等[12]在通城猪背最长肌混合RNA文库中检测出275 个miRNAs,确定miR-378与骨形成蛋白2和丝裂原活化蛋白激酶1有关。朱嘉宇[13]以猪不同类型肌肉组织为样本,采用转录组测序技术研究肌肉发育和肌纤维类型的关系。湖羊的肌肉生长发育及肉质形成与MYL3、ACTG2基因及ACTG2基因5′端非翻译区(长度83 bp)的剪接形式有关[14]。
目前应用高通量测序技术揭示水生动物肉品质形成的研究还很少。通过分析比较不同密度养殖的斑节对虾的一系列品质和风味指标,本团队已经确定随着养殖密度的增大,虾肌肉水分和灰分含量升高,粗蛋白和总糖含量降低,持水力极显著降低,感官和质构品质降低[15]。100 尾/m2密度养殖的斑节对虾品质和风味更好[16],但是具体机制尚不清楚。所以,本研究采用高通量转录组测序和实时定量聚合酶链式反应(quantitative real time polymerase chain reaction,qRT-PCR)技术,在分子水平上揭示养殖密度对斑节对虾肌肉品质的影响机理,以期为养殖户选择适宜的养殖密度、提高斑节对虾的肌肉品质、优化养殖模式、降低养殖成本等方面提供数据支持和理论指导。
1 材料与方法
1.1 材料与试剂
‘南海1号’斑节对虾苗,长度2 cm,购自中国水产科学研究院南海水产研究所,在天津横潜水产养殖有限公司进行养殖。养殖密度为100 尾/m2(M1)和300 尾/m2(M3)。高位池养殖模式,养殖条件为溶解氧5 mg/L以上,透明度30~40 cm,水温26~28 ℃,饵料人工喂养,每天投喂4~5 次,养殖周期84 d。整个养殖过程中虾生长、发育情况良好。每个养殖条件同时做3 次重复,虾长成后取样进行实验。
Green Real-time PCR Master Mix 日本Toyobo公司。
1.2 仪器与设备
NanoDrop微量紫外-可见分光光度计 美国Thermo Fisher科技公司;Qubit荧光定量仪 美国ABI公司;Agilent 2100生物分析仪 美国安捷伦公司;CFX96 qRT-PCR仪 美国伯乐公司。
1.3 方法
1.3.1 RNA提取和高通量测序
用Trizol法提取斑节对虾肌肉总RNA。通过琼脂糖凝胶电泳检测RNA是否发生降解和被污染。用微量紫外-可见分光光度计检测RNA的纯度(OD260 nm/OD280 nm)。利用荧光定量仪和生物分析仪分别对RNA浓度和完整性进行精确定量,确保用于转录组测序的RNA质量。然后由诺禾致源基因研究中心完成测序文库构建和高通量测序(Illumina HiSeqTM)。数据处理和分析参考邝良德[17]、Bemer[18]等的方法。依据测序结果中的基因功能(gene ontology,GO)注释,并结合KEGG数据库分析差异显著基因参与的代谢通路。
1.3.2 不同样本中差异基因相对表达量分析
参照Brown等[19]的方法,通过qRT-PCR技术,分析转录组测序数据中差异倍数较大的部分基因的相对表达量,所用引物如表1所示。
表1 用于qRT-PCR验证分析的引物序列
Table 1 Primer sequences for qRT-PCR used in study
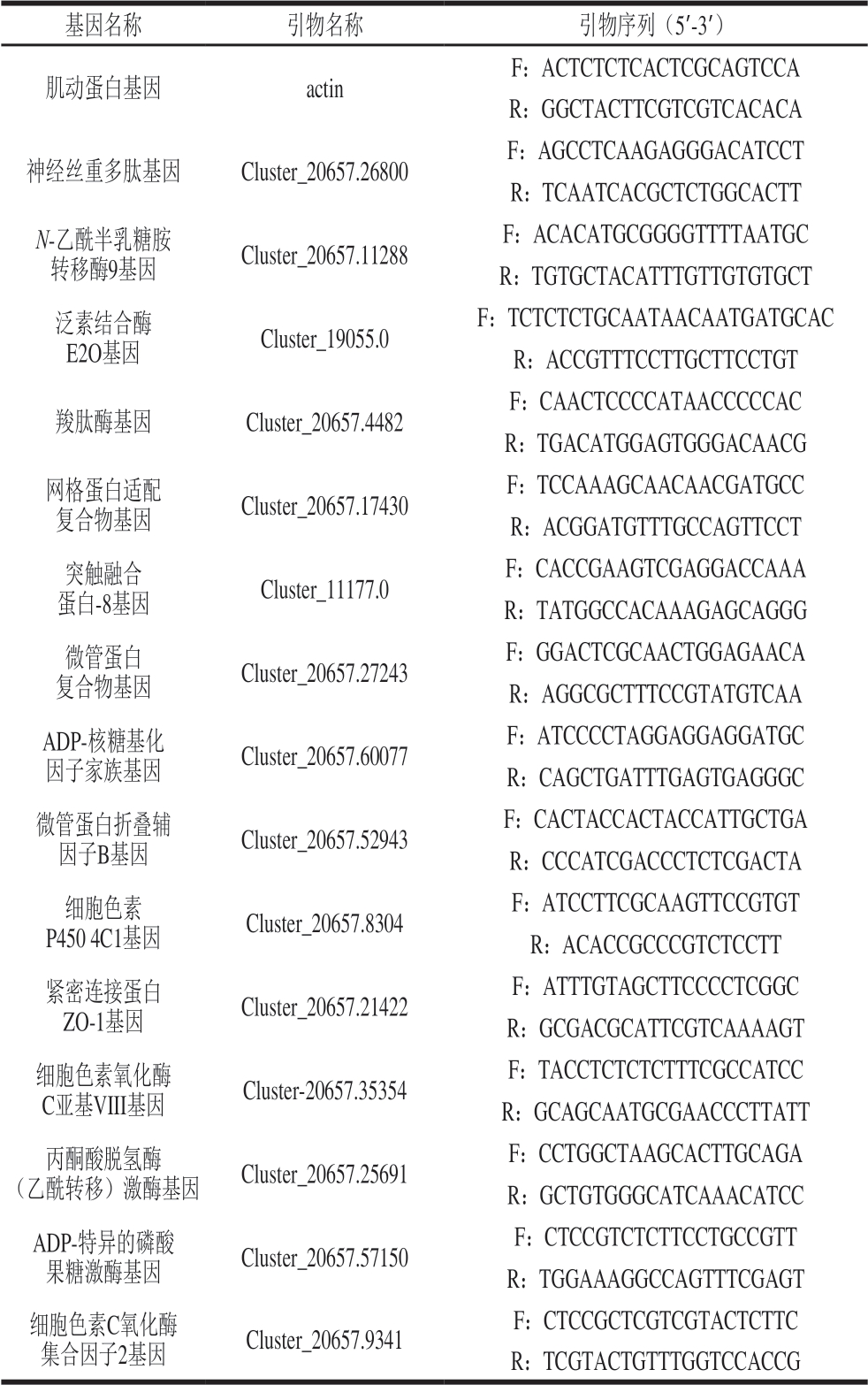
注:ADP. 二磷酸腺苷(adenosine diphosphate)。
基因名称 引物名称 引物序列(5′-3′)肌动蛋白基因 actin F:ACTCTCTCACTCGCAGTCCA R:GGCTACTTCGTCGTCACACA神经丝重多肽基因 Cluster_20657.26800 F:AGCCTCAAGAGGGACATCCT R:TCAATCACGCTCTGGCACTT N-乙酰半乳糖胺转移酶9基因 Cluster_20657.11288 F:ACACATGCGGGGTTTTAATGC R:TGTGCTACATTTGTTGTGTGCT泛素结合酶E2O基因 Cluster_19055.0 F:TCTCTCTGCAATAACAATGATGCAC R:ACCGTTTCCTTGCTTCCTGT羧肽酶基因 Cluster_20657.4482 F:CAACTCCCCATAACCCCCAC R:TGACATGGAGTGGGACAACG网格蛋白适配复合物基因 Cluster_20657.17430 F:TCCAAAGCAACAACGATGCC R:ACGGATGTTTGCCAGTTCCT突触融合蛋白-8基因 Cluster_11177.0 F:CACCGAAGTCGAGGACCAAA R:TATGGCCACAAAGAGCAGGG微管蛋白复合物基因 Cluster_20657.27243 F:GGACTCGCAACTGGAGAACA R:AGGCGCTTTCCGTATGTCAA ADP-核糖基化因子家族基因 Cluster_20657.60077 F:ATCCCCTAGGAGGAGGATGC R:CAGCTGATTTGAGTGAGGGC微管蛋白折叠辅因子B基因 Cluster_20657.52943 F:CACTACCACTACCATTGCTGA R:CCCATCGACCCTCTCGACTA细胞色素P4504C1基因 Cluster_20657.8304 F:ATCCTTCGCAAGTTCCGTGT R:ACACCGCCCGTCTCCTT紧密连接蛋白ZO-1基因 Cluster_20657.21422 F:ATTTGTAGCTTCCCCTCGGC R:GCGACGCATTCGTCAAAAGT细胞色素氧化酶C亚基VIII基因 Cluster-20657.35354 F:TACCTCTCTCTTTCGCCATCC R:GCAGCAATGCGAACCCTTATT丙酮酸脱氢酶(乙酰转移)激酶基因 Cluster_20657.25691 F:CCTGGCTAAGCACTTGCAGA R:GCTGTGGGCATCAAACATCC ADP-特异的磷酸果糖激酶基因 Cluster_20657.57150 F:CTCCGTCTCTTCCTGCCGTT R:TGGAAAGGCCAGTTTCGAGT细胞色素C氧化酶集合因子2基因 Cluster_20657.9341 F:CTCCGCTCGTCGTACTCTTC R:TCGTACTGTTTGGTCCACCG
qRT-PCR扩增体系:20 ng cDNA样品中加入2.5 μL SYBR® Green Real-time PCR Master Mix、400 nmol/L特异性引物,用双蒸水将体系体积补至25 μL。待测体系置于CFX96 qRT-PCR仪中,设定扩增条件为:95 ℃预变性1 min;95℃、15 s,60℃、15 s,72℃、45 s,以此条件扩增40 个循环;结束后在95~60 ℃(16 s)条件下绘制溶解曲线。以actin为内参基因,以2-ΔΔCt计算待测基因相对表达量,ΔΔCt按下式计算。
式中:CtTarget为待测基因循环阈(cycle threshold,Ct)值;Ctactin为内参基因Ct值;M1代表100 尾/m2密度养殖的样品;M3代表300 尾/m2密度养殖的样品。
1.4 数据处理
转录组测序数据用CASAVA Base Calling软件、RSEM软件分析,并与Nr(NCBI Non-Redundant Protein Sequences)、Nt(NCBI Nucleotide Sequences)、Pfam(Protein Family)、KOG(Eukaryotic Ortholog Groups)、Swiss-Prot(A Manually Annotated and Reviewed Protein Sequence Database)、KEGG(Kyoto Encyclopedia of Genes and Genomes)、GO 7 个数据库进行比对。最终的数据通过Origin 95软件作图。
2 结果与分析
2.1 斑节对虾转录组测序数据质量评估
将原始序列(Raw Reads)作为原始数据文件,通过CASAVA Base Calling软件进行分析。因为原始序列包含接头和低质量的序列,为了确保测序结果分析的准确性,必须对原始序列进行过滤,获得Clean Reads。随后的生物信息学分析都基于Clean Reads。测序数据的质量如表2所示。
表2 斑节对虾转录组测序数据产出质量
Table 2 Data output quality of Penaeus monodon transcriptome sequencing
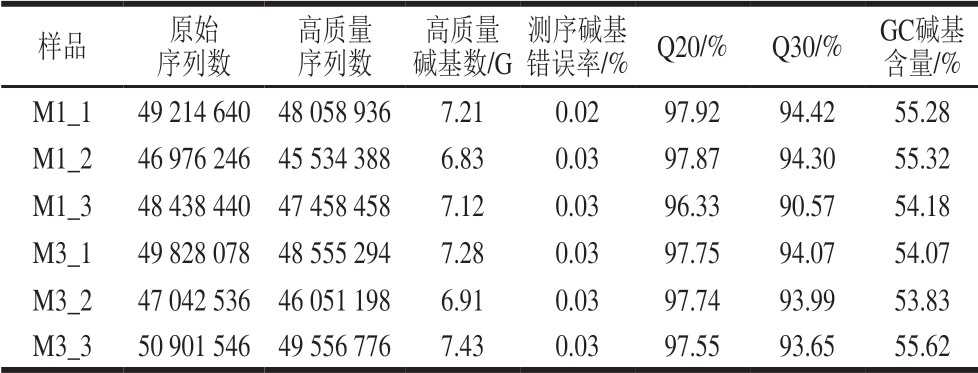
注:M1_1、M1_2、M1_3表示养殖密度100 尾/m2斑节对虾样品的3 次生物学重复;M3_1、M3_2、M3_3表示养殖密度300 尾/m2斑节对虾样品的3 次生物学重复;Q20.测序碱基质量值(Phred)数值大于20的碱基占总体碱基的百分比;Q30.Phred数值大于30的碱基占总体碱基的百分比。下同。
样品 原始序列数高质量序列数高质量碱基数/G测序碱基错误率/% Q20/% Q30/% GC碱基含量/%M1_1 4921464048058936 7.21 0.02 97.92 94.42 55.28 M1_2 4697624645534388 6.83 0.03 97.87 94.30 55.32 M1_3 4843844047458458 7.12 0.03 96.33 90.57 54.18 M3_1 4982807848555294 7.28 0.03 97.75 94.07 54.07 M3_2 4704253646051198 6.91 0.03 97.74 93.99 53.83 M3_3 5090154649556776 7.43 0.03 97.55 93.65 55.62
由表2可知,在6 组样品中,Q20的最小值为96.33%,Q30的最小值为90.57%,Q20和Q30均超过90%,表明所有测序数据均符合生物信息学分析的要求。
2.2 斑节对虾转录组测序Reads与参考序列的比对
通过Trinity拼接获得用于转录组分析的参考序列(ref),并将每个样品的Clean Reads与参考序列进行比对。比对时采用RSEM软件,Bowtie2设置为默认参数mismatches 0。
表3 斑节对虾转录组测序Reads与参考序列比对情况
Table 3 Comparisons between Reads and reference sequences of Penaeus monodon transcriptome sequencing
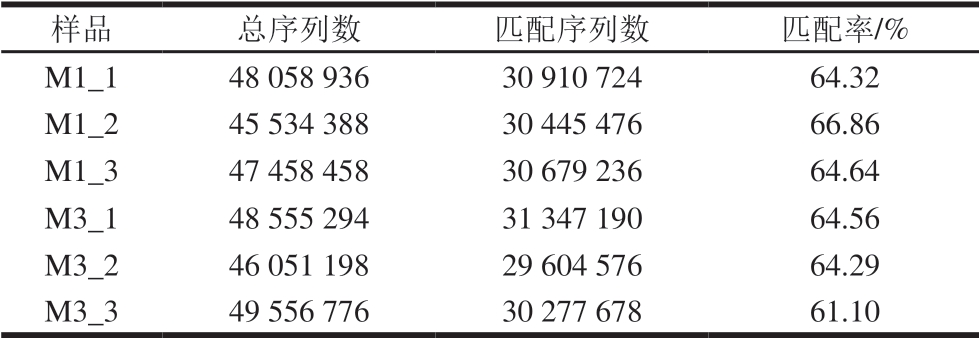
样品 总序列数 匹配序列数 匹配率/%M1_1 48058936 30910724 64.32 M1_2 45534388 30445476 66.86 M1_3 47458458 30679236 64.64 M3_1 48555294 31347190 64.56 M3_2 46051198 29604576 64.29 M3_3 49556776 30277678 61.10
由表3可知,Clean Reads与参考序列匹配率为61.10%~66.86%,说明测序结果对于无参转录组的分析是可靠的。
2.3 差异基因的筛选
以不同密度养殖的斑节对虾中基因Reads数之比为差异倍数,绘制火山图,可以直观显示Pval和log2(差异倍数)之间的关系,纵坐标为差异显著性,-lg(Pval)越大,差异越显著;P<0.05和|log2(差异倍数)|>1即为差异表达的基因。
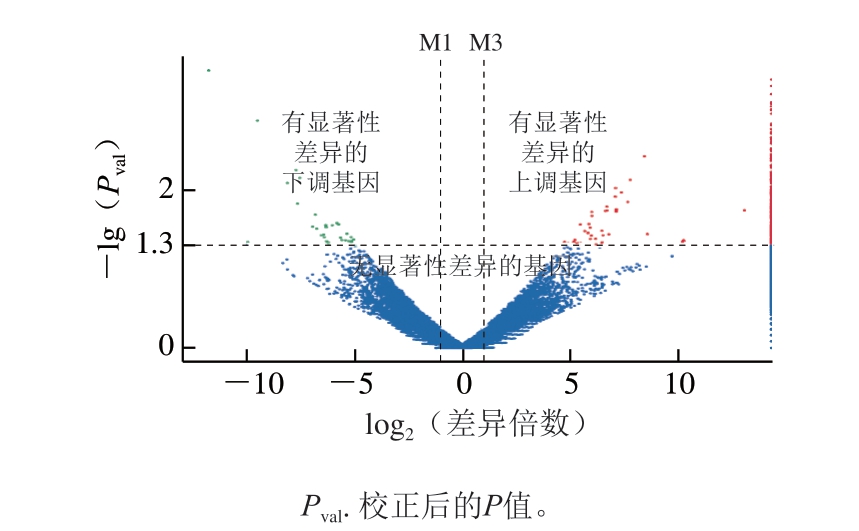
图1 不同密度养殖的斑节对虾间基因差异表达分析火山图
Fig. 1 Volcanic map of differentially expressed genes between Penaeus monodon with different stocking densities
由图1可知,不同密度养殖的斑节对虾有778 个差异表达基因,其中365 个是上调基因,413 个是下调基因。
2.4 不同密度养殖的斑节对虾肌肉中与品质相关的差异基因
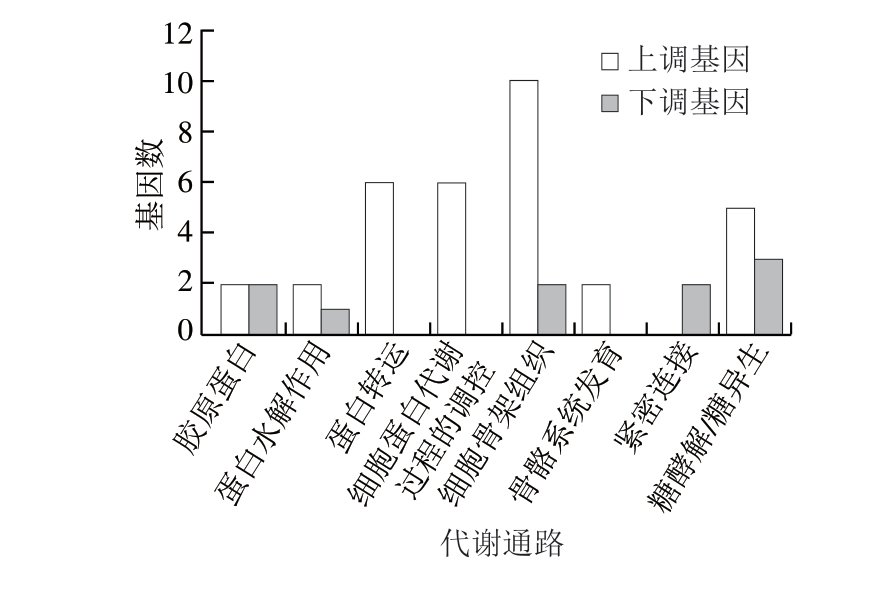
图2 不同密度养殖的斑节对虾肌肉中与代谢途径相关的差异基因
Fig. 2 Differentially expressed genes associated with metabolism pathway
in the muscle of Penaeus monodon cultured at different stocking densities
不同密度养殖的斑节对虾的差异基因涉及蛋白质代谢、肌肉发育和生长、碳水化合物代谢途径。由图2可知,100 尾/m2和300 尾/m2密度养殖的斑节对虾肌肉中存在4 个与胶原蛋白相关的差异基因,包括2 个上调基因和2 个下调基因。与蛋白水解作用相关的差异表达基因有3 个,包括2 个上调基因和1 个下调基因。6 个上调基因参与蛋白质运输。6 个上调基因与细胞蛋白质代谢调节相关。与细胞骨架组织有关的基因有12 个,包括2 个下调基因和10 个上调基因。2 个上调基因与骨骼系统发育相关。2 个下调基因与肌肉连通性相关。与糖酵解或糖异生有关的上调基因是细胞色素氧化酶C亚基Ⅷ、丙酮酸脱氢酶(乙酰转移蛋白)激酶、丙酮酸脱氢酶激酶2/3/4、保守的假设蛋白和假设蛋白CAPTEDRAFT_177854基因,下调基因是ADP特异的磷酸果糖激酶、细胞色素C氧化酶集合因子2和细胞色素C氧化酶亚基Ⅱ基因。
2.5 与肌肉品质相关的差异基因的相对表达量
表4 qRT-PCR验证M1和M3组差异基因的相对表达量
Table 4 qRT-PCR verification of the relative expression of differential genes in M1 and M3 groups
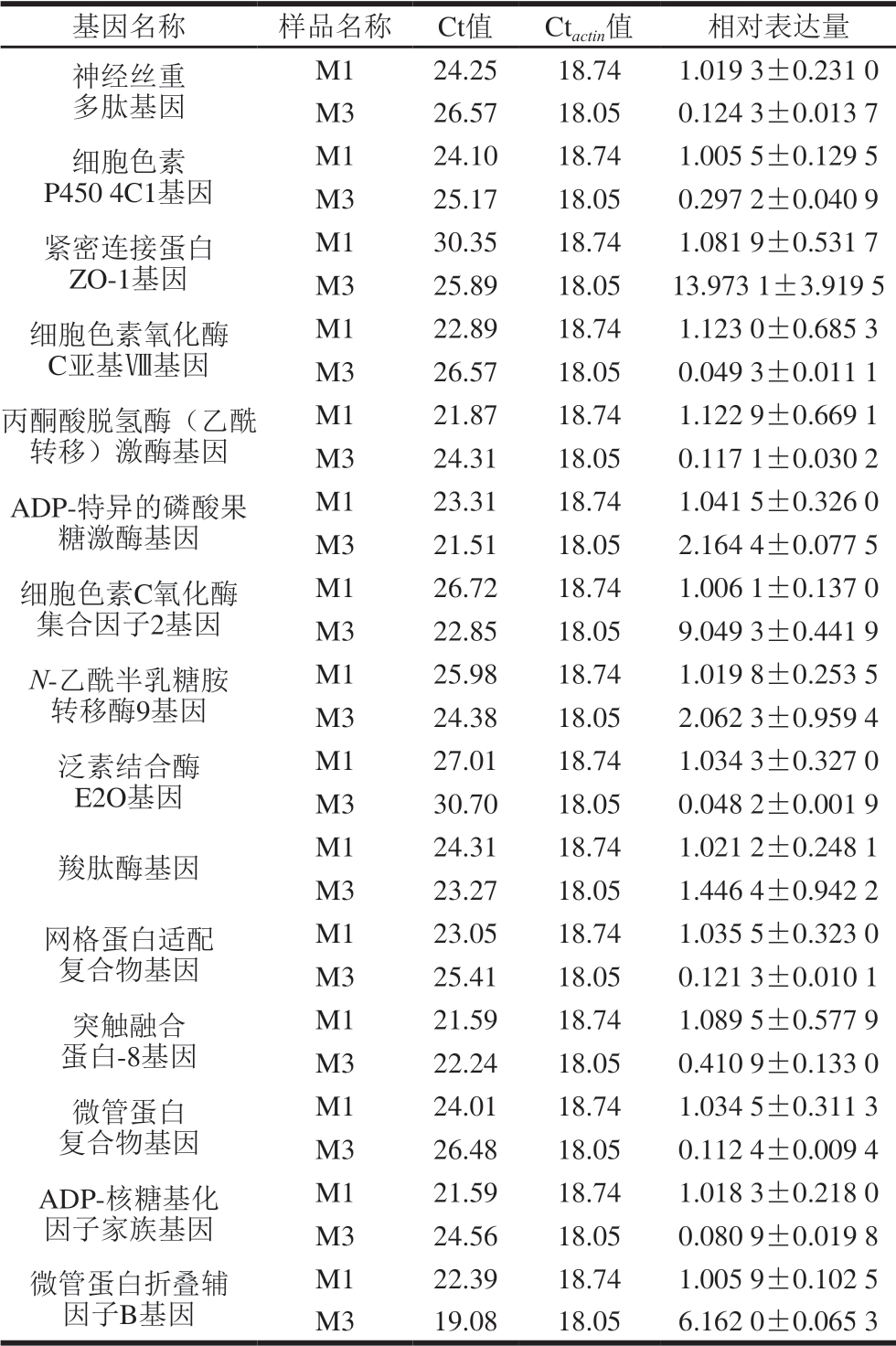
注:Ct值、Ctactin值是3 次重复测定的平均值。
基因名称 样品名称 Ct值 Ctactin值 相对表达量神经丝重多肽基因M1 24.25 18.74 1.0193±0.2310 M3 26.57 18.05 0.1243±0.0137细胞色素P4504C1基因M1 24.10 18.74 1.0055±0.1295 M3 25.17 18.05 0.2972±0.0409紧密连接蛋白ZO-1基因M1 30.35 18.74 1.0819±0.5317 M3 25.89 18.05 13.9731±3.9195细胞色素氧化酶C亚基Ⅷ基因M1 22.89 18.74 1.1230±0.6853 M3 26.57 18.05 0.0493±0.0111丙酮酸脱氢酶(乙酰转移)激酶基因M1 21.87 18.74 1.1229±0.6691 M3 24.31 18.05 0.1171±0.0302 ADP-特异的磷酸果糖激酶基因M1 23.31 18.74 1.0415±0.3260 M3 21.51 18.05 2.1644±0.0775细胞色素C氧化酶集合因子2基因M1 26.72 18.74 1.0061±0.1370 M3 22.85 18.05 9.0493±0.4419 N-乙酰半乳糖胺转移酶9基因M1 25.98 18.74 1.0198±0.2535 M3 24.38 18.05 2.0623±0.9594泛素结合酶E2O基因M1 27.01 18.74 1.0343±0.3270 M3 30.70 18.05 0.0482±0.0019羧肽酶基因 M1 24.31 18.74 1.0212±0.2481 M3 23.27 18.05 1.4464±0.9422网格蛋白适配复合物基因M1 23.05 18.74 1.0355±0.3230 M3 25.41 18.05 0.1213±0.0101突触融合蛋白-8基因M1 21.59 18.74 1.0895±0.5779 M3 22.24 18.05 0.4109±0.1330微管蛋白复合物基因M1 24.01 18.74 1.0345±0.3113 M3 26.48 18.05 0.1124±0.0094 ADP-核糖基化因子家族基因M1 21.59 18.74 1.0183±0.2180 M3 24.56 18.05 0.0809±0.0198微管蛋白折叠辅因子B基因M1 22.39 18.74 1.0059±0.1025 M3 19.08 18.05 6.1620±0.0653
选择转录组测序数据中具有显著性差异和代表性的基因进行qRT-PCR验证。以M1组基因的表达水平为基准,确定M3组相应基因的相对表达量。由表4可知,相比于100 尾/m2密度养殖,在300 尾/m2密度养殖的斑节对虾中,神经丝重多肽、细胞色素P4504C1、细胞色素氧化酶C亚基Ⅷ、丙酮酸脱氢酶(乙酰转移)激酶、泛素结合酶E2O、网格蛋白适配复合物、突触融合蛋白-8、微管蛋白复合物和ADP-核糖基化因子家族的相对表达量下调,其中细胞色素氧化酶C亚基Ⅷ、泛素结合酶E2O和ADP-核糖基化因子家族的相对表达量分别为0.0493、0.0482和0.0809。这些基因的下调表达导致斑节对虾蛋白代谢、糖代谢、蛋白质的泛素化修饰等途径受到显著抑制,对肉品质造成负面影响。其余基因在300 尾/m2密度养殖的斑节对虾中表达上调,紧密连接蛋白ZO-1在M3组的表达量是M1组的13.9731 倍,细胞色素C氧化酶集合因子2基因在M3组的相对表达量为9.0493。
3 讨 论
转录组测序结果揭示不同密度养殖的斑节对虾的差异基因位于蛋白质代谢、肌肉发育和生长、碳水化合物代谢途径。qRT-PCR进一步验证了转录组测序的结果。神经丝重多肽是轴突的主要结构蛋白,是成熟髓鞘结构形成的重要标志之一[20]。Wang等[21]证实,神经丝重多肽是一种中间纤维成分,保护足状突细胞免受损伤。细胞色素C氧化酶位于线粒体呼吸链末端,参与电子传递,并将质子从线粒体膜内侧泵到线粒体膜外侧[22]。丙酮酸脱氢酶系在糖代谢调控中起重要作用,催化丙酮酸不可逆氧化脱羧生成乙酰辅酶A,并将糖酵解途径与柠檬酸循环连通[23-24]。丙酮酸脱氢酶激酶可以通过磷酸化调节丙酮酸脱氢酶系的活性[25]。泛素化是真核生物中一种常见的蛋白质降解途径,调节细胞周期、DNA修复、逆境胁迫和蛋白质的细胞内定位等生物学过程[26]。泛素结合酶是泛素/蛋白酶体途径的重要组成部分,在蛋白质泛素化中有重要作用。网格蛋白适配器复合体在协调网格蛋白涂层的组装中发挥了重要作用[27]。Renigunta等[28]研究发现,通过网格蛋白介导的内吞作用,以协同方式将未组装的突触融合蛋白-8和钾通道内化。ADP核糖基化因子在所有真核细胞中均有表达,并涉及囊泡运输、脂质代谢、微管动力学和细胞过程等方面[29]。300 尾/m2高密度养殖导致斑节对虾额外的能量代谢增强,与倪嘉豪等[30]对银鲳幼鱼的研究一致,降低了斑节对虾的生长性能,导致肉品质远低于100 尾/m2低密度养殖组。
4 结 论
100 尾/m2密度养殖的斑节对虾肌肉品质较好是由于胶原蛋白代谢、蛋白水解、蛋白转运、细胞蛋白代谢、细胞骨架组织、肌肉连接性、葡萄糖代谢途径中相关基因,即神经丝重多肽、细胞色素P4504C1、细胞色素C氧化酶亚基Ⅷ、丙酮酸脱氢酶(乙酰转移)激酶、泛素结合酶E2O、网格蛋白适配复合物、突触融合蛋白-8、微管蛋白复合物、ADP-核糖基化家族基因表达上调,使肉质鲜美。
[1]王娟. 中国对虾、南美白对虾和斑节对虾肌肉营养成分的比较[J]. 食品科技, 2013, 38(6): 146-150. DOI:10.13684/j.cnki.spkj.2013.06.009.
[2]陈劲松, 周发林, 江世贵, 等. 斑节对虾幼虾不同家系的耐低盐能力比较[J]. 水产学报, 2017, 41(5): 687-693. DOI:10.11964/jfc.20160510392.
[3]李国辉, 谢恺舟, 戴国俊, 等. 金茅黑鸡肉品质性状的关联转录组分析[J]. 中国畜牧杂志, 2019, 55(12): 85-90. DOI:10.19556/j.0258-7033.20190301-04.
[4]孟祥忍, 张成龙, 樊永亮, 等. 基于RNA-Seq转录组测序技术揭示南方黄牛肉质嫩度调控相关的分子机制[J]. 中国畜牧杂志, 2017,53(9): 26-32; 68. DOI:10.19556/j.0258-7033.2017-09-026.
[5]LARZUL C, LEFAUCHEUR L, ECOLAN P, et al. Phenotypic and genetic parameters for Longissimus muscle fiber characteristics in relation to growth, carcass, and meat quality traits in large white pigs[J]. Journal of Animal Science, 1997, 75(12): 3126-3137.DOI:10.2527/1997.75123126x.
[6]WARNER R D, GREENWOOD P L, PETHICK D W, et al. Genetic and environmental effects on meat quality[J]. Meat Science, 2010,86(1): 171-183. DOI:10.1016/j.meatsci.2010.04.042.
[7]SALAS R C D, MINGALA C N. Genetic factors affecting pork quality: halothane and rendement napole genes[J]. Animal Biotechnology, 2016, 28(2): 148-155. DOI:10.1080/10495398.2016.1243550.
[8]张凤. Myostatin对猪肌卫星细胞和脂肪来源干细胞成脂分化的调控机制研究[D]. 武汉: 华中农业大学, 2016: 77-90.
[9]邢凯, 侯卓成, 王晓铄, 等. 背膘极端表现猪脂肪和肝脏组织的表达谱分析[C]//中国畜牧兽医学会2013年学术年会论文集. 北京: 中国畜牧兽医学会, 2013: 171.
[10]LI Guoxi, LI Yanjie, LI Xinjian, et al. MicroRNA identity and abundance in developing swine adipose tissue as determined by Solexa sequencing[J]. Journal of Cellular Biochemistry, 2011, 112(5):1318-1328. DOI:10.1002/jcb.23045.
[11] 彭兴. 猪肌肉发育相关microRNA-21靶标基因TSC1和SMAD7的鉴定[D]. 长沙: 湖南农业大学, 2013: 37-55.
[12]HOU Xinhua, TANG Zhonglin, LIU Honglin, et al. Discovery of microRNAs associated with myogenesis by deep sequencing of serial developmental skeletal muscles in pigs[J]. PLoS One, 2012, 7(12):52123. DOI:10.1371/journal.pone.0052123.
[13]朱嘉宇. 猪肌纤维类型关键基因筛选及miR-423-5p的功能研究[D].杨凌: 西北农林科技大学, 2016: 72-73.
[14]刘凤祥. γ-内脏平滑肌型actin基因的可变剪接对湖羊肌肉发育和肉质性状的调控作用[D]. 金华: 浙江师范大学, 2018: 59-62.
[15]周伟. 养殖盐度和密度对斑节对虾肉质及风味的影响[D]. 天津: 天津农学院, 2019: 18-20.
[16]周伟, 王洋, 孙学亮, 等. 养殖密度对斑节对虾肌肉品质的影响[J]. 食品工业科技, 2018, 39(23): 69-75. DOI:10.13386/j.issn1002-0306.2018.23.012.
[17]邝良德, 雷岷, 张翔宇, 等. 基于RNA-Seq转录组测序技术揭示肉兔生长发育调控的分子机制[J]. 基因组学与应用生物学, 2020(2):539-546. DOI:10.13417/j.gab.039.000539.
[18] BEMER M, KARLOVA R, BALLESTER A R, et al. The tomato FRUITFULL homologs TDR4/FUL1 and MBP7/FUL2 regulate ethylene-independent aspects of fruit ripening[J]. Plant Cell, 2012,24(11): 4437-4451. DOI:10.1105/tpc.112.103283.
[19] BROWN R L, KAZAN K, MCGRATH K C, et al. A role for the GCC-box in jasmonate-mediated activation of the PDF1.2 gene of Arabidopsis[J]. Plant Physiology, 2003, 132(2): 1020-1032.DOI:10.1104/pp.102.017814.
[20] HERRERA M I, UDOVIN L D, TORO-URREGO N, et al.Palmitoylethanolamide ameliorates hippocampal damage and behavioral dysfunction after perinatal asphyxia in the immature rat brain[J]. Frontiers in Neuroscience, 2018, 12: 145. DOI:10.3389/fnins.2018.00145.
[21] WANG J, HIDAKA T, SASAKI Y, et al. Neuro filament heavy polypeptide protects against reduction in synaptopodin expression and prevents podocyte detachment[J]. Scientific Reports, 2018, 8(1):17157. DOI:10.1038/s41598-018-35465-6.
[22]SABIR J S M, RABAH S, YACOUB H, et al. Molecular evolution of cytochrome C oxidase-I protein of insects living in Saudi Arabia[J]. PLoS One, 2019, 14(11): e0224336. DOI:10.1371/journal.pone.0224336.
[23] JEOUNG N H, HARRIS C R, HARRIS R A. Regulation of pyruvate metabolism in metabolic-related diseases[J]. Reviews in Endocrine and Metabolic Disorders, 2014, 15(1): 99-110. DOI:10.1007/s11154-013-9284-2.
[24] PATEL M S, NEMERIA N S, FUREY W, et al. The pyruvate dehydrogenase complexes: structure-based function and regulation[J].Journal of Biological Chemistry, 2014, 289: 16615-16623.DOI:10.1074/jbc.R114.563148.
[25] GRAY L R, TOMPKINS S C, TAYLOR E B. Regulation of pyruvate metabolism and human disease[J]. Cellular and Molecular Life Sciences, 2014, 71: 2577-2604. DOI:10.1007/s00018-013-1539-2.
[26] UNVER T, TURKTAS M, BUDAK H. In planta evidence for the involvement of a ubiquitin conjugating enzyme (UBC E2 clade) in negative regulation of disease resistance[J]. Plant Molecular Biology Reporter, 2013, 31(2): 323-334. DOI:10.1007/s11105-012-0502-y.
[27] ZHOU Huajun, COSTAGUTA G, PAYNE G S. Clathrin adaptor complex-interacting protein Irc6 functions through the conserved C-terminal domain[J]. Scientific Reports, 2019, 9(1): 4436.DOI:10.1038/s41598-019-40852-8.
[28] RENIGUNTA V, FISCHER T, ZUZARTE M, et al. Cooperative endocytosis of the endosomal SNARE protein syntaxin-8 and the potassium channel TASK-1[J]. Molecular Biology of the Cell, 2014,25(12): 1877-1891. DOI:10.1091/mbc.E13-10-0592.
[29] KIM D K, KESAWAT M S, HONG C B. One gene member of the ADP-ribosylation factor family is heat-inducible and enhances seed germination in Nicotiana tabacum[J]. Genes and Genomics, 2017,39(12): 1353-1365. DOI:10.1007/s13258-017-0599-3.
[30]倪嘉豪, 朱晓静, 季益平, 等. 养殖密度对银鲳幼鱼生长、代谢酶活力及其相关基因表达的影响[J]. 热带海洋学报. 2020, 39(2): 54-64.DOI:10.11978/2019045.