牦牛是一种珍稀的畜种资源,可适应高海拔、低气压的气候条件,且具有耐寒、耐劳、耐粗饲的优点。苛刻的生存环境造就了牦牛肉特有的品质,牦牛肉色泽深红、富含蛋白质和矿物质,具有低脂肪、高营养、风味独特等优良特性,越来越受到消费者的青睐[1-2]。我国牦牛产区众多,主要分布于青海、四川、甘肃、西藏、云南和新疆等以青藏高原为中心的高原地带[3-5]。各个产地的牦牛经过长期的自然选择和人工选育形成了不同的品种[2,6]。当雄高山牦牛是当地人民经过长达300多年选育出的优良品种,2016年被认定为国家地理标志产品。当雄高山牦牛在生产性能、畜产品品质和遗传稳定性上相比其他牦牛品种具有一定优势[7]。王琳琳等[8]研究发现,当雄县的高山牦牛肉在食用品质上优于麦洼牦牛肉。然而,不同地区牦牛因品牌知名度、市场接受度等不尽相同,导致牦牛肉价格也存在一定差异。低价肉冒充高价肉、普通产品冒充地理标志产品的欺诈行为,不仅侵害了消费者的合法权益,也对当地牦牛产业的发展造成了负面影响[9-11]。因此,为保护牦牛产业的健康发展,亟需开展当雄高山牦牛品种鉴定方法相关研究。
动物源性成分检测技术通常建立在蛋白质、DNA等分子结构、序列或组成特异性分析的基础上[12-13]。相对于蛋白质而言,DNA具有较高的热稳定性和普遍存在性,使得基于DNA序列的动物源性成分鉴别方法得到更加广泛的开发和应用[14-16]。聚合酶链式反应(polymerase chain reaction,PCR)技术具有灵敏度高、特异性强的特点,被认为是此类方法中鉴别动物源性成分的理想工具[17-18],其中实时荧光定量PCR(real-time PCR,RT-PCR)因操作简便、可实时反映目标DNA片段的扩增情况,近些年被大量应用于动物源性成分的定性和定量分析[19-21]。RT-PCR技术主要包括探针法和染料法[22],相比于探针法,染料法更加简单、节约成本[23],而熔解曲线分析是染料法中常用的结果分析手段,通过不同的熔解温度(melting temperature,Tm)可以实现对物种的定性鉴别。凌睿等[24]使用SYBR Green染料法,通过熔解曲线中猪((82.03±0.25) ℃)、羊((78.83±0.16) ℃)扩增产物Tm的不同对以猪肉掺假羊肉的混合物进行检测。Ishida等[25]通过线粒体DNA使用熔解曲线Tm分析法,应用2 个多重RT-PCR反应可鉴别多个动物物种。
目前,有关牦牛肉鉴别的研究主要是针对牦牛与普通牛等其他物种之间的鉴别。段庆梓等[9]利用细胞色素b(cytochrome b,Cyt b)基因设计牦牛特异性引物,对掺有猪肉、水牛肉和黄牛肉混合肉样中的牦牛源性成分进行了有效鉴别。Zhao Jie等[11]利用等位基因特异性PCR技术,通过10 个单核苷酸多态性(single nucleotide polymorphisms,SNP)位点对牦牛肉和普通牛肉进行了区分。而不同品种牦牛肉之间鉴别方法的报道还很少。不同品种的牦牛由于气候、海拔、植被和矿物质等生长环境不尽相同,在长期的自然选择过程中,牦牛的遗传物质DNA会发生不同程度、不同方向的变异,导致不同品种牦牛DNA中的个别碱基存在多态性[26-27]。有研究表明,利用多基因、多SNP位点组合以及多重PCR技术进行物种鉴别可以提高鉴别效率、准确度,节约成本[28-29]。
本研究采集西藏当雄县、青海久治县、青海祁连县、四川红原县和甘肃甘南州5 个地区的牦牛肉样品,通过对线粒体基因进行比对分析,筛选出11 个用于鉴定当雄高山牦牛肉的SNP位点,并以这些SNP位点作为引物3′端,设计出8 对特异性引物,最终建立一种基于SNP位点和多重RT-PCR技术鉴别当雄高山牦牛肉的方法。通过比较3 个反应体系熔解曲线峰的有无及Tm是否符合取值范围,来判定样品是否为当雄高山牦牛肉,对于西藏当雄牦牛的合理开发、利用及地理标志性的保护具有重要意义。
1 材料与方法
1.1 材料与试剂
当雄高山牦牛肉(霖肉,5 岁龄,32 份,1 kg/份)西藏当雄县净土牦牛产业开发有限责任公司;其他地区牦牛肉(5 岁龄,68 份,1 kg/份):青海久治地区(久治牦牛,霖肉,20 份)、青海祁连地区(环湖牦牛,霖肉,14 份)、四川红原地区(麦洼牦牛,脊肉,14 份)和甘肃甘南州地区(安多牦牛,脊肉,20 份);市售当雄牦牛肉(15 份,1 kg/份) 当雄县农贸市场和拉萨市农贸市场。
DNA提取试剂盒 德国Qiagen公司;琼脂糖西班牙Biowest公司;D2000 DNA Marker 天根生化科技有限公司;PrimeSTAR® HS(Premix) 宝生物工程(大连)有限公司;LightCycler® 480 SYBR® Green I Master酶体系预混液 德国Roche公司。
1.2 仪器与设备
Omni Prep多样品匀浆仪 美国Omni公司;NanoDrop One超微量紫外-可见分光光度计 美国Thermo Fisher公司;PCR仪、Biorad GelDoc XR凝胶成像仪美国Bio-Rad公司;LightCycler 480荧光定量PCR仪 罗氏诊断产品(上海)有限公司;5417离心机 艾本德(中国)有限公司;DYCP-31F琼脂糖水平电泳仪北京六一仪器厂;Cascada BIO超纯水系统 美国Pall公司。
1.3 方法
1.3.1 样品DNA的提取
参考Li Jinchun等[30]所述方法进行样品DNA提取。
1.3.2 线粒体基因扩增引物设计
在美国国立生物技术信息中心(NCBI)数据库下载青藏高原牦牛(Bos_grunniens,GenBank:KR011113.1)线粒体基因全序列,通过Oligo 7.0软件设计出6 对用于扩增牦牛线粒体基因(16S rRNA、Cyt b、COⅢ、ND1、ND4、ND5)的引物(表1)。
表1 部分牦牛线粒体基因测序引物
Table 1 Primers used for sequencing yak mtDNA

基因片段 引物名称 序列(5′→3′) 产物大小/bp 16S rRNA YAK-16S-F GTGTGCTTGGATAAATCAAGATAT 1571 YAK-16S-R AGGAGAGGATTTGAATCTCTGGGT Cyt b YAK-Cytb-F GGAATCTAACCATGACCAATGATATG 1140 YAK-Cytb-R AGTCTTAGGGAGGTTGGTTGTTCT COⅢ YAK-Cox3-F TCCAAGCCTATGTATTCACCCT 781 YAK-Cox3-R TACAAGAGATACTGCGAATGCTAT ND1 YAK-ND1-F GCCCGGTAATTGCGTAAAACTT 956 YAK-ND1-R TGGATTCTTAGGAGTAGGTTCAAT ND4 YAK-ND4-F CCTAGGTCTATCCCTACTAGTAAT 1378 YAK-ND4-R TCGGTAAATAAGAAGGTAATGAGT ND5 YAK-ND5-F GGATAGTAGTCATCCGTTGGTCTT 1821 YAK-ND5-R GATTAGTTATTGTAACTGGGTGGT
1.3.3 线粒体基因扩增与测序
根据6 对线粒体基因引物,分别对32 份当雄高山牦牛肉样品和68 份其他地区牦牛肉样品进行PCR扩增。PCR扩增体系(30 μL):Premix 15 μL、上游引物(5 μmol/L)3 μL、下游引物(5 μmol/L)3 μL、模板(5 ng/μL)2 μL,灭菌双蒸水补足体积至30 μL。普通PCR反应条件为:95 ℃预变性5 min,95 ℃变性30 s,60 ℃退火30 s,72 ℃延伸105 s,共计35 个循环。PCR反应结束后,使用1%琼脂糖凝胶在100 V电压条件下进行电泳,PCR产物上样量5 μL,当溴酚蓝移动到距离胶板下沿约1 cm处时停止电泳;电泳结束后在凝胶成像仪上成像,以检测有无PCR扩增产物。扩增产物委托英潍捷基(上海)有限责任公司进行测序。
1.3.4 SNP位点筛选
将不同地区牦牛线粒体基因进行对比分析,筛选SNP位点。SNP位点筛选原则:以32 头当雄高山牦牛保守序列为目标区域,筛选对应区域中其他牦牛线粒体碱基多态性高的位点,用于构建当雄高山牦牛鉴别方法。
1.3.5 鉴定当雄高山牦牛肉的引物设计
将筛选出的SNP位点作为鉴别当雄高山牦牛引物的3′端,将11 个SNP位点(表2)分别设计在8 对引物中并分组到3 个反应体系中,包括2 个三重反应体系(A、B)和1 个二重反应体系(C)。为了提高当雄高山牦牛引物的特异性和抑制非特异性扩增,引物设计过程中引入碱基错配理念[31],在部分引物SNP位点的前一位引入错配碱基(表2):DX-P2-R引物中由碱基T变为A;DX-P4-R引物中碱基A变为C;DX-P5-R引物中碱基G变为T;DX-P7-R引物中碱基A变为T。引物合成委托英潍捷基(上海)有限责任公司进行。
表2 鉴别当雄高山牦牛肉的引物信息
Table 2 Information about primers used for identification of Dangxiong yak meat
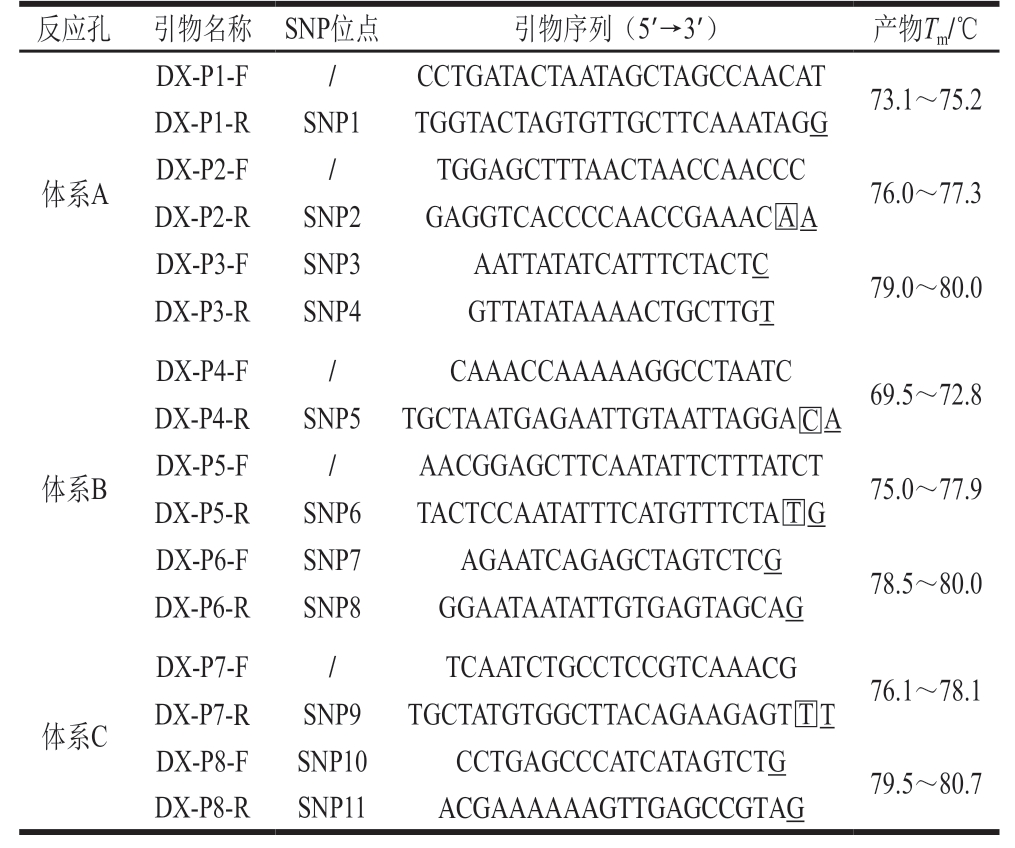
注:标有下划线的碱基为SNP位点;标有边框的碱基为错配碱基;/. 无SNP位点。
反应孔 引物名称 SNP位点 引物序列(5′→3′) 产物Tm/℃体系A DX-P1-F / CCTGATACTAATAGCTAGCCAACAT 73.1~75.2 DX-P1-R SNP1TGGTACTAGTGTTGCTTCAAATAGG DX-P2-F / TGGAGCTTTAACTAACCAACCC 76.0~77.3 DX-P2-R SNP2 GAGGTCACCCCAACCGAAAC A DX-P3-F SNP3 AATTATATCATTTCTACTC 79.0~80.0 DX-P3-R SNP4 GTTATATAAAACTGCTTGT体系B DX-P4-F / CAAACCAAAAAGGCCTAATC 69.5~72.8 DX-P4-R SNP5 TGCTAATGAGAATTGTAATTAGGA A DX-P5-F / AACGGAGCTTCAATATTCTTTATCT 75.0~77.9 DX-P5-R SNP6 TACTCCAATATTTCATGTTTCTA G DX-P6-F SNP7 AGAATCAGAGCTAGTCTCG 78.5~80.0 DX-P6-R SNP8 GGAATAATATTGTGAGTAGCAG体系C DX-P7-F / TCAATCTGCCTCCGTCAAACG 76.1~78.1 DX-P7-R SNP9 TGCTATGTGGCTTACAGAAGAGT T DX-P8-F SNP10 CCTGAGCCCATCATAGTCTG 79.5~80.7 DX-P8-R SNP11ACGAAAAAAGTTGAGCCGTAG
1.3.6 多重RT-PCR反应体系及条件
鉴别当雄地区牦牛肉的反应体系由2 个三重RT-PCR反应和1 个二重RT-PCR反应组成。多重RT-PCR反应体系总量为25 μL,组分及配比如表3所示。多重RT-PCR反应条件为:95 ℃预变性5 min,30 个循环,95 ℃变性10 s,57 ℃退火和延伸45 s;熔解曲线制作过程为:95 ℃、1 min,65 ℃、1 min,以0.02 ℃/s速率升温至95 ℃,同时连续检测荧光强度,最后冷却(40 ℃、1 min)。
表3 多重RT-PCR反应体系组分及配比
Table 3 Ingredients and formulation of multiplex RT-PCR reaction systems
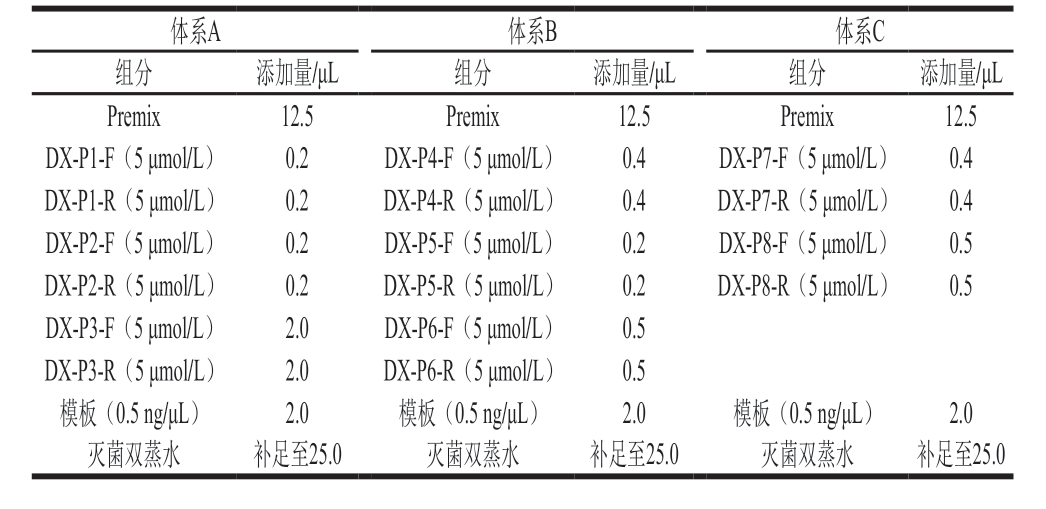
体系A 体系B 体系C组分 添加量/μL 组分 添加量/μL 组分 添加量/μL Premix 12.5 Premix 12.5 Premix 12.5 DX-P1-F(5 μmol/L) 0.2 DX-P4-F(5 μmol/L) 0.4 DX-P7-F(5 μmol/L) 0.4 DX-P1-R(5 μmol/L) 0.2 DX-P4-R(5 μmol/L) 0.4 DX-P7-R(5 μmol/L) 0.4 DX-P2-F(5 μmol/L) 0.2 DX-P5-F(5 μmol/L) 0.2 DX-P8-F(5 μmol/L) 0.5 DX-P2-R(5 μmol/L) 0.2 DX-P5-R(5 μmol/L) 0.2 DX-P8-R(5 μmol/L) 0.5 DX-P3-F(5 μmol/L) 2.0 DX-P6-F(5 μmol/L) 0.5 DX-P3-R(5 μmol/L) 2.0 DX-P6-R(5 μmol/L) 0.5模板(0.5 ng/μL) 2.0 模板(0.5 ng/μL) 2.0 模板(0.5 ng/μL) 2.0灭菌双蒸水 补足至25.0 灭菌双蒸水 补足至25.0 灭菌双蒸水 补足至25.0
1.3.7 多重RT-PCR体系特异性检验
为考察多重RT-PCR体系特异性,分别随机提取西藏当雄地区、青海久治地区、青海祁连地区、四川红原地区和甘肃甘南州地区各3 个牦牛肉样品的DNA,将DNA样品质量浓度均稀释至0.5 ng/μL,作为检测模板。然后同时进行2 个三重和1 个二重RT-PCR熔解曲线分析。通过比较3 个反应体系熔解曲线峰的个数以及Tm是否符合取值范围,来判定多重RT-PCR体系特异性。
1.3.8 多重RT-PCR体系的应用
将15 份于农贸市场采集的当雄牦牛样品按照1.3.1节样品DNA的提取方法进行处理,制备成待测模板,再根据1.3.6节多重RT-PCR反应体系及条件对15 个样品模板进行检测,每个样品进行3 次平行实验。
1.4 数据处理
通过LightCycler® 480软件中Tm Calling分析得到Tm和熔解曲线图,该曲线以Tm为横坐标,荧光信号强度(F)对Tm的一阶负导数(-dF/dTm)为纵坐标。
2 结果与分析
2.1 牦牛线粒体基因扩增结果
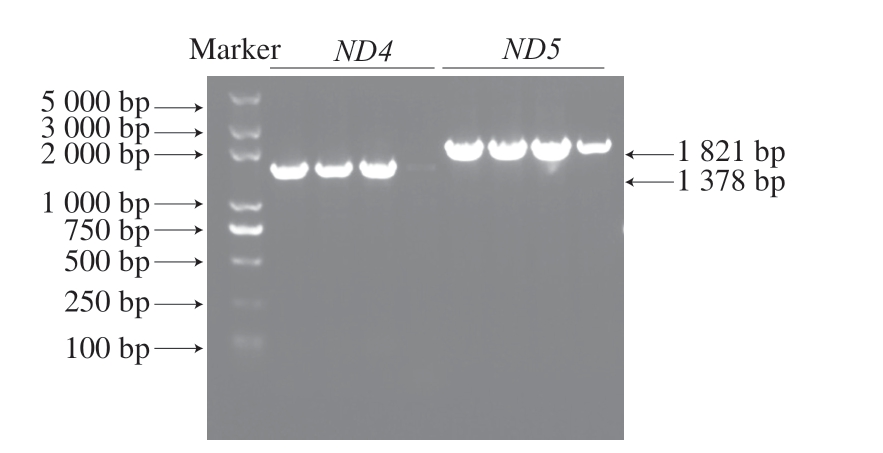
图1 部分牦牛线粒体基因扩增产物琼脂糖凝胶电泳图
Fig. 1 Agarose gel electrophoresis of amplified products of partial yak mitochondrial gene
为验证牦牛线粒体基因测序引物的扩增效果,挑选部分样品的扩增产物进行琼脂糖凝胶电泳实验。由图1可知,ND4和ND5基因扩增产物条带清晰,大小与表1中产物大小一致,未发生非特异性扩增。PCR扩增产物经测序后进行比对分析,用于筛选SNP位点。
2.2 SNP位点筛选和引物设计
以当雄高山牦牛保守序列为目标区域,筛选对应区域中其他牦牛线粒体碱基多态性较高的位点,最终筛选出11 个SNP位点,所有SNP位点分布在16S rRNA、Cyt b、COⅢ、ND1、ND4、ND5共计6 个线粒体基因上,SNP位点物理位置及碱基分布情况见表4,其中物理位置参考青海高原牦牛线粒体基因组序列(GenBank:KR011113.1)确定。将这些SNP位点作为引物3′端,设计出8 对特异性引物,用于构建多重RT-PCR体系。
表4 用于鉴别当雄高山牦牛肉的SNP位点分布情况
Table 4 Distribution of SNP loci for identifying Dangxiong alpine yak meat
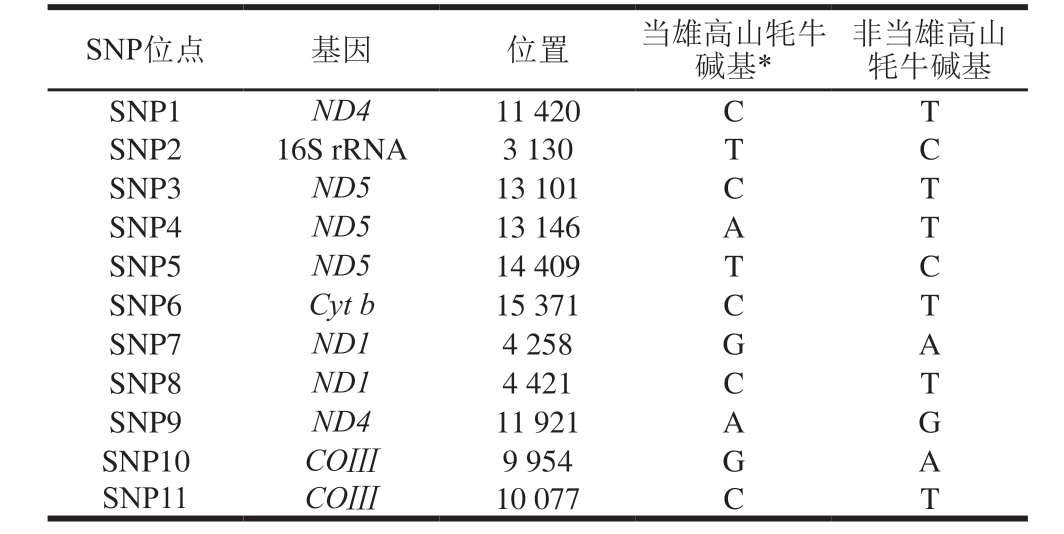
注:*. 此列碱基对应青海高原牦牛线粒体基因组序列(GenBank:KR011113.1),表2中部分SNP位点位于下游引物,因此是该列中对应SNP位点的互补碱基。
SNP位点 基因 位置 当雄高山牦牛碱基*非当雄高山牦牛碱基SNP1 ND4 11420 C T SNP2 16S rRNA 3130 T C SNP3 ND5 13101 C T SNP4 ND5 13146 A T SNP5 ND5 14409 T C SNP6 Cyt b 15371 C T SNP7 ND1 4258 G A SNP8 ND1 4421 C T SNP9 ND4 11921 A G SNP10 COⅢ 9954 G A SNP11 COⅢ 10077 C T
2.3 多重RT-PCR体系构建
在多重RT-PCR体系构建过程中,为避免多重RT-PCR体系中引物之间的交叉反应而导致非特异性扩增,相同或相邻线粒体基因上的引物应当分组到不同RT-PCR反应体系中。另外,为防止熔解曲线峰重叠而无法区分,相邻熔解曲线峰对应的ΔTm应大于其最小限值。Hernandez[32]、Ririe[33]等研究发现,二重RT-PCR中ΔTm为1.5 ℃或2 ℃时,均可通过峰图对2 种扩增产物进行区分。基于以上2 点原因,将8 对引物分组到2 个三重和1 个二重RT-PCR反应体系中(表2)。
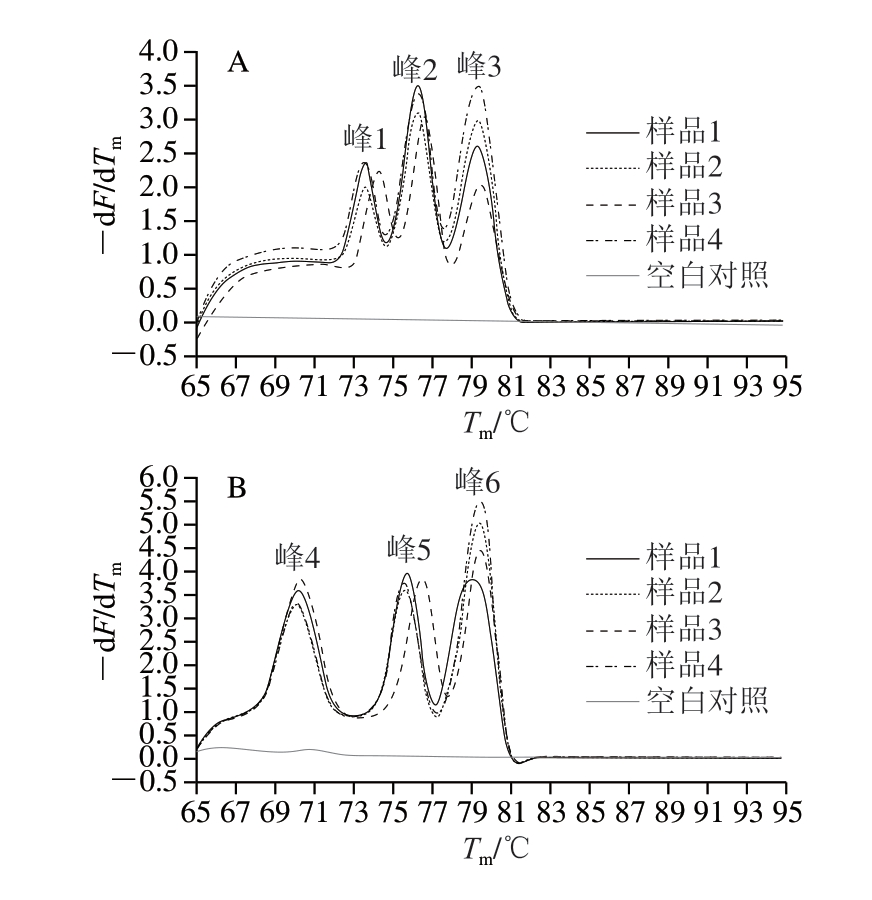

图2 多重RT-PCR反应体系熔解曲线峰图
Fig. 2 Melting peaks of multiplex RT-PCR
以当雄高山牦牛肉为阳性样品,各反应体系中模板为灭菌双蒸水作为空白对照,分别进行3 个多重RT-PCR反应,得到8 个扩增产物的熔解曲线峰图,由图2可知,反应体系A和B均出现3 个峰,反应体系C出现2 个峰。
单重RT-PCR到多重RT-PCR反应过程中,由于引物之间的竞争及引物与模板之间的竞争,相同扩增产物的含量也不尽相同,导致其Tm存在明显差异[34]。统计当雄高山牦牛肉各反应体系中扩增产物Tm的温度范围,反应体系A扩增产物对应的Tm范围分别为73.1~75.2、76.0~77.3、79.0~80.0 ℃;反应体系B扩增产物对应的Tm范围分别为69.5~72.8、75.0~77.9、78.5~80.0 ℃;反应体系C扩增产物对应的Tm范围分别为76.1~78.1、79.5~80.7 ℃。当样品经过上述3 个多重RT-PCR体系出现上述8 个熔解峰,且各Tm均在相应温度范围之内时,则可判定为当雄牦牛肉阳性样品,否则判定为非当雄牦牛肉样品。
2.4 多重RT-PCR体系特异性检验
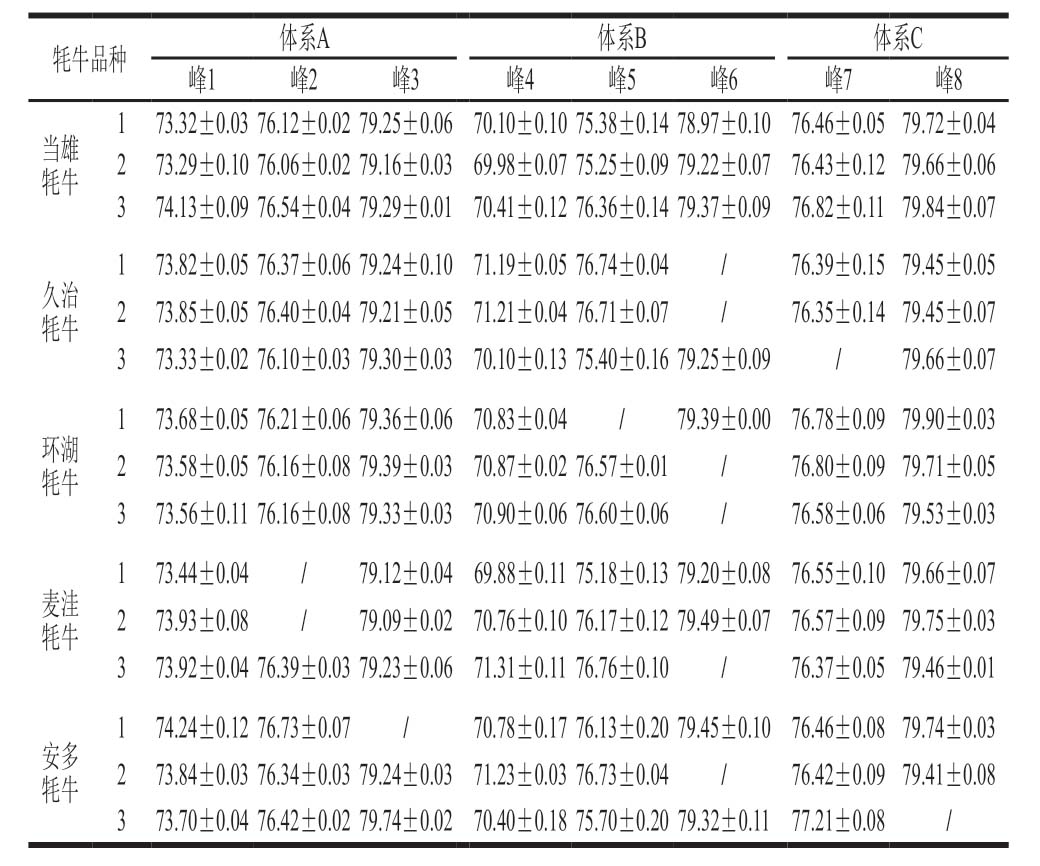
表5 5 个地区牦牛肉在多重RT-PCR反应体系中各产物特异性熔解峰Tm
Table 5Tm of specific melting peaks in multiplex RT-PCR reactions for yaks from five different regions℃
注:/. 未出现熔解峰。表6同。
牦牛品种 体系A 体系B 体系C峰1 峰2 峰3 峰4 峰5 峰6 峰7 峰8当雄牦牛1 73.32±0.0376.12±0.0279.25±0.0670.10±0.1075.38±0.1478.97±0.1076.46±0.0579.72±0.042 73.29±0.1076.06±0.0279.16±0.0369.98±0.0775.25±0.0979.22±0.0776.43±0.1279.66±0.063 74.13±0.0976.54±0.0479.29±0.0170.41±0.1276.36±0.1479.37±0.0976.82±0.1179.84±0.07久治牦牛1 73.82±0.0576.37±0.0679.24±0.1071.19±0.0576.74±0.04 / 76.39±0.1579.45±0.052 73.85±0.0576.40±0.0479.21±0.0571.21±0.0476.71±0.07 / 76.35±0.1479.45±0.073 73.33±0.0276.10±0.0379.30±0.0370.10±0.1375.40±0.1679.25±0.09 / 79.66±0.07环湖牦牛1 73.68±0.0576.21±0.0679.36±0.0670.83±0.04 / 79.39±0.0076.78±0.0979.90±0.032 73.58±0.0576.16±0.0879.39±0.0370.87±0.0276.57±0.01 / 76.80±0.0979.71±0.053 73.56±0.1176.16±0.0879.33±0.0370.90±0.0676.60±0.06 / 76.58±0.0679.53±0.03麦洼牦牛1 73.44±0.04 / 79.12±0.04 69.88±0.1175.18±0.1379.20±0.0876.55±0.1079.66±0.072 73.93±0.08 / 79.09±0.0270.76±0.1076.17±0.1279.49±0.0776.57±0.0979.75±0.033 73.92±0.0476.39±0.0379.23±0.0671.31±0.1176.76±0.10 / 76.37±0.0579.46±0.01安多牦牛1 74.24±0.1276.73±0.07 / 70.78±0.1776.13±0.2079.45±0.1076.46±0.0879.74±0.032 73.84±0.0376.34±0.0379.24±0.0371.23±0.0376.73±0.04 / 76.42±0.0979.41±0.083 73.70±0.0476.42±0.0279.74±0.0270.40±0.1875.70±0.2079.32±0.11 77.21±0.08 /
为验证多重RT-PCR反应体系的特异性,随机选取5 个地区不同品种牦牛肉样品进行验证,由表5可知,所有样品的Tm均在阳性样品Tm范围内,其中当雄牦牛肉样品熔解曲线峰达到8 个,其他4 个地区的非当雄牦牛肉样品熔解曲线峰均少于8 个。例如,久治牦牛肉样品1缺少体系B中的第3个熔解曲线峰(峰6),可推断该样品在SNP7位点的碱基不是G,或SNP8位点的碱基不是C,因此该方法可以达到通过符合Tm范围的熔解曲线峰的有无来判断各SNP位点的基因型,进而判断样品是否为当雄高山牦牛肉的目的。经过验证,当雄高山牦牛肉样品均出现8 个熔解曲线峰,且Tm符合取值范围,而非当雄高山牦牛肉样品熔解曲线峰均少于8 个,说明该多重RT-PCR体系具有良好的特异性。
2.5 市售当雄牦牛肉样品检测
为检验该方法的实际鉴别能力,对从拉萨市和当雄县当地农贸市场购买的15 个当雄牦牛肉样品进行检测。通过分析熔解曲线峰图发现,其中的11 个市售当雄牦牛肉样品均出现8 个熔解曲线峰,Tm范围分别为反应体系A:73.3~74.1、76.2~76.6、79.0~79.6 ℃;反应体系B:70.0~71.0、75.2~76.4、78.9~79.5 ℃;反应体系C:76.4~76.9、79.7~80.0 ℃,均符合判定标准中Tm的范围,因此判定该11 个市售样品为当雄高山牦牛肉。而剩余4 个市售样品缺失反应体系B中Tm 78.5~80.0 ℃的第3 个熔解曲线峰(峰6)(表6),因此判定该4 个样品为非当雄高山牦牛肉。通过此方法验证的市售样品中,有26.67%的当雄牦牛肉并非真实的当雄高山牦牛肉。
表6 部分市售样品熔解峰Tm
Table 6Tm of specific melting peaks for commercial yak meat samples℃

市售样品体系A 体系B 体系C峰1 峰2 峰3 峰4 峰5 峰6 峰7 峰82 73.57±0.0576.16±0.0379.25±0.05 70.95±0.0676.54±0.06 / 76.39±0.1079.41±0.068 73.58±0.0476.22±0.0479.19±0.07 71.01±0.0876.59±0.08 / 76.40±0.0879.41±0.049 73.76±0.0376.26±0.0379.25±0.02 71.19±0.0776.72±0.06 / 76.41±0.0779.42±0.0214 73.65±0.0376.21±0.0279.23±0.07 71.03±0.0876.61±0.07 / 76.44±0.0679.45±0.02
3 结 论
本研究开发了一种基于SNP位点和3 个多重RT-PCR反应体系熔解曲线分析鉴别当雄高山牦牛肉的方法,结果表明:3 个RT-PCR反应体系扩增产物对应的Tm范围分别为反应体系A:73.1~75.2、76.0~77.3、79.0~80.0 ℃,反应体系B:69.5~72.8、75.0~77.9、78.5~80.0 ℃,反应体系C:76.1~78.1、79.5~80.7 ℃,根据3 个反应体系中熔解曲线峰的有无以及Tm是否符合取值范围,来鉴定样品是否为当雄高山牦牛肉。该方法具有良好的特异性,可用于实际样品检测。同时,该方法具有操作简便、成本低、结果直观可靠的优点,有利于推广应用。该方法的建立将对保护当雄高山牦牛作为地理标志性产品、提升品牌价值、促进牦牛产业稳健发展起到重要作用。
[1]辜雪冬, 赵娟红, 孙术国, 等. 不同工艺制备的干牦牛肉的理化特征分析[J]. 食品与发酵工业, 2019, 45(1): 165-170. DOI:10.13995/j.cnki.11-1802/ts.017025.
[2]胡强, 郑玉才, 金素钰, 等. 用通用引物扩增细胞色素b基因进行牦牛肉的鉴定[J]. 食品科学, 2007, 28(11): 297-300.
[3]李爱民, 马云, 蓝贤勇, 等. 牦牛分子标记研究进展[J]. 中国牛业科学, 2011, 37(4): 30-34. DOI:10.3969/j.issn.1001-9111.2011.04.009.
[4]孟庆辉, 陈永杏, 董红敏, 等. 牦牛分布特点及其种群数量[J]. 家畜生态学报, 2017, 38(3): 80-85. DOI:10.3969/j.issn.1673-1182.2017.03.017.
[5]WANG Zhaofeng, SHEN Xin, LIU Bin, et al. Phylogeographical analyses of domestic and wild yaks based on mitochondrial DNA: new data and reappraisal[J]. Journal of Biogeography, 2010, 37(12): 2332-2344. DOI:10.1111/j.1365-2699.2010.02379.x.
[6]郭文场, 张嘉保, 陈树宁. 中国牦牛品种(类群)的遗传资源、生态特性、繁殖、饲牧管理与利用(1)[J]. 特种经济动植物, 2014, 17(3):8-12.
[7]任战军, 常洪, 陈国宏, 等. 西藏当雄牦牛品种资源研究[J]. 西北农业学报, 2002, 11(2): 5-9. DOI:10.7606/j.issn.1004-1389.2002.2.002.
[8]王琳琳, 陈炼红. 红原县麦洼牦牛肉和当雄县高山牦牛肉品质差异性的比较分析[J]. 西南民族大学学报(自然科学版), 2019, 45(5):449-457. DOI:10.11920/xnmdzk.2019.05.002.
[9]段庆梓, 尚柯, 张彪, 等. 肉及肉制品中牦牛源性成分的PCR鉴别[J]. 食品研究与开发, 2017, 38(9): 167-170. DOI:10.3969/j.issn.1005-6521.2017.09.035.
[10] WANG Ping, HU Yue, YANG Hairong, et al. DNA-based authentication method for detection of yak (Bos grunniens) in meat products[J]. Journal of AOAC International, 2013, 96(1): 142-146.DOI:10.5740/jaoacint.11-398.
[11]ZHAO Jie, XU Zhenzhen, CHEN Ailiang, et al. Identification of meat from yak and cattle using SNP markers with integrated allele-specific polymerase chain reaction-capillary electrophoresis method[J]. Meat Science, 2019, 148: 120-126. DOI:10.1016/j.meatsci.2018.08.019.
[12] LI Yingying, ZHANG Yingying, LI Huichen, et al. Simultaneous determination of heat stable peptides for eight animal and plant species in meat products using UPLC-MS/MS method[J]. Food Chemistry,2018, 245: 125-131. DOI:10.1016/j.foodchem.2017.09.066.
[13]HOSSAIN M A M, ALI M E, HAMID S B A, et al. Targeting double genes in multiplex PCR for discriminating bovine, buffalo and porcine materials in food chain[J]. Food Control, 2016, 73: 175-184.DOI:10.1016/j.foodcont.2016.08.008.
[14] BALLIN N Z, VOGENSEN F K, KARLSSON A H. Species determination: can we detect and quantify meat adulteration?[J]. Meat Science, 2009, 83(2): 165-174. DOI:10.1016/j.meatsci.2009.06.003.
[15] 何玮玲. 应用多重PCR及实时荧光PCR方法鉴别食品中肉类成分[D].南京: 南京农业大学, 2013: 2-11.
[16] FARAG M R, ALAGAWANY M, EL-HACK M E A, et al.Identification of different animal species in meat and meat products:trends and advances[J]. Advances in Animal and Veterinary Sciences,2015, 3(6): 334-346. DOI:10.14737/journal.aavs/2015/3.6.334.346.
[17] GIRISH P S, KARABASANAVAR N S. Meat quality analysis:advanced evaluation methods, techniques, and technologies[M].Elsevier Inc., 2020: 133-151. DOI:10.1016/B978-0-12-819233-7.00009-4.
[18] ALIKORD M, MOMTAZ H, KERAMAT J, et al. Species identification and animal authentication in meat products: a review[J].Journal of Food Measurement and Characterization, 2018, 12(1):145-155. DOI:10.1007/s11694-017-9625-z.
[19]KANG T S. Basic principles for developing real-time PCR methods used in food analysis: a review[J]. Trends in Food Science and Technology, 2019, 91: 574-585. DOI:10.1016/j.tifs.2019.07.037.
[20]周彤, 李家鹏, 李金春, 等. 一种基于多重实时荧光聚合酶链式反应熔解曲线分析的肉及肉制品掺假鉴别方法[J]. 食品科学, 2017,38(12): 217-222. DOI:10.7506/spkx1002-6630-201712033.
[21] WANG Wenjun, LIU Jianjian, ZHANG Qingde, et al. Multiplex PCR assay for identification and quantification of bovine and equine in minced meats using novel specific nuclear DNA sequences[J]. Food Control, 2019, 105: 29-37. DOI:10.1016/j.foodcont.2019.05.016.
[22]FARRELL R E, Jr. RNA methodologies[M]. 5th ed. New York:Academic Press, 2017: 283-328. DOI:10.1016/b978-0-12-804678-4.00009-9.
[23] LEVIN R E, EKEZIE F G C, SUN D W. DNA-based technique:polymerase chain reaction (PCR)[M]//SUN Dawen. Modern techniques for food authentication. 2nd ed. New York: Academic Press, 2018: 527-616. DOI:10.1016/b978-0-12-814264-6.00014-1.
[24] 凌睿, 薛建丽, 杨军, 等. Real-Time PCR溶解曲线及Myostatin基因在肉类掺假快速鉴别中的应用[J]. 食品科技, 2013, 38(5): 318-322.
[25] ISHIDA N, SAKURADA M, KUSUNOKI H, et al. Development of a simultaneous identification method for 13 animal species using two multiplex real-time PCR assays and melting curve analysis[J]. Legal Medicine, 2018, 30: 64-71. DOI:10.1016/j.legalmed.2017.11.007.
[26]张小波, 何慧, 吴潇, 等. 基于SNP标记的肉类溯源技术[J]. 肉类研究, 2011, 25(5): 40-45. DOI:10.3969/j.issn.1001-8123.2011.05.010.
[27] 陈晓勇, 向海, BRAHI O H D, 等. 四引物扩增受阻突变体系PCR在绵羊线粒体基因组SNP检测中的应用[J]. 中国草食动物科学, 2013,33(6): 5-8. DOI:10.3969/j.issn.2095-3887.2013.06.001.
[28] 杨倩倩, 刘苏汶, 俞晓平. DNA条形码分析方法研究进展[J]. 应用生态学报, 2018, 29(3): 1006-1014. DOI:10.13287/j.1001-9332.201803.032.
[29] 赵杰. 基于SNP和SSR标记的牛肉产品溯源鉴定技术研究与应用[D].北京: 中国农业科学院, 2018: 1-10.
[30] LI Jinchun, LI Jiapeng, XU Suigen, et al. A rapid and reliable multiplex PCR assay for simultaneous detection of fourteen animal species in two tubes[J]. Food Chemistry, 2019, 295: 395-402. DOI:10.1016/j.foodchem.2019.05.112.
[31]李金春, 李家鹏, 周彤, 等. 引物3'端不同碱基错配情况下实时荧光定量PCR非特异性扩增的发生规律[J]. 食品科学, 2017, 38(10):277-283. DOI:10.7506/spkx1002-6630-201710045.
[32] HERNÁNDEZ M, RODRÍGUEZ-LÁZARO D, ESTEVE T, et al.Development of melting temperature-based SYBR Green I polymerase chain reaction methods for multiplex genetically modified organism detection[J]. Analytical Biochemistry, 2003, 323(2): 164-170.DOI:10.1016/j.ab.2003.07.015.
[33] RIRIE K M, RASMUSSEN R P, WITTWER C T. Product differentiation by analysis of DNA melting curves during the polymerase chain reaction[J]. Analytical Biochemistry, 1997, 245(2):154-160. DOI:10.1006/abio.1996.9916.
[34] LI Jinchun, LI Jiapeng, LIU Ruixi, et al. Identification of eleven meat species in foodstuff by a hexaplex real-time PCR with melting curve analysis[J]. Food Control, 2021, 121: 107599. DOI:10.1016/j.foodcont.2020.107599.