撒坝火腿主产于云南省昆明市禄劝县马鹿塘乡,马鹿塘海拔2750 米,常年平均气温10.4 ℃,属于冷凉高寒山区[1]。当地农民采用本地撒坝猪的后腿,修割成琵琶形状,进行低温冷凉、排血,再将配制好的盐料分3 次上盐、堆码、清表面,挂于通风阴凉处,在微生物协助下经过长达12 个月或以上的自然熟化,成熟后的火腿具有特殊香味且有玫瑰红色切面,是人们佐食的佳肴。近年来由于撒坝猪种稀缺,养殖规模急剧下降,致使撒坝火腿产业出现生产技术创新滞后、质量下降等问题[2]。
在火腿成熟过程中微生物对风味的形成和品质起着重要作用[3]。在没有内源性酶的情况下,微生物可以降解火腿中的蛋白质;另外微生物的群落组成和消长受气候及地域的影响,从而促进火腿品质特征的形成,如云南的宣威火腿、诺邓火腿、鹤庆圆腿、三川火腿和老窝火腿等[4]。宣威火腿独特风味形成的基础与葡萄球菌、微球菌的代谢活动和火腿表面霉菌的生长有关[5]。胡永金等[6]研究发现,三川火腿的优势菌为马尾葡萄球菌和模仿葡萄球菌。张诗意等[7]从贵州威宁火腿中分离得到马胃葡萄球菌、木糖葡萄球菌、乳酸片球菌、变平滑假丝酵母菌和近平滑假丝酵母菌。Alapont等[8]研究表明,青霉属、产黄青霉属、纳尔青霉菌属和枝孢枝霉属等菌株可作为西班牙特鲁火腿的发酵剂。
云南传统发酵肉制品中微生物的测定大多采用经典的分离培养和菌落鉴定方法,或是分离后在显微镜下直接进行形态观察,但是这些方法仅能检测出完整微生物种群的一部分,限制了人们对微生物的认识及利用[9]。近年来,16S rDNA分子生物学技术促进了微生态研究的发展。16S rDNA测序比传统方法具有更高的检测效率,因此在未培养细菌的分类中得到广泛应用[10]。此外,16S rDNA测序技术相对便宜,并且有一套用于数据分析的完善计算工具及数据库,而且16S rDNA基因存在于所有细菌物种中[11],因此被认为是研究细菌多样性的理想方法[12]。郭明亮[13]采用Illumina Miseq高通量测序方法对4 个月和2 年的金华火腿与成品宣威火腿的16S rDNA V3区进行研究,结果表明,葡萄球菌和乳酸菌是主要菌群。党喜军[14]采用纯培养和16S/内部转录间隔区(internal transcribed spacer,ITS)测序对诺邓火腿表面微生物进行研究,结果表明,酵母菌、曲霉菌和葡萄球菌是主要优势菌群。
目前关于撒坝火腿中微生物方面的系统研究还很少,与其他火腿的微生物构成是否相似尚不清楚。基于此,本研究对云南撒坝火腿表面和内部微生物的菌群构相、微生物的丰富程度及多样性进行系统分析,旨在为云南开发利用撒坝火腿微生物、发展火腿文化产业提供理论依据。
1 材料与方法
1.1 材料与试剂
撒坝火腿采购自云南省昆明市禄劝县马鹿塘乡当地农户,为腌制1 年以上的火腿,共3 只。
土壤(HiPure Soil)DNA提取试剂盒 广州美基生物科技有限公司;AxyPrep DNA凝胶提取试剂盒 美国Axygen Biosciences公司;TruSeqTM DNA样品制备试剂盒美国Illumina公司;10×KOD(Kodakara品牌)缓冲液、KOD DNA聚合酶 日本Toyobo生物科技有限公司。
1.2 仪器与设备
NanoPhotometer NP80超微量分光光度计 德国Implen公司;-80 ℃冰箱 合肥美菱股份有限公司;DYY-6C电泳仪 北京六一仪器厂;聚合酶链式反应(polymerase chain reaction,PCR)扩增仪 德国耶拿公司;WD-9413B凝胶成像分析系统 北京六一生物科技有限公司;Multiskan Sky全波长酶标仪 美国Thermo Fisher Scientific公司;ABI StepOnePlus实时PCR系统美国Life Technologies公司;PE250 Illumina平台 广州基迪奥生物科技有限公司。
1.3 方法
1.3.1 样品处理
选择3 只质量相近的撒坝火腿随机编号,并作统一处理,无菌条件下每只火腿以相同的方式在半膜肌表面切厚度0.5 cm、边长4 cm的四方形,相应编号为BM1、BM2、BM3,立即放入已灭菌的冻存管中;在内部切割厚度2 cm、边长3 cm的肉块,相应编号为NB1、NB2、NB3,立即放入已灭菌的冻存管中。表面和内部样品均采集2 份,分别装入无菌密封袋中,贮存于-80 ℃冰箱中,一份用于细菌16S rDNA测序,一份用于真菌ITS测序[15]。
1.3.2 总DNA的提取
在无菌条件下取10 g肉样充分混匀,用HiPure Soil DNA试剂盒按操作指南提取样品总DNA。然后用超微量分光光度计测定DNA浓度和纯度,再用2%琼脂糖凝胶电泳检测DNA提取质量。
1.3.3 PCR扩增
经纯化后的DNA用于细菌16S rDNA和真菌ITS基因扩增。以341F(5′-CCTACGGGNGGCWGCAG-3′)和806R(5′-GGACTACHVGGGTATCTAAT-3′)为引物对细菌V3~V4可变区进行PCR扩增[16];以ITS3-KYO2F(5′-GATGAAGAACGYAGYRAA-3′)和ITS4R(5′-TCCTCCGCTTATTGATATGC-3′)为引物对真菌ITS2区进行PCR扩增[17]。扩增程序:94 ℃、2 min,然后98 ℃、10 s,62 ℃、30 s,68 ℃、30 s进行30 个循环,最后68 ℃、5 min。扩增体系(50 μL):5 μL 10×KOD缓冲液、5 μL 2 mmol/L dNTPs、3 μL 25 mmol/L MgSO4、上下游引物各1.5 μL(10 μmol/L)、1 μL DNA聚合酶、100 ng模板DNA。
1.3.4 Illumina测序
从2%琼脂糖凝胶中搜集扩增子,使用AxyPrep DNA凝胶提取试剂盒按操作说明进行纯化,并使用ABI StepOnePlus实时PCR系统进行定量[13]。纯化后的扩增子根据标准操作在Illumina平台上进行双端测序。
1.4 数据处理
使用Omicsmart动态实时数据分析平台(http://www.omicsmart.com)进行数据分析。利用FASTP软件(版本0.18.0)对原始测序数据进行过滤,使用QIIME软件(版本1.9.1)进行序列拼接。参考数据库(http://drive5.com/uchime/uchime_download.html),使用UCHIME算法[18]进行tag嵌合体检查进行后续分析。使用UPARSE(版本9.2.64)流程将Clean Tag按≥97%相似度聚类为操作分类单元(operational taxonomic unit,OTU),利用RDP软件(版本2.2)进行物种分类注释,比对SILVA数据库[19]和ITS2数据库[20]。使用R语言进行多元统计分析。使用QIIME软件计算α多样性指数。使用Graphpad Prism软件(版本8.0.1)进行双因素方差分析,判定表面和内部真菌与细菌的差异,P<0.05被认为有统计学差异,判断为差异显著;P<0.01判断为差异极显著。
2 结果与分析
2.1 撒坝火腿测序结果
由表1可知,样品表面和内部有效真菌序列和有效细菌序列比较,真菌序列数大于细菌。真菌和细菌平均OTU数量差异极显著(P<0.01),细菌远大于真菌;但表面和内部比较差异不显著,表明细菌多样性高于真菌,菌群种类存在差异。所有样品的真菌和细菌覆盖率均为99%,表明测序深度已足够反映样品所包含的微生物群落。
表1 火腿样品数据统计分析
Table 1 Statistical analysis of sequencing data of bacterial and fungal DAN from ham samples

注:**. 与细菌相比差异极显著(P<0.01)。
样品 平均序列数 平均OTU数 覆盖率/%真菌 细菌 真菌 细菌 真菌 细菌BM 299840 280319 272** 2339 99.97 99.72 NB 301990 289013 352** 2617 99.97 99.75
2.2 撒坝火腿微生物α多样性分析
表2 撒坝火腿中微生物α多样性
Table 2 Alpha diversity of bacteria and fungi in ham samples
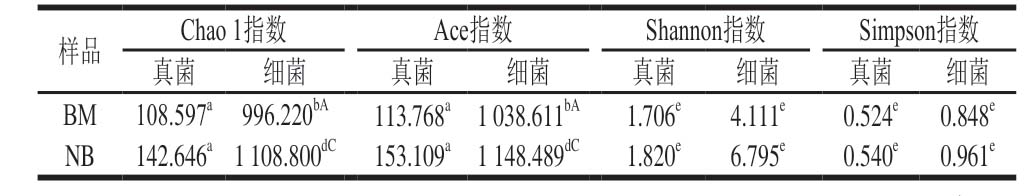
注:同行小写字母不同,表示真菌和细菌间差异极显著(P<0.01);同列大写字母不同,表示表面和内部差异极显著(P<0.01)。
样品 Chao 1指数 Ace指数 Shannon指数 Simpson指数真菌 细菌 真菌 细菌 真菌 细菌 真菌 细菌BM 108.597a 996.220bA 113.768a 1038.611bA 1.706e 4.111e 0.524e 0.848e NB 142.646a1108.800dC 153.109a 1148.489dC 1.820e 6.795e 0.540e 0.961e
微生物群落丰富度与Chao 1指数和Ace指数呈正相关,群落多样性与Shannon指数和Simpson指数呈正相关。由表2可知,样品的真菌丰富度与细菌相比差异极显著(P<0.01),但多样性无显著性差异。表面真菌的丰富度与内部真菌相比差异不显著,表面细菌与内部细菌相比差异极显著(P<0.01);而微生物多样性表面和内部均无显著性差异。
2.3 撒坝火腿微生物β多样性分析
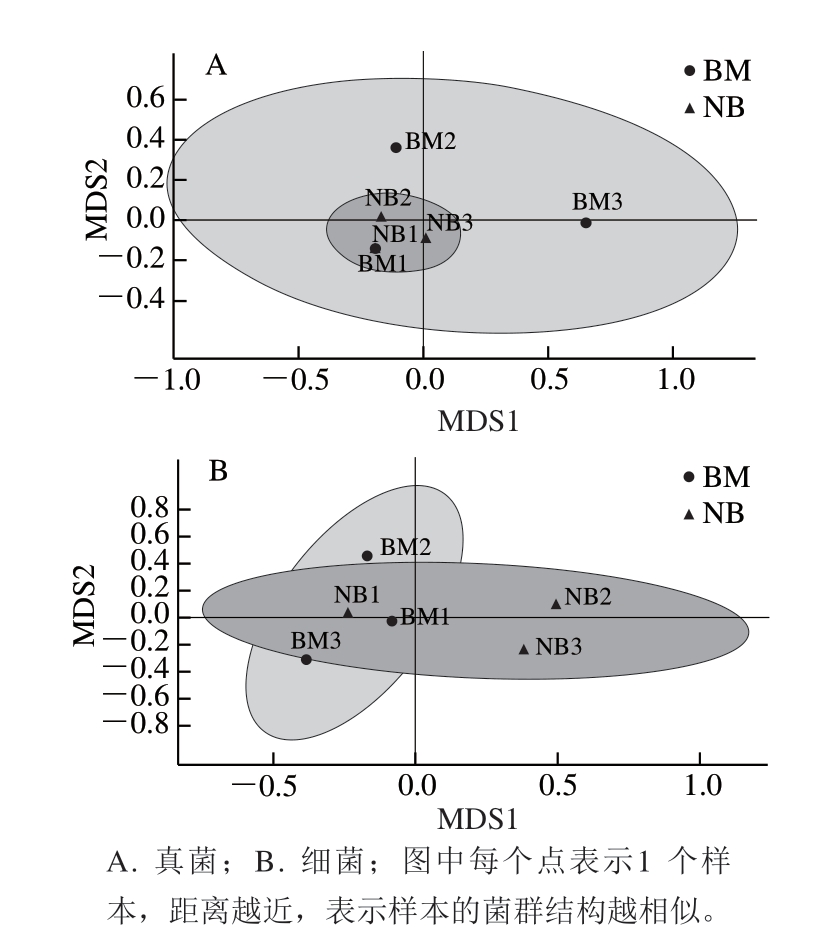
图1 基于OTU水平的NMDS分析
Fig. 1 Nonmetric multi-dimensional scaling analysis based on OTU level
由图1可知,基于真菌和细菌OTU水平的非度量多维尺度(nonmetric multi-dimensional scaling,NMDS)分析,样品真菌和细菌主要集中在2 个区域,但也有交叉。说明样品组间真菌和细菌群落结构和物种多样性差异较大,内部真菌群落结构比较相似,物种多样性差异相对较小;而细菌群落差异较大,物种多样性存在较大差异。
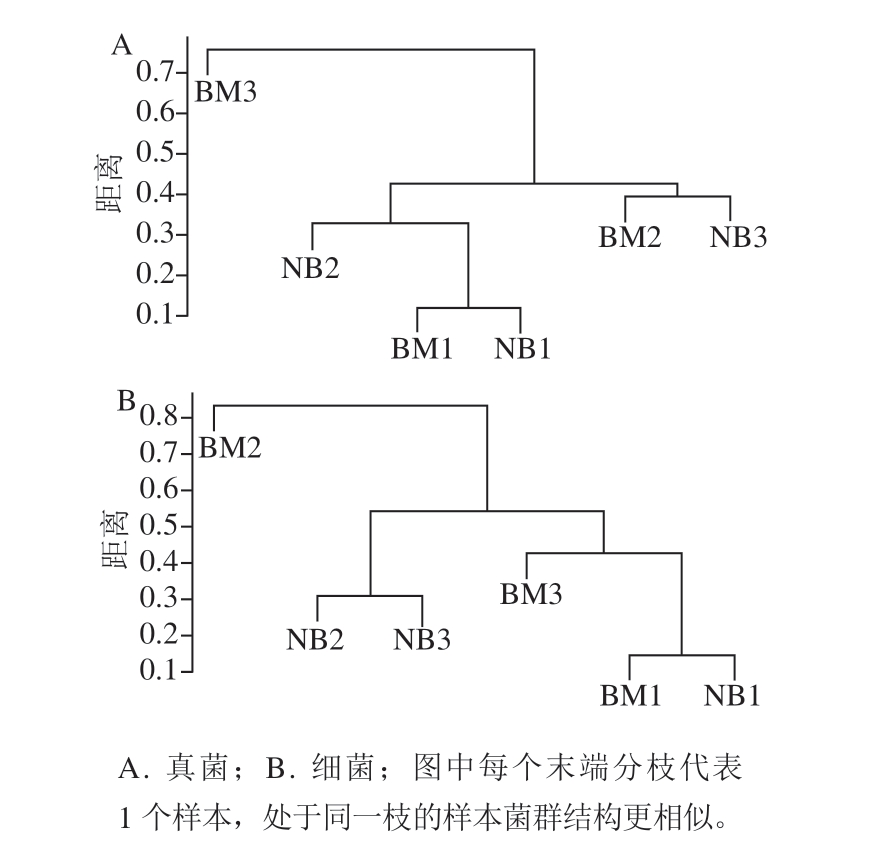
图2 基于OTU水平的非加权组平均聚类树
Fig. 2 Unweighted pair group method with arithmetic mean cluster dendrogram based on OTU level
为了深入了解NMDS结果,对样品进行非加权组平均聚类分析。由图2可知,同一火腿的表面和内部真菌OTU水平相近,内部的细菌群落相似性更高。
2.4 撒坝火腿微生物物种组成分析
2.4.1 真菌物种组成分析
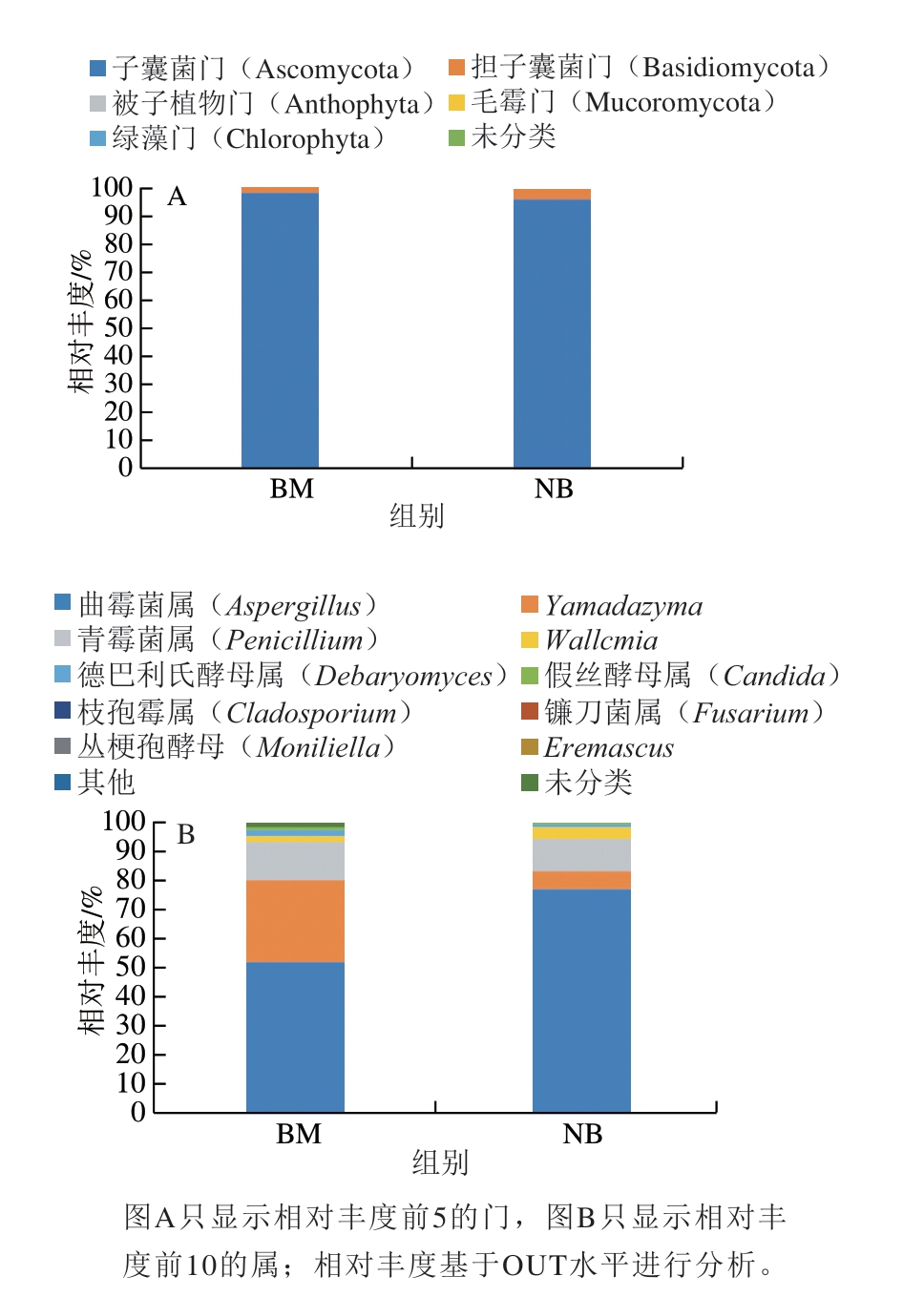
图3 撒坝火腿中真菌门(A)和属(B)水平分布图
Fig. 3 Phylum- (A) and genus-level (B) distribution of fungal community composition in ham samples
由图3可知,在门水平上,子囊菌门是撒坝火腿表面和内部的优势菌门,相对丰度分别约为98.4%和96.0%。在属水平上,曲霉菌属是撒坝火腿表面的优势菌群,相对丰度52.40%,其次是Yamadazyma(27.88%)和青霉菌属(13.22%)。在撒坝火腿内部,曲霉菌属(77.13%)为优势菌群,其次是青霉菌属(11.23%),但Yamadazyma和青霉菌属相对丰度均低于火腿表面。可见表面和内部优势微生物群落存在一定差异,但曲霉菌属均为主要优势菌群。
2.4.2 细菌物种组成分析
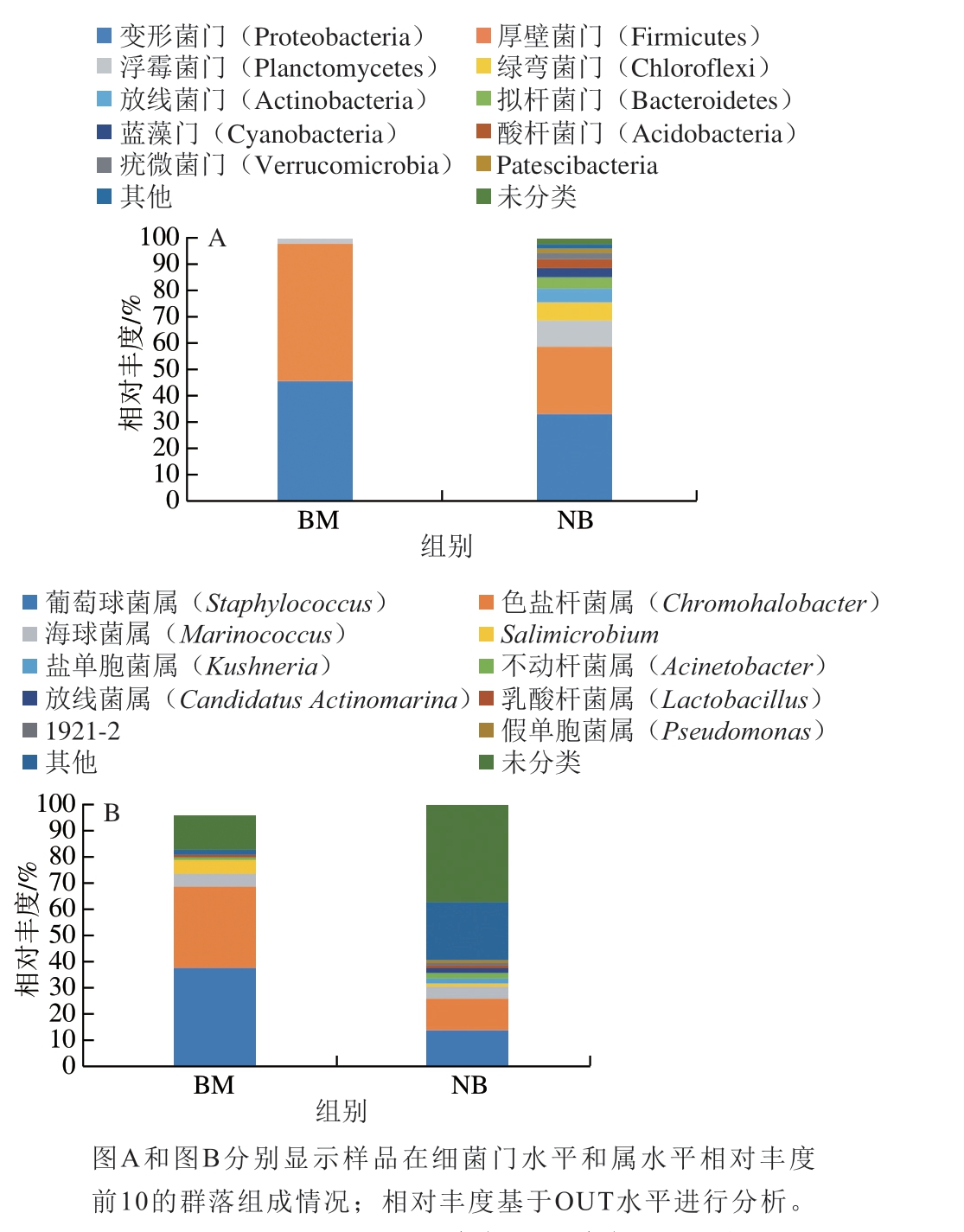
图4 撒坝火腿中细菌门(A)和属(B)水平分布图
Fig. 4 Phylum- (A) and genus-level (B) distribution of bacterial community composition in ham samples
由图4可知,在门水平上,厚壁菌门是撒坝火腿表面的优势菌群,其次是变形菌门,相对丰度分别约为51.79%和45.88%。在撒坝火腿内部,变形菌门(33.53%)是主要优势菌群,其次是厚壁菌门(25.43%)和浮霉菌门(Planctomycetes,9.77%),此外,扁平菌门、绿弯菌门、放线杆菌门和拟杆菌门等相对丰度均高于表面。在属水平上,葡萄球菌和色盐杆菌是撒坝火腿表面的优势菌群,相对丰度分别约为38.38%和31.48%。在撒坝火腿内部,未分类和其他细菌相对丰度分别约为36.56%和21.95%,其次是葡萄球菌(14.38%)和色盐杆菌(12.00%)。可见撒坝火腿内部存在大量未知微生物,表面和内部的优势微生物也存在一定差异,但葡萄球菌均为主要优势菌群。
2.5 撒坝火腿微生物指示物种LefSe分析
2.5.1 真菌指示物种LefSe分析
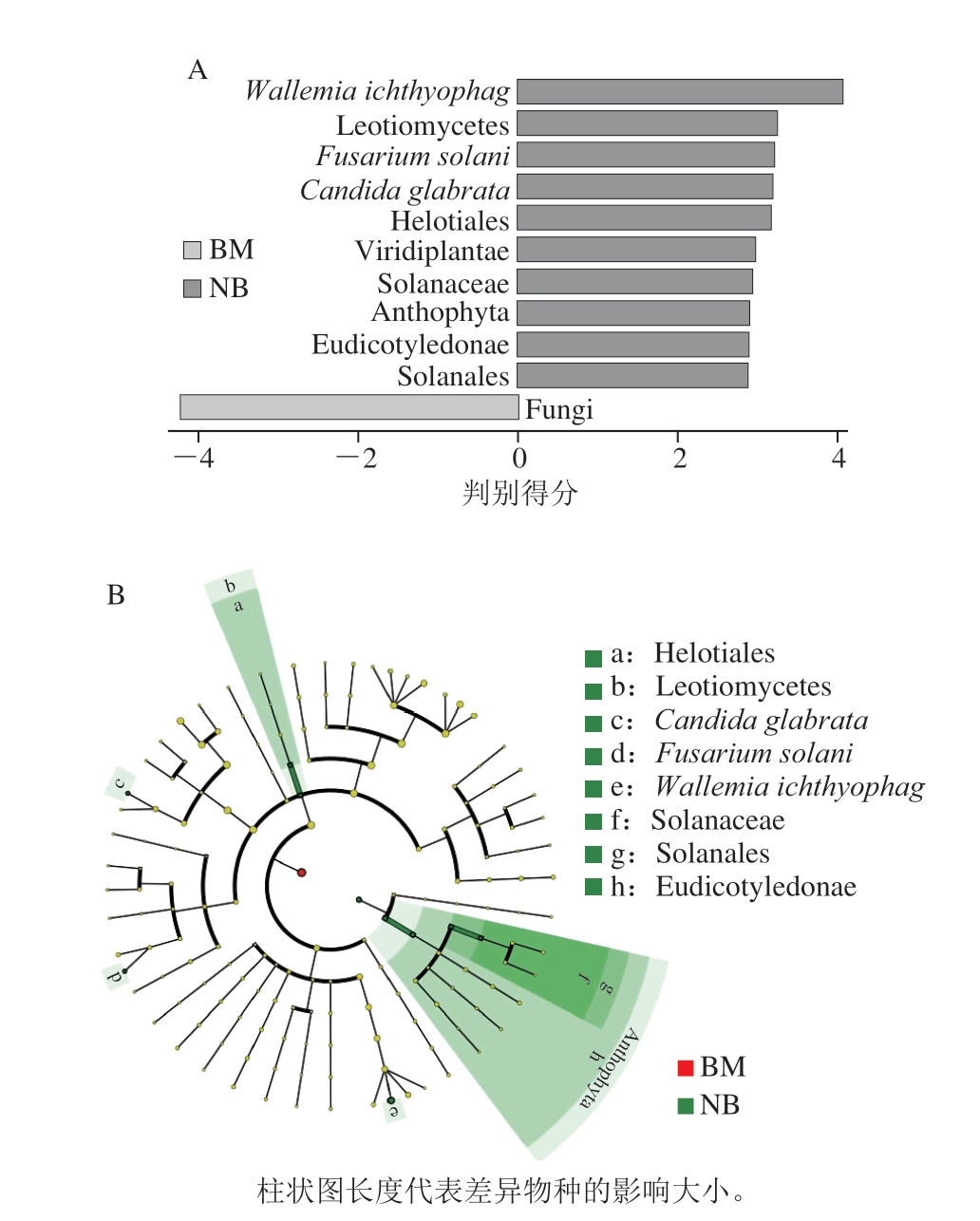
图5 撒坝火腿中真菌LefSe分析
Fig. 5 LefSe analysis of fungal communities in Ham samples
通过LefSe分析组间菌群差异,可以找出组间特异的主要菌群。由图5A可知,撒坝火腿表面和内部样本中真菌共有11 个分类单元(2 界、1 门、2 纲、2 目、1 科、3 种),其中表面样本只注释1 个分类单元真菌界(Fungi),其余10 个分类单元在内部样本均注释。由图5B可知,在内部主要有担子菌门下Wallemia ichthyophag、子囊菌门下腐皮镰孢菌(Fusarium solani)、锤舌菌纲(Leotiomycetes)和光滑假丝酵母(Candida glabrata)等5 类和被子植物门下柔膜菌目(Helotial)等2 类及其他种类,表明撒坝火腿内部真菌存在显著差异的物种最多,子囊菌门占主体,其次是Wallemia ichthyophaga,此类真菌只有在低水分活度下才能生长,是迄今为止最嗜盐的真菌之一[21],而撒坝火腿表面微生物只集中在真菌界。
2.5.2 细菌指示物种LefSe分析
由图6A可知,撒坝火腿表面和内部样本中细菌共有53 个分类单元(2 界、3 门、4 纲、9 目、13 科、17 属、5 种),其中表面样本注释11 个分类单元,内部样本注释42 个分类单元。由图6B可知,撒坝火腿表面细菌主要为厚壁菌门下光杆菌属(Mitsuokella)、葡萄球菌属(Staphylococcus)和链球菌属(Streptococcus)等5 类;内部主要为变形菌门下Planktomarina、鞘脂菌属(Sphingobium)、Bacteria p25和柄杆菌科(Caulobacteraceae)等12 类;其次表面还有放线菌门下奇异菌属(Atopobiaceae)、红蝽菌纲(Coriobacteriia)和诺卡氏菌(Nocardioides)3 类和变形菌门下嗜冷杆菌属(Psychrobacter)等2 类及其他1 类;而内部有放线菌门下放线菌纲(Actinobacteria)和酸微菌纲(Acidimicrobiia)等11 类、疣微菌门下丰祐菌目(Opitutales)和紫红球菌科(Puniceicoccaceae)等5 类、芽单胞菌门下芽单胞菌科(Gemmatimonadaceae)等3 类、拟杆菌门下腐螺旋菌科(Saprospiraceae)1 类和其他8 类。表明撒坝火腿内部细菌存在显著差异的物种最多,主要集中在变形菌门和放线菌门下Planktomarina和鞘脂菌属及放线菌门下放线菌属和酸微菌纲。
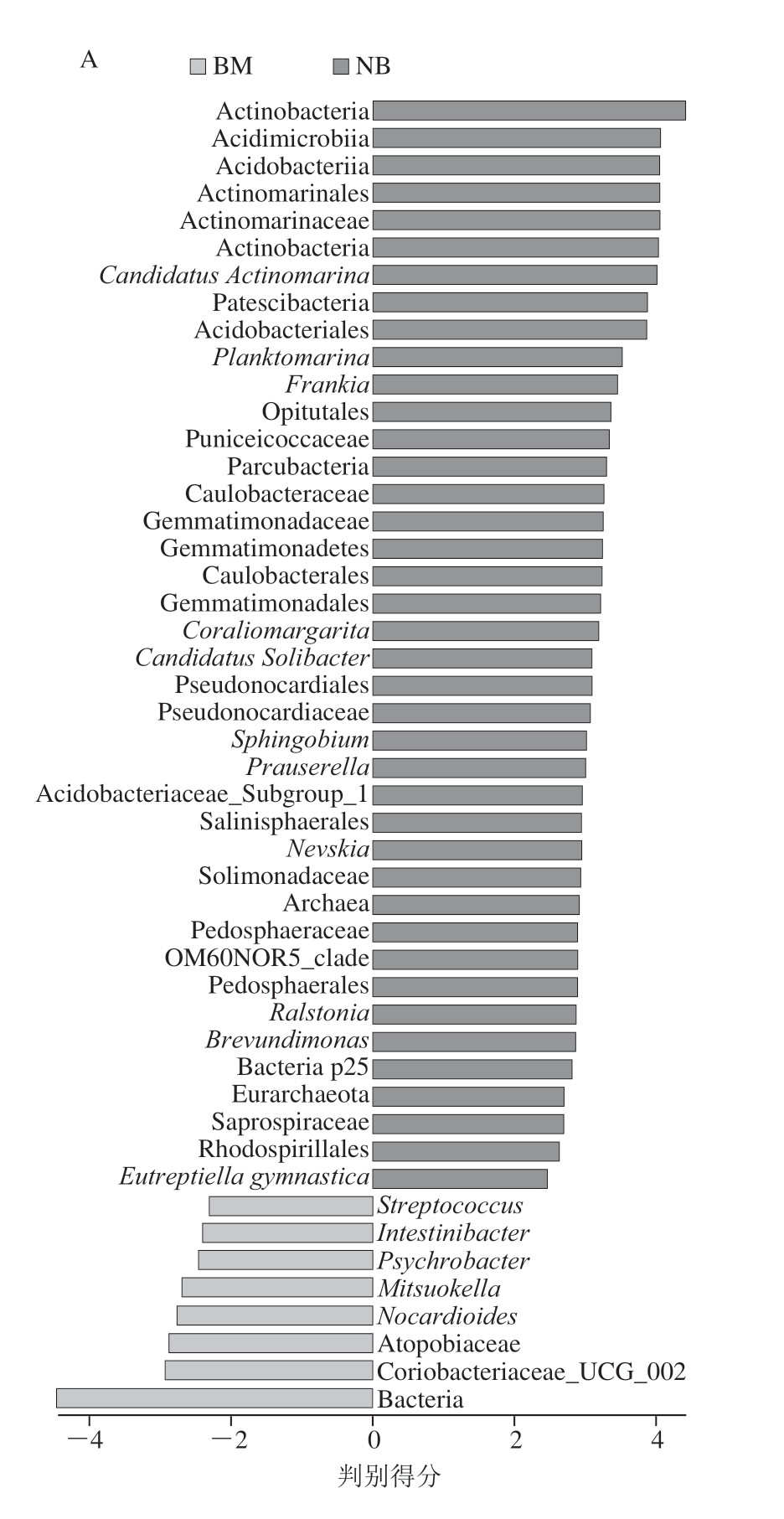

图6 撒坝火腿中细菌LefSe分析
Fig. 6 LefSe analysis of bacterial communities in ham samples
3 讨 论
目前采用高通量测序方法检测发酵肉制品中微生物的研究较多,主要集中在香肠和火腿的微生物区系及成熟过程中微生物种群的动态变化研究等方面。在本研究中,采用16S rDNA和ITS测序方法对撒坝火腿表面和内部微生物进行研究,结果表明,细菌多样性和丰富度高于真菌,且内部微生物较为丰富,与母雨等[22]采用Illumina高通量测序方法研究盘县火腿表明细菌多样性和丰富度高于真菌相似。
微生物菌群在干腌火腿加工过程中起着极其重要的作用。大量研究表明,霉菌、酵母菌、葡萄球菌、微球菌和乳酸菌等是干腌火腿的优势微生物[23]。黄占旺等[24]发现,霉菌和酵母菌是安福火腿表层的主要微生物,酵母菌和球菌则是内部的主要微生物。Battilani等[25]从意大利Parma火腿表面检测出曲霉菌和青霉菌,青霉菌占大多数,其中纳地青霉占青霉属总量的60%。在本研究中,曲霉菌是撒坝火腿表面和内部真菌中的优势菌群,Yamadazyma和青霉菌也是表面的主要菌群。曲霉菌在我国金华火腿、诺邓火腿和盘县火腿及意大利San Daniele火腿中同样为优势真菌[14,22,26-27],具有隔绝空气、抑制有害菌生长等多种功能[28]。Cebrián等[29]研究表明,青霉菌和酵母菌联合使用能防治干腌火腿黄曲霉素的危害。葡萄球菌是撒坝火腿表面和内部细菌中的优势菌群,但内部存在许多未知的微生物。其次,酸杆菌和链球菌是撒坝火腿表面微生物的主要差异菌群,放线菌属、杆菌属、酸微菌纲及球菌属则是内部的主要差异菌群。其中,葡萄球菌属在我国宣威火腿、剑门火腿和三川火腿及西班牙火腿中同样为优势菌群[6,13,30-31],具有较高的蛋白酶和脂肪酶活性,在维持火腿颜色、品质和抑制不良气味产生方面起着重要作用[32]。Mu Yu等[33]研究表明,葡萄球菌属、嗜色杆菌属、曲霉属和青霉菌属等对盘县火腿的感官品质和安全性至关重要。说明不同工艺和环境会导致不同火腿中的优势微生物不尽相同,这也促使不同种类火腿品质各具特色。
4 结 论
本研究对云南撒坝火腿表面和内部微生物多样性进行分析,结果表明,微生物菌群种类存在差异,细菌多样性和丰富度高于真菌,内部的微生物组成相比表面较为丰富,曲霉菌和葡萄球菌是表面和内部的优势菌群。本研究结果为撒坝火腿的风味结构、发酵菌群的变化、病原菌的存在及微生物安全性提供了理论依据。
[1]王燕, 吉杰丽, 朱仁俊. 撒坝火腿成熟过程中游离氨基酸的变化研究[J]. 现代食品科技, 2013, 29(5): 1010-1013. DOI:10.13982/j.mfst.1673-9078.2013.05.036.
[2]朱春贤, 赵桂英, 俞英. 提升撒坝火腿产业水平的意义和措施[J]. 当代畜牧, 2016(10): 71-77.
[3]黄盼盼, 蒋先芝, 田建卿. 火腿微生物研究进展[J]. 生物工程学报,2018, 34(9): 1410-1418. DOI:10.13345/j.cjb.170496.
[4]SHAN Luying, LI Yinjiao, ZHANG Shi, et al. Analysis of the bacterial floral structure and diversity of Xuanwei ham by 16S rDNA sequencing[J]. Journal of Food Safety, 2020, 40: e12800. DOI:10.1111/jfs.12800.
[5]李平兰, 沈清武, 吕燕妮, 等. 宣威火腿成熟产品中主要微生物菌相构成分析[J]. 中国微生态学杂志, 2003, 15(5): 12-13. DOI:10.13381/j.cnki.cj m.2003.05.007.
[6]胡永金, 陈红, 薛桥丽, 等. 云南三川火腿加工中微生物区系变化规律研究[J]. 轻工学报, 2017, 32(5): 8-15. DOI:10.3969/j.issn.2096-1553.2017.5.002.
[7]张诗意, 唐楠, 黄攀, 等. 威宁火腿中微生物的分离鉴定及其耐受特性[J]. 肉类研究, 2019, 33(8): 12-17. DOI:10.7506/rlyj1001-8123-20190625-147.
[8]ALAPONT C, MARTÍNEZ-CULEBRAS P V, LÓPEZ-MENDOZA M C.Determination of lipolytic and proteolytic activities of mycoflora isolated from dry-cured teruel ham[J]. Journal of Food Science and Technology,2015, 52(8): 5250-5256. DOI:10.1007/s13197-014-1582-5.
[9]王燕, 朱仁俊. PCR-DGGE技术及其在云南传统火腿微生物研究中的应用展望[J]. 肉类工业, 2012(9): 50-52.
[10]曹荣, 张井, 孟辉辉, 等. 高通量测序与传统纯培养方法在牡蛎微生物群落分析中的应用对比[J]. 食品科学, 2016, 37(24): 137-141.DOI:10.7506/spkx1002-6630-201624021.
[11] DE FILIPPIS F, PARENTE E, ERCOLINI D. Metagenomics insights into food fermentations[J]. Microbial Biotechnology, 2017, 10(1):91-102. DOI:10.1111/1751-7915.12421.
[12] KUCZYNSKI J, LAUBER C, WALTERS W, et al. Experimental and analytical tools for studying the human microbiome[J]. Nature Reviews Genetics, 2012, 13: 47-58. DOI:10.1038/nrg3129.
[13]郭明亮. 火腿菌群分析及优良菌株对发酵香肠中菌群构成的影响[D].扬州: 扬州大学, 2015: 1-3.
[14]党喜军. 诺邓火腿化学成分测定及表面微生物多样性研究[D]. 昆明:云南大学, 2018: 1-2.
[15]中华人民共和国卫生部, 中国国家标准化管理委员会. 食品卫生微生物学检验 肉与肉制品检验: GB/T 4789.17—2003[S]. 北京: 中国标准出版社, 2003.
[16] GUO Mengjiao, WU Fahao, HAO Guangen, et al. Bacillus subtilis improves immunity and disease resistance in rabbits[J]. Frontiers in Immunology, 2017, 8(2): 354. DOI:10.3389/fimmu.2017.00354.
[17] TOJU H, TANABE A S, YAMAMOTO S, et al. High-coverage ITS primers for the DNA-based identification of ascomycetes and basidiomycetes in environmental samples[J]. PLoS One, 2012, 7(7):e40863. DOI:10.1371/journal.pone.0040863.
[18] EDGAR R C, HAAS B J, CLEMENTE J C, et al. UCHIME improves sensitivity and speed of chimera detection[J]. Bioinformatics, 2011,27(16): 2194-2200. DOI:10.1093/bioinformatics/btr381.
[19] PRUESSE E, QUAST C, KNITTEL K, et al. SILVA: a comprehensive online resource for quality checked and aligned ribosomal RNA sequence data compatible with ARB[J]. Nucleic Acids Research, 2007,35(21): 7188-7196. DOI:10.1093/nar/gkm864.
[20] DESANTIS T Z, HUGENHOLTZ P, LARSEN N, et al. Greengenes, a chimera-checked 16S rRNA gene database and workbench compatible with ARB[J]. Applied and Environmental Microbiology, 2006, 72(7):5069-5072. DOI:10.1128/AEM.03006-05.
[21]GOSTINAR C, SUN Xiaohuan, ZAJC J, et al. Population genomics of an obligately halophilic basidiomycete Wallemia ichthyophaga[J].Frontiers in Microbiology, 2019, 10: 1-12. DOI:10.3389/fmicb.2019.02019.
[22]母雨, 苏伟, 母应春. 盘县火腿微生物多样性及主体挥发性风味解析[J]. 食品研究与开发, 2019, 40(15): 77-85. DOI:10.12161/j.issn.1005-6521.2019.15.013.
[23] 王磊. 干腌火腿生产中微生物作用研究进展[J]. 肉类研究, 2008,22(10): 16-18.
[24] 黄占旺, 徐明生, 汤凯洁, 等. 安福火腿发酵微生物分离与发酵工艺研究[J]. 江西农业大学学报(自然科学), 2003, 25(4): 635-638.DOI:10.13836/j.jjau.2003145.
[25]BATTILANI P, PIETRI A, GIORNI P, et al. Penicillium populations in dry-cured ham manufacturing plants[J]. Journal of Food Protection,2007, 70: 975-980. DOI:10.4315/0362-028X-70.4.975.
[26]贺稚非, 甄宗圆, 李洪军, 等. 金华火腿发酵过程中微生物区系研究[J].食品科学, 2008, 29(1): 190-195.
[27]COMI G, IACUMIN L. Ecology of moulds during the pre-ripening and ripening of San Daniele dry cured ham[J]. Food Research International,2013, 54(1): 1113-1119. DOI:10.1016/j.foodres.2013.01.031.
[28] NUNES C, COIMBRA M A, SARAIVA J, et al. Study of the volatile components of a candied plum and estimation of their contribution to the aroma[J]. Food Chemistry, 2008, 111(4): 897-905. DOI:10.1016/j.foodchem.2008.05.003.
[29] CEBRIÁN E, RODRÍGUEZ M, PEROMINGO B, et al. Efficacy of the combined protective cultures of Penicillium chrysogenum and Debaryomyces hansenii for the control of ochratoxin A hazards in drycured ham[J]. Toxins, 2019, 11(12): 710. DOI:10.3390/toxins11120710.
[30]李欣蔚, 迟原龙, 贾冬英, 等. 剑门火腿菌相构成及主要腐败菌特性分析[J]. 中国酿造, 2012, 31(11): 132-134. DOI:10.3969/j.issn.0254-5071.2012.11.035.
[31] CORDERO M R, ZUMALACARREGUI J M. Characterization of Micrococcaceae isolated from salt used for Spanish dry-cured ham[J].Letters in Applied Microbiology, 2010, 31(4): 303-306. DOI:10.1046/j.1472-765x.2000.00818.x.
[32]王桥美, 杨瑞娟, 严亮. 微生物多样性与宣威火腿品质关系的研究进展[J]. 食品安全导刊, 2016(33): 137-139. DOI:10.16043/j.cnki.cfs.2016.33.102.
[33]MU Yu, SU Wei, MU Yingchun, et al. Combined application of highthroughput sequencing and metabolomics reveals metabolically active microorganisms during Panxian ham processing[J]. Frontiers in Microbiology, 2020, 10: 3012. DOI:10.3389/fmicb.2019.03012.