头足类(Cephalopoda)为软体动物门(Mollusca)的重要类群,现存总类800余种[1],是海洋中最大的食物资源之一。头足类水产品一直以来都属于全世界重点开发的海洋渔业资源,近几十年来,世界头足类捕捞量在海洋捕捞渔业中的份额呈现持续增长趋势[2]。头足类海产品数量庞大,种类丰富,目前已经进行商业性开发的主要有柔鱼类(Ommastrephidae)、枪乌贼类(Loliginidae)、乌贼类(Sepiidae)和章鱼类(Octopodidae)等,其中,柔鱼类的开发比重最大[3]。柔鱼类生长周期短,味道鲜美,富含蛋白质和多种人体必需氨基酸,营养价值很高[4],可食用部分可达80%以上,素有“穷人的鲍鱼”之称。
茎柔鱼(Dosidicus gigas)亦称“秘鲁茎柔鱼”,柔鱼科(Ommastrephidae)、茎柔鱼属(Dosidicus)[5]。广泛分布于中部太平洋以东海域,尤其在秘鲁沿岸及外海资源丰富[6-7]。秘鲁茎柔鱼生长速度快、体型较大、世界范围内产量较高,是全球最重要的经济头足类资源之一[8]。据联合国粮食及农业组织(Food and Agriculture Organization,FAO)统计,2003—2012年世界秘鲁茎柔鱼的年平均产量达到77.84 万t[9]。澳洲双柔鱼(Nototodarus gouldi)亦称“澳大利亚鱿”,只分布于澳大利亚东南部和西部海域以及新西兰海域[10],目前开发力度不大,年最高产量不足万吨,因此价值较高。
秘鲁茎柔鱼营养丰富,但是由于体内非蛋白氮(nonprotein nitrogen,NPN)和某些游离氨基酸的存在,会散发出氨的气味,且肉质偏苦[11]。因此,秘鲁茎柔鱼的市场价值远远低于相对稀少的澳洲双柔鱼。往往会有不法商贩为了追求更高的经济利益,而将秘鲁茎柔鱼假冒或掺入澳洲双柔鱼中进行市场销售。传统的柔鱼鉴定方式主要是通过形态学特征,如大小的差异、形状的不同及色泽的多变来加以区分和鉴定[12-13],但在不同环境生长、处于不同发育阶段、经历不同加工方式的同种柔鱼之间,外观形态也会产生很大差异。并且为了保证远洋柔鱼的鲜度,一般制成冻品或半成品保存[14],或是通过加工成柔鱼丁、柔鱼圈、柔鱼丝和烤柔鱼片等柔鱼制品进行市场流通[15]。形态学鉴定适合整条鱼的物种鉴定,一旦进行了切片、绞碎等工艺,形态学特征彻底缺失以后,柔鱼的鉴定将变得异常困难[16]。因此,寻求一种高效、快速鉴别澳洲双柔鱼和秘鲁茎柔鱼的方法显得尤为重要。
DNA分子标记技术由于具有特异性好、灵敏度高、操作简单且重复性好等优势,已被越来越多地运用到物种鉴定中,它能够反映不同物种个体或种群之间基于基因组的某些差异特征,是DNA分子水平上遗传多样性的直接反映[17-18]。在鱼类基因组序列中,核基因和线粒体基因为常用的两大类分子标记,但由于核基因中存在大量内含子等非编码序列,因此很难找到合适的分子来进行标记。线粒体基因组作为独立存在于高等动物细胞核外唯一的遗传物质,基因排列结构紧密,进化速度快,严格母系遗传且在细胞质中大量存在,相对简单且适合运用于DNA分子标记生物学研究。本研究通过聚合酶链式反应(polymerase chain reaction,PCR)技术扩增秘鲁茎柔鱼和澳洲双柔鱼的线粒体16S rRNA基因片段,并进行测序和分析,根据2 种柔鱼品种测序结果序列之间单核苷酸多态性(single nucleotide polymorphism,SNP)位点的差异,设计品种特异性引物,从而通过多重PCR体系对2 种柔鱼单独样品和混合样品进行区分和鉴定。
1 材料与方法
1.1 材料与试剂
秘鲁茎柔鱼和澳洲双柔鱼由某公司提供,产地分别为秘鲁和澳大利亚。
海洋动物组织基因组DNA提取试剂盒(DP324-02)天根生化科技(北京)有限公司;2×Premix DNA polymerase、Agarose、100 bp PlusII DNA Ladder(BM321)、双蒸水 北京全式金生物技术有限公司;无水乙醇(分析纯) 天津市富宇精细化工有限公司。
1.2 仪器与设备
TG18W台式高速微量离心机 长沙湘智离心机仪器公司;T100TM PCR仪 美国Bio-Rad公司;DYY-10C电泳仪 北京六一仪器厂;Microchemi 4.2凝胶成像系统 以色列DNR公司。
1.3 方法
1.3.1 基因组DNA的提取
以所选秘鲁茎柔鱼和澳洲双柔鱼的冷冻样品作为原料,剪取肌肉组织保存于95%乙醇中。取保存样品肌肉组织30 mg左右,剪碎后置于已灭菌的研钵内,混合液氮研磨至粉末状,采用海洋动物组织基因组DNA提取试剂盒进行提取,所得基因组DNA于-20 ℃条件下保存备用。
1.3.2 PCR扩增与测序
对于所提取2 种柔鱼基因组DNA片段,针对海洋动物线粒体16S rRNA基因序列,使用16S rRNA通用引物进行PCR扩增[19]。用于扩增线粒体16S rRNA基因的通用引物由英潍捷基(上海)贸易有限公司合成,序列分别为16SF:5’-CGCCTGTTTAACAAAAACAT-3’和16SR:5’-CCGGTCTGAACTCAGATCACGT-3’。
PCR反应总体系为20 μL:模板DNA 10 ng、2×Premix DNA polymerase 10 μL、正向及反向引物各2 μL(0.5 μmol/L),加灭菌双蒸水至20 μL。PCR反应条件:94 ℃预变性4 min,94 ℃变性30 s,58 ℃退火30 s,72 ℃延伸30 s,共33 个循环,72 ℃延伸5 min。
PCR扩增结束后,通过混有荧光染料溴化乙锭(ethidium bromide,EB)的1%琼脂糖凝胶电泳对PCR产物进行检测,并将在凝胶成像系统观测下条带清晰、明亮且单一的产物送至英潍捷基(上海)贸易有限公司北京实验室进行正反双向测序。
1.3.3 特异性引物设计
测序完成获得原始序列数据后,将正向、反向引物数据导入SeqMan软件,去除两端不可信部分后进行人工拼接,秘鲁茎柔鱼和澳洲双柔鱼得到长度分别约为552、553 bp的16S rRNA基因序列(Genbank登录号分别为GU324169和GQ365324)。通过GenBank数据库下载得相应柔鱼的线粒体16S rRNA基因序列,并在数据库中进行Nucleotide Blast比对,根据序列相似度对样品的物种进行鉴别,再次确保所选样品物种与标签一致。通过Clustal X软件对测序结果进行多序列对比和分析,并用Primer Premier 5软件设计秘鲁茎柔鱼和澳洲双柔鱼的品种特异性引物。
1.3.4 多重PCR鉴定
以秘鲁茎柔鱼和澳洲双柔鱼的双位点特异性引物为正向引物,利用Primer Premier 5软件筛选二者的反向引物。筛选反向引物时注意3 条引物的退火温度保持接近,并避免模板错配及引物间二聚体的产生,确定利用16SR作为反向引物。利用3 条引物对2 种柔鱼的模板DNA及混合样品进行多重PCR扩增。多重PCR反应总体系为20 μL:模板DNA 10 ng、2×Premix DNA polymerase 10 μL、正向引物DGF 1 μL(0.5 μmol/L)、正向引物NGF 1 μL(0.5 μmol/L)、反向引物16SR 2 μL(0.5 μmol/L),加灭菌双蒸水至20 μL。PCR反应条件:94 ℃预变性4 min,94 ℃变性30 s,54 ℃退火30 s,72 ℃延伸30 s,共33 个循环,72 ℃延伸5 min。多重PCR扩增结束后,通过混有荧光染料EB的1%琼脂糖凝胶电泳,在凝胶成像系统中对多重PCR产物进行检测。
1.3.5 灵敏度测定及市售样品检测
为测定该多重PCR体系的灵敏度,将DNA模板总量设置为10 ng,以2 种柔鱼的混合样品为模板,其中秘鲁茎柔鱼DNA的质量分数依次为0%、1%、2%、10%及50%。同时分别对市售的2 种秘鲁茎柔鱼和2 种澳洲双柔鱼样品进行检测,以验证本方法在实际中应用的可行性。多重PCR反应条件如1.3.4节所述。扩增结束后,在凝胶成像系统中对多重PCR产物进行检测。
2 结果与分析
2.1 16S rRNA序列比对
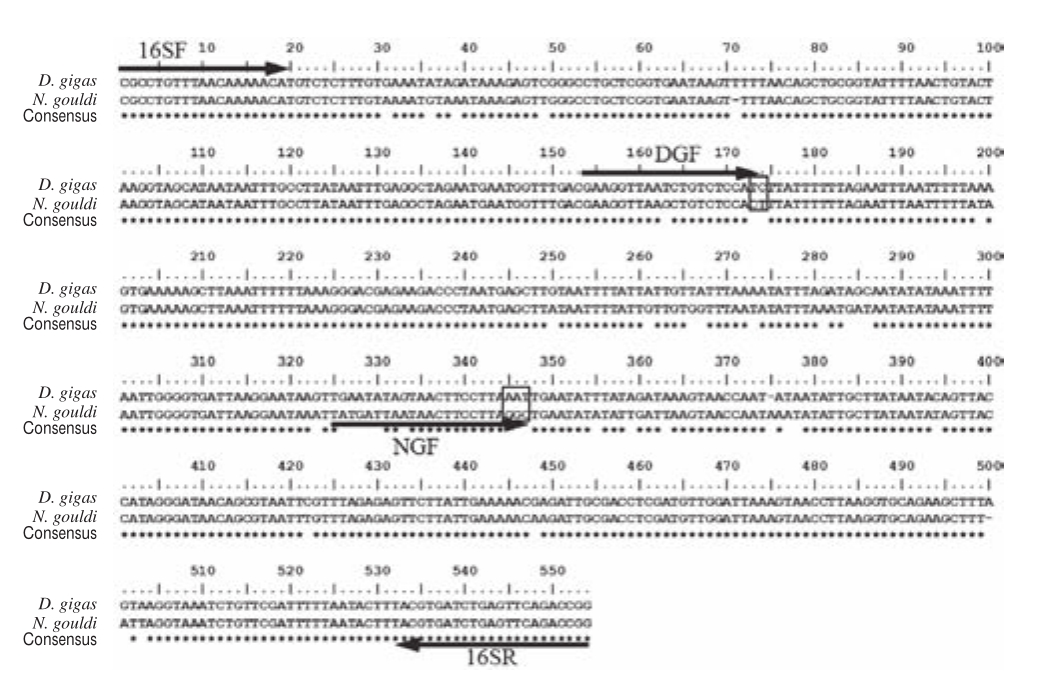
图1 秘鲁茎柔鱼和澳洲双柔鱼的序列比对结果和选用的SNP位点
Fig.1 16S rRNA sequence comparison and selected SNP sites of D.gigas and N.gouldi
通过线粒体16S rRNA通用引物扩增出的秘鲁茎柔鱼和澳洲双柔鱼基因序列长度分别约为552、553 bp,并上传至GenBank数据库。由图1可知:对2 种柔鱼品种的扩增基因序列进行比对后,第173和174号位秘鲁茎柔鱼显示为T、C碱基,而澳洲双柔鱼则为C、T碱基,根据这2 个错配碱基设计秘鲁茎柔鱼的正向特异性引物DGF;第345、346、347号位秘鲁茎柔鱼显示为A、A、T碱基,而澳洲双柔鱼则为G、G、C碱基,根据这3 个错配碱基设计澳洲双柔鱼的正向特异性引物NGF。
2.2 特异性引物设计结果
根据秘鲁茎柔鱼的SNP位点,设计其正向特异性引物DGF(5’-GAAGGTTAATCTGTCTCCATC-3’),利用通用引物1 6 S R作为反向引物;根据澳洲双柔鱼的S N P位点,设计其正向特异性引物N G F(5’-TATGATTAATAACTTCCTTAGGC-3’),同样利用通用引物16SR作为反向引物。2 种柔鱼的品种特异性引物在16S rRNA基因序列中的相对位置如图1所示。
2.3 多重PCR鉴定结果
以秘鲁茎柔鱼和澳洲双柔鱼的基因组DNA及混合样品DNA作为模板,利用秘鲁茎柔鱼的特异性引物DGF、澳洲双柔鱼的特异性引物NGF及16SR 3 条引物进行多重PCR扩增。在凝胶成像系统中对多重PCR产物进行检测。
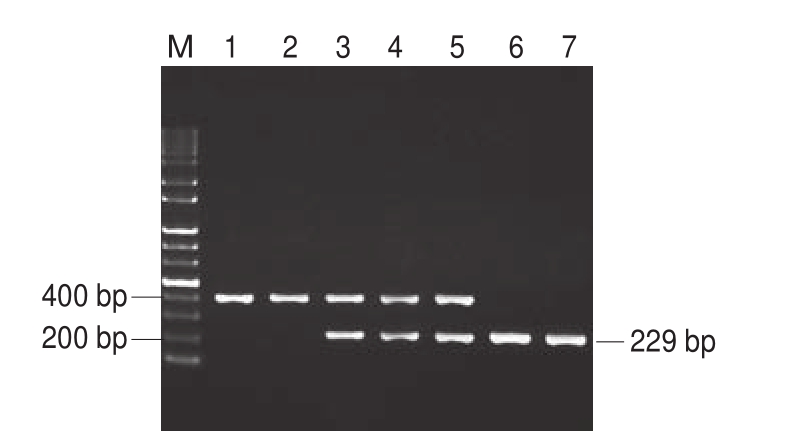
图2 秘鲁茎柔鱼和澳洲双柔鱼及其混合样品的多重PCR鉴定凝胶电泳图
Fig.2 Multiplex PCR identification of D.gigas,N.gouldi and their mixtures
泳道M.100 bp PlusII DNA Ladder;泳道1~2.秘鲁茎柔鱼DNA;泳道3~5.混合样品(秘鲁茎柔鱼DNA质量分数50%)DNA;泳道6~7.澳洲双柔鱼DNA。
由图2可知,经过多重PCR扩增后,秘鲁茎柔鱼和澳洲双柔鱼DNA分别扩增出各自的品种特异性条带,同时,2 种柔鱼的混合样品DNA同时扩增出2 种特异性条带。将2 种条带切胶回收后进行克隆测序,测得2 种特异性条带的序列及长度与图1中2 种柔鱼的序列完全一致,长度分别为400、229 bp。由此可见,2 种柔鱼的特异性引物对相应柔鱼品种产生了绝对特异性。
对秘鲁茎柔鱼和澳洲双柔鱼的多个样品进行重复性实验后,检测结果稳定可靠,重复性好。表明利用秘鲁茎柔鱼和澳洲双柔鱼特异性引物所建立的多重PCR体系不仅可以有效鉴定2 个柔鱼品种,也能对其混合样品进行快速、有效地区分与鉴定。
2.4 灵敏度测定结果
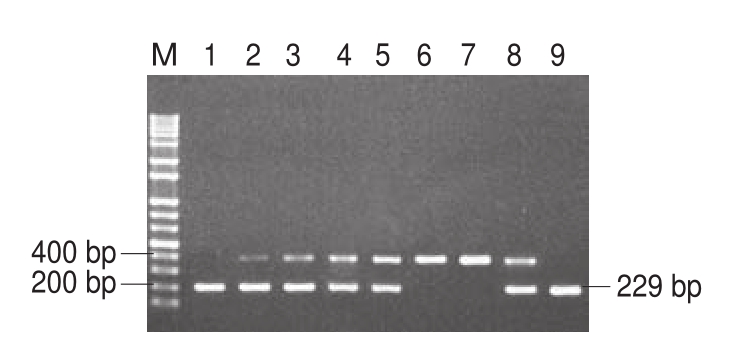
图3 秘鲁茎柔鱼和澳洲双柔鱼混合样品及市售样品的多重PCR检测凝胶电泳图
Fig.3 Multiplex PCR results of mixed and commercial samples of D.gigas and N.gouldi
泳道M.100 bp PlusII DNA Ladder;泳道1~5.混合样品DNA,其中秘鲁茎柔鱼DNA质量分数依次为0%、1%、2%、10%、50%;泳道6~7.市售秘鲁茎柔鱼DNA;泳道8~9.市售澳洲双柔鱼DNA。
由图3可知,秘鲁茎柔鱼和澳洲双柔鱼的混合样品均产生2 条特异性条带,其中秘鲁茎柔鱼的特异性条带长度为400 bp,澳洲双柔鱼的特异性条带长度为229 bp,且随着混合样品中秘鲁茎柔鱼DNA含量的增加,秘鲁茎柔鱼的特异性条带亮度也逐渐增强。表明该多重PCR体系可以检测澳洲双柔鱼中1%秘鲁茎柔鱼的掺入,检测限为0.1 ng。在市售的2 种秘鲁茎柔鱼和2 种澳洲双柔鱼样品检测中发现,1 种澳洲双柔鱼样品存在掺入秘鲁茎柔鱼现象,而其他3 种柔鱼样品均可正确检测其样品来源。因此,本方法灵敏度高,可对市场上的柔鱼原料来源进行准确检测。
3 结 论
通过16S rRNA基因序列确定的品种特异性引物建立多重PCR体系,一管多检,有效减少了试剂消耗和操作时间。在秘鲁茎柔鱼、澳洲双柔鱼混合DNA样品中,目标DNA也能高效扩增并且不受非目标DNA的干扰,可以在同一个PCR反应中同时对秘鲁茎柔鱼、澳洲双柔鱼的单独样品及混合样品进行有效区分和鉴定,大大提高了检测效率,满足了相关柔鱼类品种的检测需要。进行多次重复性检测后,实验结果重复性好、准确率高,方法具有操作简单、检测时间短、无污染、应用广泛等优点。
对于鱼类水产品的物种鉴定,伴随着当今现代分子生物学技术的蓬勃发展,基于DNA分子水平的鉴定方法愈发展现出巨大的潜力。常见的DNA分子标记技术有简单重复序列(simple sequence repeats,SSR)[20-21]、限制性片段长度多态性(restriction fragment length polymorphism,RFLP)[22]、扩增片段长度多态性(amplified fragment length polymorphism,AFLP)[23]、随机扩增多态性DNA(randomly amplified polymorphic DNA,RAPD)[24]和DNA条形码(DNA Barcoding)[25-27]等。其中,SSR分子标记需经过聚丙烯酰胺凝胶电泳,且电泳条带紧密,区分往往不太明显;RFLP分子标记作为最早的分子标记技术,需用限制性内切酶对DNA进行消化,并使用放射性标记探针,操作复杂繁琐,且DNA消耗量大;AFLP分子标记也需对DNA进行限制性内切酶双酶切处理,再进行人工拼接,扩增片段常需要经过聚丙烯酰胺凝胶电泳和银染等手段进行分离检测,操作繁杂;RAPD分子标记为显性遗传,存在共迁移问题,并且实验重复性较差;DNA条形码技术需要对每次PCR扩增出的片段进行测序处理,比较准确,但也耗费较多时间。相较于其他方法,本方法简单高效,针对秘鲁茎柔鱼和澳洲双柔鱼的未知样品,只需DNA提取、PCR扩增和凝胶电泳3 步即可完成鉴定,鉴定成本较低且结果差异明显。本方法从分子水平上对秘鲁茎柔鱼、澳洲双柔鱼的单独样品及其混合样品进行检测,范围更加广泛,结果更加准确,为我国柔鱼品种真伪鉴别提供了新依据,起到规范市场和保护消费者利益的作用,也为我国柔鱼产业的健康发展和相关制度的实施提供了技术支持。
[1]ALLCOCK A L,LINDGREN A,STRUGNELL J M.The contribution of molecular data to our understanding of cephalopod evolution and systematics: a review[J].Journal of Natural History,2015,49(21/24):1373-1421.DOI:10.1080/00222933.2013.825342.
[2]ARKHIPKIN A I,BIZIKOV V A,DOUBLEDAY Z A,et al.Techniques for estimating the age and growth of molluscs:cephalopoda[J].Journal of Shell fish Research,2018,37(4): 783-792.DOI:10.2983/035.037.0409.
[3]陈新军.世界头足类资源开发现状及我国远洋鱿钓渔业发展对策[J].上海海洋大学学报,2019,28(3): 321-330.DOI:10.12024/jsou.20181102445.
[4]宋旻鹏,汪金海,郑小东.中国经济头足类增养殖现状及展望[J].海洋科学,2018,42(3): 149-156.DOI:10.11759/hykx20180128001.
[5]陈新军,刘必林,王尧耕.世界头足类[M].北京: 海洋出版社,2009:190-194.
[6]WALUDA C M,YAMASHIRO C,ELVIDGE C D,et al.Quantifying light- fishing for Dosidicus gigas in the eastern Pacific using satellite remote sensing[J].Remote Sensing of Environment,2004,91(2):129-133.DOI:10.1016/j.rse.2004.02.006.
[7]章寒,郑基,虞聪达,等.东太平洋赤道海域茎柔鱼主要生物学特性比较研究[J].中国水产科学,2019,26(4): 745-755.DOI:10.3724/SP.J.1118.2019.19010.
[8]IBÁÑEZ C M,SEPÚLVEDA R D,ULLOA P,et al.The biology and ecology of the jumbo squid Dosidicus gigas (cephalopoda) in Chilean waters: a review[J].Latin American Journal of Aquatic Research,2015,43(3): 402-414.DOI:10.3856/vol43-issue3-fulltext-2.
[9]Food and Agriculture Organization.The state of world fisheries and aquaculture 2016: contributing to food security and nutrition for all[M].Rome: FAO,2016.
[10]BRALEY M,GOLDSWORTHY S D,PAGE B,et al.Assessing morphological and DNA-based diet analysis techniques in a generalist predator,the arrow squid Nototodarus gouldi[J].Molecular Ecology Resources,2010,10(3): 466-474.DOI:10.1111/j.1755-0998.2009.02767.x.
[11]PALAFOX H,CÓRDOVA-MURUETA J H,NAVARRETE DEL TORO M A,et al.Protein isolates from jumbo squid (Dosidicus gigas) by pH-shift processing[J].Process Biochemistry,2009,44(5):584-587.DOI:10.1016/j.procbio.2009.02.011.
[12]苏杭.几何形态测量学法在大洋性头足类种间及种群鉴别中的应用[D].上海: 上海海洋大学,2016: 29-62.
[13]张静,李渊,宋娜,等.我国沿海棱鳀属鱼类的物种鉴定与系统发育[J].生物多样性,2016,24(8): 888-895.DOI:10.17520/biods.2016079.
[14]顾仲朝,吴媛,王萍亚,等.头足类水产品挥发性盐基氮变化规律的研究[J].食品工业,2018,39(3): 173-177.
[15]曲映红,陈新军,陈舜胜.我国鱿鱼加工利用技术研究进展[J].上海海洋大学学报,2019,28(3): 357-364.DOI:10.12024/jsou.20190102498.
[16]王嘉鹤,陈双雅,陈伟玲,等.DNA检测方法在鱼类物种鉴定中的应用[J].海南大学学报(自然科学版),2012,30(3): 293-298.DOI:10.3969/j.issn.1004-1729.2012.03.022.
[17]陈星,高子厚.DNA分子标记技术的研究与应用[J].分子植物育种,2019,17(6): 1970-1977.
[18]施文瑞,王磊,陈军平,等.鱼类性别特异性DNA分子标记研究进展[J].水产科学,2018,37(6): 862-866.DOI:10.16378/j.cnki.1003-1111.2018.06.023.
[19]SIMON C,FRATI F,BECKENBACH A,et al.Evolution,weighting,and phylogenetic utility of mitochondrial gene sequences and a compilation of conserved polymerase chain reaction primers[J].Annals of the Entomological Society of America,1994,87(6):651-701.DOI:10.1093/aesa/87.6.651.
[20]LIU Lianwei,CHEN Xinjun,XU Qianghua,et al.Development of 39 polymorphic microsatellite markers for the jumbo squid Dosidicus gigas using Illumina paired-end sequencing[J].Conservation Genetics Resources,2014,6(3): 673-676.DOI:10.1007/s12686-014-0179-7.
[21]隋宥珍,刘连为,陈新军.三种柔鱼类SSR标记的通用性检测[J].浙江海洋学院学报(自然科学版),2015,34(6): 532-536.DOI:10.3969/j.issn.1008-830X.2015.06.006.
[22]MCKEOWN N J,ROBIN J P,SHAW P W.Species-specific PCRRFLP for identification of early life history stages of squid and other applications to fisheries research[J].Fisheries Research,2015,167:207-209.DOI:10.1016/j.fishres.2015.02.007.
[23]HAN Zhiqiang,LIN Longshan,SHUI Bonian,et al.Genetic diversity of small yellow croaker Larimichthys polyactis revealed by AFLP markers[J].African Journal of Agricultural Research,2009,4(7):605-610.DOI:10.1021/jf900470c.
[24]ROCCO L,VALENTINO I V,SCAPIGLIATI G,et al.RAPDPCR analysis for molecular characterization and genotoxic studies of a new marine fish cell line derived from Dicentrarchus labrax[J].Cytotechnology,2014,66(3): 383-393.DOI:10.1007/s10616-013-9586-y.
[25]HEBERT P D N,CYWINSKA A,BALL S L,et al.Biological identifications through DNA barcodes[J].Proceedings of the Royal Society B: Biological Sciences,2003,270: 313-321.DOI:10.1098/rspb.2002.2218.
[26]WARD R D,ZEMLAK T S,INNES B H,et al.DNA barcoding Australia’s fish species[J].Philosophical Transactions of the Royal Society B-Biological Sciences,2005,360: 1847-1857.DOI:10.1098/rstb.2005.1716.
[27]KATUGIN O N,CHICHVARKHINA O V,ZOLOTOVA A O,et al.DNA barcoding for squids of the family Gonatidae (Cephalopoda:Teuthida) from the boreal North Pacific[J].Mitochondrial DNA Part A,2017,28(1/2): 41-49.DOI:10.3109/19401736.2015.1110792.