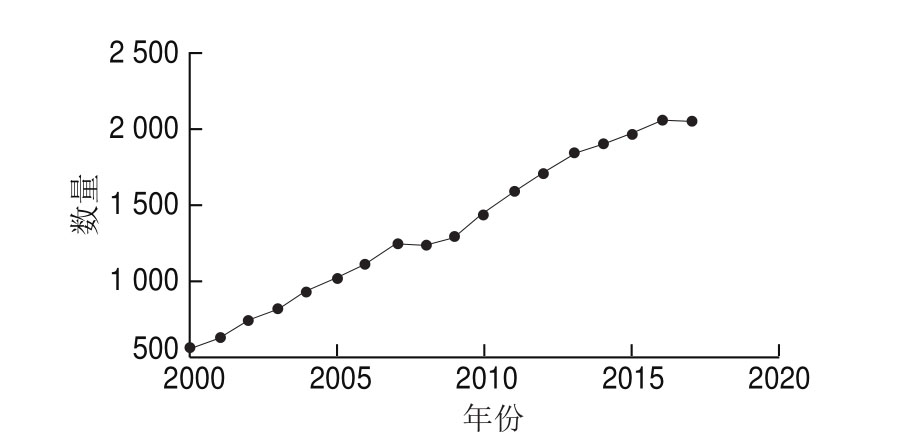
图1 2000—2017年全球每年发表乙酰化相关论文数量统计(PubMed数据库)[3]
Fig. 1 Annual number of worldwide published articles about acetylation from 2000 to 2017 (from PubMed)[3]
近年来,研究发现蛋白质翻译后修饰对生命的调控发挥着越来越重要的作用,且其形式众多,达数百种。其中典型的修饰有磷酸化、乙酰化、泛素化、糖基化及甲基化等,这些修饰普遍存在于包括植物、酵母、小鼠及人类在内的各种生命形式。蛋白质翻译后修饰主要通过在特定的氨基酸侧链上添加新的基团来改变蛋白质的结构和功能,赋予蛋白质新的性质,如改变酶活性、亚细胞定位、蛋白质相互作用及蛋白质的稳定性等[1]。蛋白质赖氨酸乙酰化是一种非常重要且普遍的翻译后修饰,自20世纪60年代文森特·奥弗雷在组蛋白上发现了赖氨酸残基上的可逆乙酰化后,蛋白质乙酰化就一直备受关注[2]。进入21世纪后,随着乙酰化检测技术的不断发展,乙酰化修饰的关注程度逐年上升(图1,搜索关键词为acetylation)。

图1 2000—2017年全球每年发表乙酰化相关论文数量统计(PubMed数据库)[3]
Fig. 1 Annual number of worldwide published articles about acetylation from 2000 to 2017 (from PubMed)[3]
宰后异质肉的形成与肌肉能量代谢和糖酵解速率密切相关[4-5],而参与肌肉能量代谢的关键酶类往往会经过可逆的蛋白质翻译后修饰,进而参与调控整个代谢途径。众多研究表明,蛋白质磷酸化修饰对于宰后肌肉能量代谢起到关键的调控作用[6-7];科学家近年来研究发现,赖氨酸乙酰化修饰在能量代谢调控方面也起着至关重要的作用,并且与磷酸化修饰同样广泛,存在于动植物细胞中的众多部位[8]。
本文描述宰后异质肉的形成与能量代谢之间的关系,并深入总结赖氨酸乙酰化在能量代谢调控中所发挥的关键作用,最后讨论赖氨酸乙酰化对肉品品质的调控作用以及赖氨酸乙酰化在肉品科学领域的发展方向。
宰后异质肉的形成受能量代谢及糖酵解途径的影响。动物屠宰之后,其肌肉细胞氧气供应停止,细胞的呼吸方式转变为无氧呼吸,肌糖原无氧酵解,并最终产生大量乳酸,随着时间的推移,乳酸不断累积,导致肌肉的pH值快速下降。较低的pH值会使得肌浆蛋白变性、肌原纤维收缩,肌肉表面的光反射能力加强,呈现出苍白、柔软、表面失水的异质肉,即PSE(pale, soft and exudative)肉[9]。另一种极端情况是动物在宰前经历长期或过多的消耗,导致宰后肌肉糖原含量过低,肌细胞内的糖酵解程度相比于正常肉较低,肌肉能量代谢不够活跃,无氧糖酵解的产物乳酸相对较少,导致宰后肌肉pH值在24 h后依然维持在6.0以上。高pH值条件下,蛋白质变性程度较低,肌肉失水较少,肌肉表面干燥,对光线的反射能力差,肉色发暗;肌肉中的线粒体摄氧功能没有被抑制,大量的氧被线粒体摄取,氧合肌红蛋白含量低,脱氧肌红蛋白含量高,加剧肉色发暗,不易流动水没有渗出,导致肉质较硬,产生DFD(dark, firm and dry)肉[10]。
蛋白质赖氨酸乙酰化是一种普遍存在于生命体中、动态可逆、并受高度调控的蛋白质翻译后修饰方式[11],其包括组蛋白和非组蛋白乙酰化修饰,在生命过程中起到非常关键的作用,它参与转录调控、信号通路调控、蛋白质稳定性调控及细胞代谢等许多重要的生理调控过程[12]。
乙酰化修饰在赖氨酸乙酰化酶和去乙酰化酶的相互作用下受到精确的调控[13],其生化过程涉及3 类蛋白质:第1类是赖氨酸乙酰转移酶(acetyltrahsferases,KATs),它们的作用是将乙酰基团添加到蛋白质中;第2类是赖氨酸脱乙酰酶(lysine deacetylases,KDACs),其可去除乙酰化蛋白质中的乙酰基;第3类是乙酰赖氨酸结合物,其选择性地与乙酰化蛋白质相互作用。目前,已有22 种不同的KATs在人类和鼠细胞中被发现[14],它们可以分为GCN5、CBP/p300和MYST11 3 个主要的家族[15-17]。在人类和小鼠基因组中鉴定出的18 种KDACs属于2 个不同的家族,它们的催化机制也有所不同,一种是Zn2+依赖性组蛋白去乙酰化酶(histone deacetylase1-11,HDAC1-11)催化[18],另一种是烟酰胺腺嘌呤二核苷酸(nicotinamide adenine dinucleotide,NAD+)依赖性Sirtuin脱乙酰酶(sirtuin deacetylases1-70,SIRT1-70)催化[19-20]。
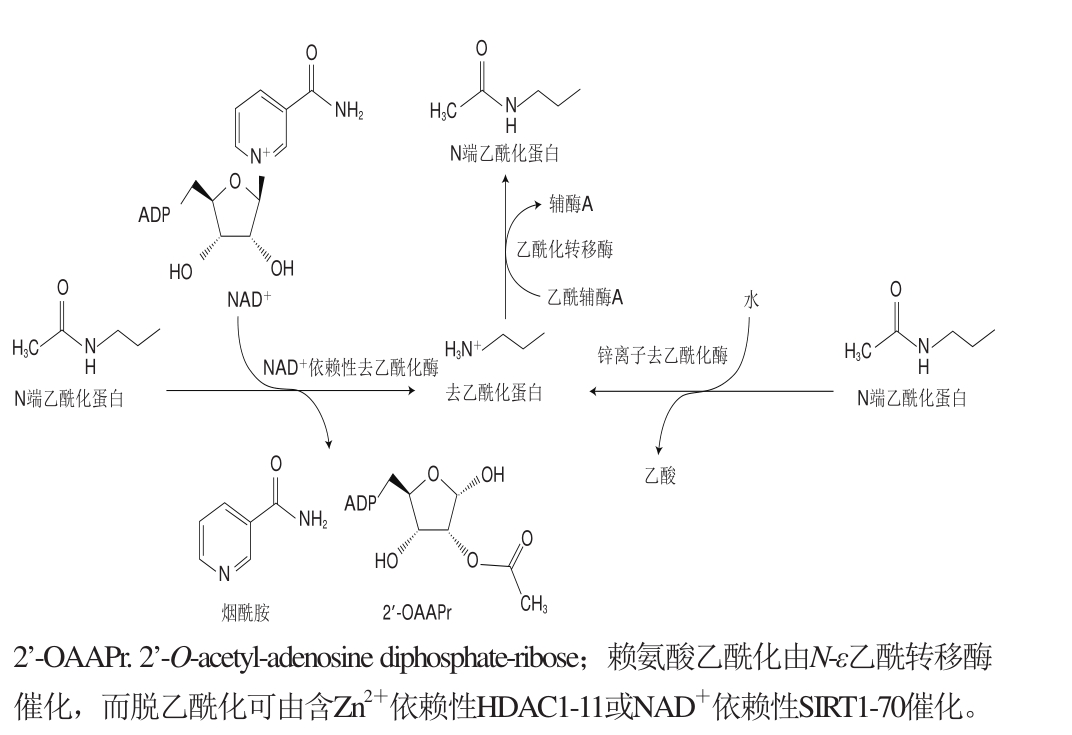
图2 乙酰化和脱乙酰化的机理[22]
Fig. 2 Mechanism of acetylation and deacetylation[22]
KATs以乙酰辅酶A作为必需的辅助因子,将乙酰基转移到特定的赖氨酸残基上。而KDACs可以将Zn2+作为辅助因子,或以NAD+作为共底物从赖氨酸中除去乙酰基(图2)。KATs与去乙酰酶共同调节细胞内蛋白质的乙酰化修饰,在众多的代谢调控及代谢相关疾病中发挥着重要作用[21]。
赖氨酸乙酰化在细胞能量代谢中发挥着关键作用。随着修饰蛋白质组学检测技术的不断更新,人们发现细胞内存在着大量受乙酰化修饰调控的代谢酶或代谢相关蛋白。研究发现,人肌肉细胞中乙酰化修饰的必要前体物质乙酰辅酶A的水平在运动10 min时升高,并在整个运动过程中持续保持升高;与休息时相比,所有运动时间的乙酰辅酶A水平都更高,这从侧面说明随着运动量的加大或能量消耗的增加,机体内的乙酰化修饰水平可能提高[23]。
Kim等[24]研究小鼠的肝脏细胞,发现线粒体中有超过20%的蛋白质发生乙酰化修饰,其中包括很多参与代谢的酶类。Lin Ruiting[25]、Sutendra[26]等的研究表明,乙酰辅酶A的浓度与细胞所处的环境相关,环境中的葡萄糖浓度与相关代谢酶及其水平有很大联系。葡萄糖转变为乙酰辅酶A过程中有2 类重要的代谢酶,即ATP-柠檬酸裂解酶(ATP-citrate lyase,ACLY)和丙酮酸脱氢酶复合体(pyruvatedehy drogenase complex,PDC),这2 类蛋白质的活性与组蛋白的乙酰化水平显著相关。

图3 能量代谢通路调控酶的乙酰化修饰[11]
Fig. 3 Acetylation of enzymes regulating the energy metabolism pathway[11]
HK. 己糖激酶(hexokinase);PGI. 磷酸己糖异构酶(phosphate isomerase);FBPase. 果糖二磷酸酶(fructose bisphosphatase);PFK-1. 果糖-1,6-二磷酸酶(fructose-1,6-bisphosphatase);ALD. 果糖-1,6-二磷酸醛缩酶(fructose-1,6-bisphosphate aldolase);TPI. 丙糖磷酸异构酶(triosephosphate isomerase);GAPDH. 甘油醛-3-磷酸脱氢酶(glyceraldehyde 3-phosphate dehydrogenase);PGK. 磷酸甘油酸激酶(phosphoglycerate kinase);PGAM. 磷酸甘油酸变位酶(phospho-glycerate mutase);Enolase. 烯醇化酶;PK. 丙酮酸激酶(pyruvate kinase);PDH. 丙酮酸脱氢酶(pyruvate dehydrogenase);PC. 丙酮酸羧化酶(pyruvate carboxylase);PEPCK. 磷酸烯醇式丙酮酸羧激酶(phosphoenolpyruvate carboxykinase);UDP-GP. 焦磷酸化酶(pyrophosphorylase);GS. 糖原合酶(glycogen synthase);GP. 糖原磷酸化酶(glycogen phosphorylase);PGM3. 磷酸葡萄糖变位酶(phosphoglucomutase);MDH. 苹果酸脱氢酶(dehydrogenase);CS. 柠檬酸合酶(citrate synthetase);ACO. 顺乌头酸酶(aconitase);IDH3. 异柠檬酸脱氢酶3(isocitric dehydrogenase 3);α-KGDH. α-酮戊二酸脱氢酶(α-ketoglutarate dehydrogenase);SCS. 琥珀酰辅酶A合成酶(succinyl-CoA synthetase);SDH. 琥珀酸脱氢酶(succinate dehydrogenase);FH. 延胡索酸水化酶(fumarate hydratase)。其中,PGI和PGM3是没有发生乙酰化的酶,其余是已经发现的乙酰化代谢酶。代谢酶周围的箭头代表其调控作用,箭头向上表示乙酰化修饰使得酶的活性增强,对通路起到促进作用;箭头向下表示乙酰化修饰使得酶的活性减弱,对通路起到抑制作用。a. 糖酵解与糖异生;b. 糖原代谢;c. 三羧酸循环。
由图3可知,首先,乙酰化修饰在调控糖酵解过程中起到关键作用,众多代谢路径上的酶均发生了乙酰化(图3a)。丙酮酸激酶M2(pyruvate kinase M2,PKM2)和乳酸脱氢酶A(lactate dehydrogenase A,LDHA)在糖类分解代谢中均可能发生乙酰化修饰,而它们在糖酵解途径中起着关键作用。通过乙酰化修饰,丙酮酸激酶会增强与分子伴侣热激同源蛋白70的相互作用,进而激活其自噬分解[27]。Lü Lei等[28]进一步研究PKM2的乙酰化修饰发现,PKM2上具有多个乙酰化位点,这些位点的修饰使其催化特性发生改变,活性增强。Li Tingting等[29]发现,糖酵解中的GAPDH经过乙酰化修饰后活性增强,进而可以促进糖酵解途径的完成。另外,Xu Yanping等[30]研究发现,PGAM的2 个同工酶同样会发生乙酰化修饰。其中,PGAM1经过修饰后,其活性会增强,促进糖分解代谢;与此相反,PGAM2被乙酰化后活性受到抑制。同样,Wang Qijun等[31]通过研究肠道沙门氏菌发现,磷酸甘油醛脱氢酶(glyceraldehyde phosphate dehydrogenase,GapA)双向引导糖酵解和糖异生,其与异柠檬酸裂解酶(isocitrate lyase,ICL)和异柠檬酸脱氢酶激酶均通过可逆的乙酰化修饰来调节自身活性,从而调控糖酵解及其他的能量代谢通路。
糖异生通路中的众多关键酶类受乙酰化修饰的调控[32]。Zhao Shimin等[33]在肝细胞中发现,PEPCK1上有3 个赖氨酸残基发生乙酰化修饰。随着葡萄糖浓度的升高,PEPCK1的乙酰化程度提高;在低浓度葡萄糖条件下,PEPCK1的乙酰化程度降低,其稳定性提高,从而使得代谢趋于糖异生。同样,Jiang Wenqing等[32]的研究也进一步证实,PEPCK1的乙酰化修饰水平提高,其稳定性就会降低,反之稳定性升高,从而调控糖异生途径。
乙酰化亦可以调控磷酸戊糖途径,在磷酸戊糖途径中,葡萄糖-6-磷酸脱氢酶(glucose-6-phosphatedehydrogenase,G6PD)和6-磷酸葡萄糖酸脱氢酶(6-phosphogluconate dehydrogenase,6-PGD)2 个代谢酶参与烟酰胺腺嘌呤二核苷酸磷酸(nicotinamide adenine dinucleotide phosphate,NADPH)的生成[34]。Wang Yiping等[35]研究发现,G6PD通过乙酰化修饰来改变蛋白质的聚合状态,使得其催化活性受到抑制,从而调控磷酸戊糖途径。
乙酰化还可以调控糖原分解过程中的关键酶(图3b)。糖原磷酸化酶是保持机体细胞中糖原稳态的关键酶,Zhang Tengfei等[36]研究肝细胞发现,细胞经过脱乙酰酶抑制剂处理后,GP的乙酰化修饰水平提高,但其酶活性也随之降低,这表明乙酰化对GP的活性起到负调控作用,对糖原的稳定性起到关键作用。
乙酰化修饰同样能调控三羧酸循环(图3c)。Zhao Shimin等[33]研究发现,在动物细胞中,参与三羧酸循环的7 个酶均会经过乙酰化修饰。苹果酸脱氢酶(malate dehydrogenase,MDH)就是其中的关键酶之一,Hebert等[37]研究发现,乙酰化修饰会提高MDH的酶活,进而影响三羧酸循环。
乙酰化修饰还参与脂肪酸代谢、尿素循环等许多重要的生命代谢通路,对自然界的各种生命体系都具有重要的意义。
肉品品质的形成受很多因素影响,包括宰前管理、温度、季节、动物品种以及宰后的能量调控等。大量研究表明,乙酰化修饰通过对能量代谢通路当中相关酶的调控影响机体细胞中的能量代谢进程,而能量代谢的调控对宰后肉品品质的形成起着关键作用。因此,乙酰化修饰可能与磷酸化修饰一样对动物宰后肉品品质起着重要作用。Li Zhongwen等[38]研究表明,不同的宰前应激会造成小鼠骨骼肌中蛋白质总乙酰化水平的差异,表现在宰后45 min总乙酰化程度相对较高,同时伴随着pH值的降低、乳酸含量的增加以及丙酮酸激酶和己糖激酶活性的提高,这说明较高的乙酰化水平会加快能量代谢进程,并可能导致宰后早期肌肉的持水力下降。进一步研究表明,对宰前小鼠体内注射乙酰转移酶抑制剂,会造成宰后肌肉乙酰化水平降低。与此同时,宰后糖酵解速率也随之降低;而宰前注射去乙酰化酶抑制剂会造成乙酰化水平的增加,由此引发丙酮酸激酶的活性提高,这也证明了糖酵解速率的提高[39]。最近的研究显示,生猪宰后肌肉乙酰化蛋白质的差异表达与猪肉的肉色、嫩度及持水力关系密切,能量代谢、肌肉收缩、钙离子信号通路中的蛋白质发生乙酰化,并参与调控猪肉品质的形成[40]。这些研究充分证明,乙酰化修饰可能通过调控相关蛋白来影响宰后猪肉品质。
蛋白质乙酰化和去乙酰化受乙酰化酶和去乙酰化酶直接调控,进而完成可逆的修饰过程,但2 种催化酶会受到众多因素的影响。首先,研究表明,NAD+是影响乙酰化发生的重要条件,其对Sirtuin去乙酰化酶的活性起到至关重要的作用,调节蛋白组的乙酰化状态,最终影响细胞的代谢平衡[41]。其次,烟酰胺(nicotinamide,NAM)对乙酰化的调控也起到一定作用,其是影响Sirtuin蛋白的重要代谢物,可以非竞争性地抑制Sir2P蛋白(一种特定的去乙酰化酶),并能够负向调控特定哺乳动物的去乙酰化酶[42]。乙酰辅酶A作为所有乙酰转移酶的底物,是乙酰化修饰反应的重要基础。乙酰辅酶A受到细胞代谢状态的影响,并能够直接影响KATs和乙酰转移酶(N-acetyltransferase,NATs)的功能[43-44]。此外,乙酰辅酶A也可以通过非酶乙酰化反应影响蛋白质乙酰化[45]。辅酶A也是影响乙酰化修饰的重要因素之一,其涉及KAT产物的抑制。KAT2A、KAT2B等乙酰化转移酶均受到辅酶A水平变化的调控[46]。同样,丁酸和β-羟基丁酸也是不可忽视的重要因素。丁酸是由肠道微生物从膳食纤维中产生的一种短链脂肪酸,其可以作为一类和二类KDACs的抑制剂,进而导致组蛋白乙酰化修饰的增加[47];而β-羟基丁酸的增加可以降低一类KADC的活力,最终引起整体乙酰化水平的提高[48]。另外,锌离子在某些物种和疾病中可以对KDAC起到调控作用[49]。
众多的宰前或宰后因素可能会造成宰后肌肉中NAD+、NAM、乙酰辅酶A等代谢物水平的变化,影响乙酰转移酶和去乙酰转移酶的含量,调控乙酰化修饰并改变能量代谢进程,最终改变肉品品质。
肉的颜色、持水力和嫩度是肉食用品质的关键指标,对消费者的购买欲望及肉制品加工影响深远。乙酰化作为肌肉细胞中非常普遍的修饰,调控着糖酵解进程,进而影响宰后肌肉的pH值,并在极端情况下可能导致异质肉的发生,最终改变肌肉的持水性能。与此同时,肌肉细胞中的肌浆蛋白可能发生乙酰化修饰,加上肌原纤维收缩性能的改变,进而可能会影响宰后肌肉的嫩化进程,最终导致肉的嫩度发生改变。在以往的研究中,蛋白磷酸化对肉色的调控起着重要作用,主要是基于糖酵解途径中的蛋白质以及肌红蛋白的修饰,乙酰化修饰可能同样调控这些蛋白,影响肌肉表面的颜色。目前,肉品科学领域对乙酰化调控肉色、嫩度和持水力等食用品质还未深入研究,需要不断探索。
在不同环境中,动物机体内会呈现不同的反应。高温或低温等不同的环境会对宰前动物有很大影响。Nannoni等[50]在调查生猪宰前的死亡情况时发现,夏季高温往往会造成更大的死亡比例,这说明高温在宰前使生猪产生很大的应激反应。同样,宰前的驱赶、电击等忽略动物福利的行为也会使动物产生很大应激。这些应激反应可能会造成动物的机体紊乱,进而可能造成肌肉细胞内的蛋白质,尤其是一些代谢通路上的催化酶类发生修饰或脱去修饰基团;不仅如此,动物在宰后,肌细胞内的蛋白修饰可能会随着应激环境的不同,而有不同的修饰程度和状态。Li Xiao等[51]研究春夏秋冬4 个季节生猪宰后肌原纤维蛋白的磷酸化情况,发现季节对宰后猪肉磷酸化修饰水平有显著影响。
同样,对于不同的季节、温度、宰前状况、饲养环境和动物品种,动物宰前或宰后肌肉细胞也可能会发生不同水平的乙酰化修饰。
磷酸化作为机体内最普遍、最为重要的蛋白质翻译后修饰,其参与调控的代谢通路及相关的酶类非常多。同样,乙酰化修饰也是常见的翻译后修饰,在调控机体代谢通路方面也起到非常大的作用。所以在机体细胞当中,一种蛋白质或酶类很有可能既发生磷酸化修饰,也发生乙酰化修饰。Huang等[52-54]在研究动物宰后肌肉时发现,糖酵解途径中的GAPDH发生磷酸化,其活性也增强。Li Tingting等[29]发现,丙酮酸激酶同样会发生乙酰化。Zhang Tengfei等[36]通过研究糖原磷酸化酶的磷酸化和乙酰化修饰发现,糖原磷酸化酶Lys470位点的乙酰化修饰增强了其与磷酸酶-1底物靶向亚基的交互作用,从而促进糖原磷酸化酶去磷酸化并使其失活。这表明乙酰化修饰不仅对某些酶类呈现负调控作用,而且还会抑制其磷酸化修饰的发生。因此,对于肌肉的颜色、嫩度和持水力而言,2 种修饰可能同时发挥调控作用。在机体细胞中,还可能会有很多类似的蛋白质或催化酶同时发生2 种修饰,尤其是能量代谢通路中的调控酶。就肉品科学而言,参与调控肉品品质的蛋白质种类很多,未来也应重点关注磷酸化和乙酰化的共同作用。可以利用修饰蛋白质组学技术将大量蛋白质鉴定出来,从宏观角度分析2 种蛋白质修饰的交互作用,并在此基础上专门分析某几种或某几类关键催化酶在能量代谢通路上所起到的重要作用。随着蛋白质组学技术的不断发展及质谱技术的不断更新,磷酸化修饰与乙酰化修饰的交互作用对肉品品质的控制机制将逐步阐明。
不久的将来,随着乙酰化在肉品领域的深入研究,其调控肉品品质的机制也会被解释。科学家们可以在充分理解其调控原理的基础之上,在饲养环节加入调控乙酰化的饲料或在宰后环节加入调控乙酰化的天然、无害的制剂,从而改善宰后肉品的质量。这不仅能够对肉品的加工做出贡献,也将为动物生产和饲养提供一定的理论指导。
乙酰化修饰作为众多生命过程中重要的修饰方式之一,对动物肌肉的能量代谢起着至关重要的作用,其可能通过影响糖酵解、糖原代谢、三羧酸循环等能量代谢通路中的相关调控蛋白,改变某些关键催化酶的催化活性,进而影响能量的代谢速率,最终形成不同品质的肉类。近年来,随着质谱技术及生物信息学的不断发展,乙酰化修饰在生命体中的功能将逐步被揭示。乙酰化蛋白质组学技术也将广泛运用于肉品科学领域,为研究肉品品质的形成机制做出重要贡献。然而,乙酰化修饰在细胞中是一个动态可逆的过程,且乙酰化蛋白的数量往往很少,因此,乙酰化在肉品科学中需要更精确的实验方法和更科学的分析手段进行研究,研究人员也需要不断探索更合适的实验条件。
目前,乙酰化在肉品科学领域的研究和应用还很少,其调控肉品质量的具体机制尚不明确。因此,未来肉品科学研究人员应当注重乙酰化修饰在肌肉体系中的调控作用,应特别注重利用乙酰化蛋白质组学技术,更全面、客观地分析乙酰化对肉的颜色、保水性及嫩度的影响。
[1] VERDIN E, OTT M. 50 years of protein acetylation: from gene regulation to epigenetics, metabolism and beyond[J]. Nature Reviews Molecular Cell Biology, 2014, 16(4): 258-264. DOI:10.1038/nrm3931.
[2] PHILLIPS D M P. The presence of acetyl groups in histones[J].Biochemical Journal, 1963, 87(2): 258-263. DOI:10.1097/00006254-195712030-00020.
[3] National Center for Biotechnology Information[EB/OL]. [2019-06-16].https://www.ncbi.nlm.nih.gov/pubmed/.
[4] SHEN Q W, MEANS W J, THOMPSON S A, et al. Pre-slaughter transport, AMP-activated protein kinase, glycolysis, and quality of pork loin[J]. Meat Science, 2006, 74(2): 388-395. DOI:10.1016/j.meatsci.2006.04.007.
[5] XING Tong, XU Xinglian, JIANG Nannan, et al. Effect of transportation and pre-slaughter water shower spray with resting on AMP-activated protein kinase, glycolysis and meat quality of broilers during summer[J]. Animal Science Journal, 2016, 87(2): 299-307.DOI:10.1111/asj.12426.
[6] LI Chunbao, ZHOU Guanghong, XU Xinglian, et al.Phosphoproteome analysis of sarcoplasmic and myof i brillar proteins in bovine Longissimus muscle in response to postmortem electrical stimulation[J]. Food Chemistry, 2015, 175: 197-202. DOI:10.1016/j.foodchem.2014.11.139.
[7] LI Meng, LI Xin, XIN Jianzeng, et al. Effects of protein phosphorylation on color stability of ground meat[J]. Food Chemistry,2017, 219: 304-310. DOI:10.1016/j.foodchem.2016.09.151.
[8] LUNDBY A, LAGE K, WEINERT B T, et al. Proteomic analysis of lysine acetylation sites in rat tissues reveals organ specificity and subcellular patterns[J]. Cell Reports, 2012, 2(2): 419-431.DOI:10.1016/j.celrep.2012.07.006.
[9] 李霄. 季节对猪肉蛋白磷酸化和肉品品质的影响及品质快速预测方法研究[D]. 南京: 南京农业大学, 2015: 1-7.
[10] CHAUHAN S S, ENGLAND E M. Postmortem glycolysis and glycogenolysis: insights from species comparisons[J]. Meat Science,2018, 144: 118-126. DOI:10.1016/j.meatsci.2018.06.021.
[11] GUAN Kunliang, XIONG Yue. Regulation of intermediary metabolism by protein acetylation[J]. Trends in Biochemical Sciences,2011, 36(2): 108-116. DOI:10.1016/j.tibs.2010.09.003.
[12] BOYCHEVA I, VASSILEVA V, IANTCHEVA A. Histone acetyltransferases in plant development and plasticity[J]. Current Genomics,2014, 15(1): 28-37. DOI:10.2174/138920291501140306112742.
[13] LIN Ruiting, ZHOU Xin, HUANG Wei, et al. Acetylation control of cancer cell metabolism[J]. Current Pharmaceutical Design, 2014,20(15): 2627-2633. DOI:10.2174/13816128113199990487.
[14] CHOUDHARY C, WEINERT B T, NISHIDA Y, et al. The growing landscape of lysine acetylation links metabolism and cell signalling[J].Nature Reviews Molecular Cell Biology, 2014, 15(8): 536-550.DOI:10.1038/nrm3841.
[15] ROTH S Y, DENU J M, ALLIS C D. Histone acetyltransferases[M]//Encyclopedia of Cancer. Berlin: Springer-Verlag Berlin Heidelberg,2005. DOI:10.1146/annurev.biochem.70.1.81.
[16] LEE K K, WORKMAN J L. Histone acetyltransferase complexes: one size doesn’t fi t all[J]. Nature Reviews Molecular Cell Biology, 2007,8(4): 284-295. DOI:10.1038/nrm2145.
[17] BERNDSEN C E, DENU J M. Catalysis and substrate selection by histone/protein lysine acetyltransferases[J]. Current Opinion in Structural Biology, 2008, 18(6): 682-689. DOI:10.1016/j.sbi.2008.11.004.
[18] HABERLAND M, MONTGOMERY R L, OLSON E N. The many roles of histone deacetylases in development and physiology:implications for disease and therapy[J]. Nature Reviews Genetics,2009, 10(1): 32-42. DOI:10.1038/nrg2485.
[19] HOUTKOOPER R H, PIRINEN E, AUWERX J. Sirtuins as regulators of metabolism and healthspan[J]. Nature Reviews Molecular Cell Biology, 2012, 13(4): 225-238. DOI:10.1038/nrm3293.
[20] HALL J A, DOMINY J E, LEE Y, et al. The sirtuin family’s role in aging and age-associated pathologies[J]. Journal of Clinical Investigation, 2013, 123(3): 973-979. DOI:10.1172/JCI64094.
[21] THAO S, ESCALANTE-SEMERENA J C. Control of protein function by reversible Nɛ-lysine acetylation in bacteria[J]. Current Opinion in Microbiology, 2011, 14(2): 200-204. DOI:10.1016/j.mib.2010.12.013.
[22] MENZIES K J, ZHANG H, KATSYUBA E, et al. Protein acetylation in metabolism-metabolites and cofactors[J]. Nature Reviews Endocrinology, 2016, 12(1): 43-60. DOI:10.1038/nrendo.2015.181.
[23] WATT M J, HEIGENHAUSER G J, DYCK D J, et al. Intramuscular triacylglycerol, glycogen and acetyl group metabolism during 4 h of moderate exercise in man[J]. Journal of Physiology, 2010, 541(3):969-978. DOI:10.1113/jphysiol.2002.018820.
[24] KIM S C, SPRUNG R, CHEN Y, et al. Substrate and functional diversity of lysine acetylation revealed by a proteomics survey[J]. Molecular Cell, 2006, 23(4): 607-618. DOI:10.1016/j.molcel.2006.06.026.
[25] LIN Ruiting, TAO Ren, GAO Xue, et al. Acetylation stabilizes ATP-citrate lyase to promote lipid biosynthesis and tumor growth[J]. Molecular Cell, 2013, 51(4): 506-518. DOI:10.1016/j.molcel.2013.07.002.
[26] SUTENDRA G, KINNAIRD A, DROMPARIS P, et al. A nuclear pyruvate dehydrogenase complex is important for the generation of acetyl-CoA and histone acetylation[J]. Cell, 2014, 158(1): 84-97.DOI:10.1016/j.cell.2014.04.046.
[27] LÜ Lei, LI Dong, ZHAO Di, et al. Acetylation targets the M2 isoform of pyruvate kinase for degradation through chaperone-mediated autophagy and promotes tumor growth[J]. Molecular Cell, 2011,42(6): 719-730. DOI:10.1016/j.molcel.2011.04.025.
[28] LÜ Lei, XU Yanping, ZHAO Di, et al. Mitogenic and oncogenic stimulation of K433 acetylation promotes PKM2 protein kinase activity and nuclear localization[J]. Molecular Cell, 2013, 52(3):340-352. DOI:10.1016/j.molcel.2013.09.004.
[29] LI Tingting, LIU Mengxi, FENG Xu, et al. Glyceraldehyde-3-phosphate dehydrogenase is activated by lysine 254 acetylation in response to glucose signal[J]. Journal of Biological Chemistry, 2014,289(6): 3775-3785. DOI:10.1074/jbc.M113.531640.
[30] XU Yanping, LI Fulong, LÜ Lei, et al. Oxidative stress activates SIRT2 to deacetylate and stimulate phosphoglycerate mutase[J].Cancer Research, 2014, 74(13): 3630-3642. DOI:10.1158/0008-5472.CAN-13-3615.
[31] WANG Qijun, ZHANG Yakun, YANG Chen, et al. Acetylation of metabolic enzymes coordinates carbon source utilization and metabolic flux[J]. Science, 2010, 327: 1004-1007. DOI:10.1126/science.1179687.
[32] JIANG Wenqing, WANG Shiwen, XIAO Mengtao, et al. Acetylation regulates gluconeogenesis by promoting PEPCK1 degradation via recruiting the UBR5 ubiquitin ligase[J]. Molecular Cell, 2011, 43(1):33-44. DOI:10.1016/j.molcel.2011.04.028.
[33] ZHAO Shimin, XU Wei, JIANG Wenqing, et al. Regulation of cellular metabolism by protein lysine acetylation[J]. Science, 2010, 327(5968):1000-1004. DOI:10.1126/science.1179689.
[34] XU Shuangnian, WANG Tianshi, LI Xi, et al. SIRT2 activates G6PD to enhance NADPH production and promote leukaemia cell proliferation[J]. Scientific Reports, 2016, 6: 32734. DOI:10.1038/srep32734.
[35] WANG Yiping, ZHOU Lisha, ZHAO Yuzheng, et al. Regulation of G6PD acetylation by SIRT2 and KAT9 modulates NADPH homeostasis and cell survival during oxidative stress[J]. EMBO Journal, 2014, 33(12): 1304-1320. DOI:10.1002/embj.201387224.
[36] ZHANG Tengfei, WANG Shiwen, YAN Lin, et al. Acetylation negatively regulates glycogen phosphorylase by recruiting protein phosphatase 1[J]. Cell Metabolism, 2012, 15(1): 75-87. DOI:10.1016/j.cmet.2011.12.005.
[37] HEBERT A S, DITTENHAFERREED K E, YU W, et al. Calorie restriction and SIRT3 trigger global reprogramming of the mitochondrial protein acetylome[J]. Molecular Cell, 2013, 49(1):186-199. DOI:10.1016/j.molcel.2012.10.024.
[38] LI Zhongwen, LI Xin, WANG Zhenyu, et al. Antemortem stress regulates protein acetylation and glycolysis in postmortem muscle[J]. Food Chemistry, 2016, 202: 94-98. DOI:10.1016/j.foodchem.2016.01.085.
[39] LI Qiong, LI Zhongwen, LOU Aihua, et al. Histone acetyltransferase inhibitors antagonize AMP-activated protein kinase in postmortem glycolysis[J]. Asian-Australasian Journal of Animal Sciences, 2017,30(6): 857-864. DOI:10.5713/ajas.16.0556.
[40] ZHOU Bing, SHEN Zhenglei, LIU Yisong, et al. Proteomic analysis reveals that lysine acetylation mediates the effect of antemortem stress on postmortem meat quality development[J]. Food Chemistry, 2019,293: 396-407. DOI:10.1016/j.foodchem.2019.04.122.
[41] IMAI S, ARMSTRONG C M, KAEBERLEIN M, et al. Transcriptional silencing and longevity protein Sir2 is an NAD-dependent histone deacetylase[J]. Nature, 2000, 403: 795-800. DOI:10.1038/35001622.
[42] BITTERMAL K J, ANDERSON R M, COHEN H Y, et al. Inhibition of silencing and accelerated aging by nicotinamide, a putative negative regulator of yeast Sir2 and human SIRT1[J]. Journal of Biological Chemistry, 2002, 277: 45099-45107. DOI:10.1074/jbc.m205670200.
[43] YI C H, PAN H L, SEEBACHER J, et al. Metabolic regulation of protein N-alpha-acetylation by Bcl-xL promotes cell survival[J]. Cell,2011, 146(4): 607-620. DOI:10.1016/j.cell.2011.06.050.
[44] TAKAHASHI H, MCCAFFERY J M, IRIZARRY R A, et al.Nucleocytosolic acetyl-coenzyme a synthetase is required for histone acetylation and global transcription[J]. Molecular Cell, 2006, 23(2):207-217. DOI:10.1016/j.molcel.2006.05.040.
[45] WEINERT B T, IESMANTAVICIUS V, MOUSTAFA T, et al.Acetylation dynamics and stoichiometry in Saccharomyces cerevisiae[J]. Molecular Systems Biology, 2014, 10(1): 716.DOI:10.1002/msb.134766.
[46] BEMDSEN C E, ALBAUGH B N, TAN S, et al. Catalytic mechanism of a MYST family histone acetyltransferase[J]. Biochemistry, 2007,46(3): 623-629. DOI:10.1021/bi602513x.
[47] DAVIE J R. Inhibition of histone deacetylase activity by butyrate[J].Journal of Nutrition, 2003, 133: 2485S-2493S. DOI:10.1038/sj.ijo.0802291.
[48] SHIMAZU T, HIRSCHEY M D, NEWMAN J, et al. Suppression of oxidative stress by β-hydroxybutyrate, an endogenous histone deacetylase inhibitor[J]. Science, 2013, 339: 211-214. DOI:10.1126/science.1227166.
[49] ZHAO Yantao, ZHONG Wei, SUN Xiuhua, et al. Zinc deprivation mediates alcohol-induced hepatocyte IL-8 analog expression in rodents via an epigenetic mechanism[J]. American Journal of Pathology, 2011,179(2): 693-702. DOI:10.1016/j.ajpath.2011.04.006.
[50] NANNONI E, LIUZZO G, SERRAINO A, et al. Evaluation of preslaughter losses of Italian heavy pigs[J]. Animal Production Science,2016, 57(10): 2072-2081. DOI:10.1071/AN15893.
[51] LI Xiao, TIAN Fang, ZONG Menghuan, et al. Phosphorproteome changes of myof i brillar proteins at early post-mortem time in relation to pork quality as affected by season[J]. Journal of Agricultural and Food Chemistry, 2015, 63(47): 10287-10294. DOI:10.1021/acs.jafc.5b03997.
[52] HUANG H, LARSEN M R, LAMETSCHA R. Changes in phosphorylation of myofibrillar proteins during postmortem development of porcine muscle[J]. Food Chemistry, 2012, 134(4):1999-2006. DOI:10.1016/j.foodchem.2012.03.132.
[53] HUANG H, LARSEN M R, PALMISANO G, et al. Quantitative phosphoproteomic analysis of porcine muscle within 24 h postmortem[J]. Journal of Proteomics, 2014, 106: 125-139.DOI:10.1016/j.jprot.2014.04.020.
[54] HUANG H, SCHEFFLER T L, GERRARD D E, et al. Quantitative proteomics and phosphoproteomics analysis revealed different regulatory mechanisms of halothane and rendement napole genes in porcine muscle metabolism[J]. Journal of Proteome Research, 2018,17(8): 2834-2849. DOI:10.1021/acs.jproteome.8b00294.
Association of Meat Quality with Acetylation and Energy Metabolism
ZOU Bo, LI Chunbao. Association of meat quality with acetylation and energy metabolism[J]. Meat Research, 2019, 33(8):58-64. DOI:10.7506/rlyj1001-8123-20190617-134. http://www.rlyj.net.cn