牛源性大肠杆菌O157:H7的分离鉴定及其耐酸性比较研究
付文静,姬赛赛,禹金龙,张 苏,徐媛媛,江 芸*
(南京师范大学食品与制药工程学院,江苏 南京 210 023)
摘 要:为比较大肠杆菌(Escherichia coli)O157:H7产毒菌株耐受盐酸和乳酸的差异性,首先采集309 份牛粪及牛肉样,进行菌株分离鉴定,接着采用多重聚合酶链式反应(polymerase chain reaction,PCR)方法检测分离株及其他收集菌株的4 种毒力基因(eae、hly、stx1、stx2),进而对携带毒力基因的产 毒菌株分别进行盐酸和乳酸应激实验。结果表明:共分离鉴定出8 株大肠杆菌O157菌 株,样品阳性检出率为2.59%;毒力基因检测表明,8 株菌株均不携带stx1和stx2基因,其中6 株菌株携带eae及hly基因;所有产毒菌株耐酸性实验结果表明,盐酸或乳酸处理2 h后20 株产毒菌株存活菌数均显著下降(P<0.05),但下降程度呈现明显的菌株差异性,同一菌株对盐酸、乳酸呈现明显的耐受差异。
关键词:大肠杆菌O157:H7;分离鉴定;耐酸性;菌株差异;产毒菌株
肠出血性大肠杆菌(enterohermorrhangic Escherichia coli,EHEC)是一种重要的人畜共患病原菌,也是食品中常见的食源性致病菌之一,O157:H7是其最主要的血清型[1]。大肠杆菌O157:H7感染剂量非常低,该菌吸附在宿主肠上皮细胞表面并分泌志贺毒素(shiga toxin,Stx),可引起出血性肠炎、继发性溶血性尿毒综合症等严重并发症,重者可导致死亡。大肠杆菌O157:H7对人类健康的威胁已成为主要的世界性公共卫生问题[2-3]。
许多食源性致病菌,如大肠杆菌O157:H7、伤寒沙门氏菌、单增李斯特菌等均为嗜中性菌,即在中性pH值环境下最适合生长,低pH值环境不利于它们的生长繁殖,因此降低pH值是食品加工中抑制致病菌的有效措施。乳酸通常被认为是一种安全的有机酸,欧盟委员会于2013年批准乳酸用于牛胴体表面,以降低微生物污染[4]。此外,机体胃液的低pH值环境可有效杀死食源性致病菌。然而,研究表明大肠杆菌O157:H7具有较高的耐酸性,在pH 2.0~2.5的低酸环境数小时,其生长及繁殖未受到抑制[5-6],这可能是该菌感染剂量较低的原因之一[7]。关于酸应激对食源性致病菌存活的影响,除了与酸种类有关外[8-9],还与pH值、作用温度、持续时间等密切相关[10-11],且无机酸(如HCl)和有机酸(如乳酸)应激引起的微生物失活机制不同[7,12]。较多研究表明,食源性致病菌对各种应激的耐受性存在菌株差异,而实际食品安全风险监测和控制的对象一般是毒力基因阳性的产毒菌株。因此,本研究首先从牛粪及牛肉样中分离、鉴定大肠杆菌O157:H7,选取产毒菌株与前期收集的其他产毒菌株一起进行盐酸和乳酸应激处理,比较不同产毒菌株耐受盐酸和乳酸的差异,为大肠杆菌O157:H7的风险评估和科学有效控制提供科学依据。
1 材料与方法
1.1 菌种与试剂
前期收集了14 株大肠杆菌O157:H7:其中6 株(编号分别为CICC21530、NCTC12900、89、95、JS2和JS3)由南京农业大学江苏省动物源食品生产与安全保障重点实验室惠赠;8 株(编号分别为D1、109、110、111、112、113、114和115)由山东农业大学食品科学与工程学院惠赠。另外,大肠杆菌DH5α由江苏省疾病控制与预防中心惠赠。
改良EC肉汤(mEC+n)(modified E. coli broth)、胰蛋白胨大豆肉汤(tryptic soy broth,TSB)、胰蛋白胨大豆琼脂(tryptic soy agar,TSA)、酵母浸粉(yeast extract,YE)及大肠杆菌O157:H7/NM干制生化鉴定试剂盒 北京陆桥技术有限公司;大肠杆菌O157和H7诊断血清、大肠杆菌O157胶体金快速检测卡 上海慧耘生物有限公司;科马嘉CHROMagarTM O157显色培养基上海欣中生物工程有限公司;抗E. coli O157免疫磁珠北京东方赛瑞公司;聚合酶链式反应(polymerase chain reaction,PCR)相关试剂 日本TaRaKa公司;大肠杆菌O157:H7的rfbO157、fliCH7、eae、hly、stx1和stx2引物的上、下游序列如表1所示[13-14],引物由生工生物工程(上海)股份有限公司合成。
1.2 仪器与设备
IS128C pH计 上海仪迈仪器科技有限公司;LDZX-30FBS立式压力蒸汽灭菌锅 上海申安医疗器械厂;SG403A Sterile GDRD生物安全柜 美国Baker公司;DHP-9052电热恒温培养箱 上海一恒科技有限公司;ZQTY-70台式振荡培养箱 上海知楚仪器有限公司;Mastercycler personal PCR仪 德国Eppendorf公司;Gel Doc 2000 system凝胶成像仪 美国Bio Rad公司;TCL-16G高速离心机 上海安亭仪器厂;2-16KL高速冷冻离心机 美国Sigma公司。
1.3 方法
1.3.1 样品采集
采集安徽3 个养牛场的牛粪样250 份,南京集贸市场及超市牛肉样59 份,共309 份。每份样品分别放入一次性自封袋内,置于低温冰盒内于24 h内送至实验室检 测。
1.3.2 大肠杆菌O157:H7的分离、鉴定
以无菌操作取待检样25 g加入到含有225 mL mEC+n肉汤的均质袋中,在拍击式均质器上连续均质1~2 min,于(36±1) ℃条件下培养24 h,参考GB 4789.36—2016《食品安全国家标准 食品微生物学检验 大肠埃希氏菌O157:H7/NM检验》[15],应用免疫磁珠法富集菌株,于科玛嘉O157显色平板进行分离培养,可疑菌株进行下一步鉴定。
1.3.2.1 特异基因的PCR检测
O157特异基因rfbO157反应体系(25 μL):10×Buffer 2.5 μL、2.5 mmol/L dNTP 2 μL、Mg2+1.5 μL、rfbO157上下游引物各0.5 μL、Taq DNA聚合酶0.15 μL,ddH2O补足。rfbO157基因的反应程序:94 ℃预变性3 min,94 ℃变性1 min,59 ℃退火1 min,72 ℃延伸1 min,30 个循环;72 ℃延伸10 min。H7特异基因fliCH7反应体系25 μL:10×Buffer 2.5 μL、25 mmol/L dNTP 0.5 μL、Mg2+1.5 μL、fliCH7上下游引物各0.25 μL、Taq DNA聚合酶0.125 μL,ddH2O补足。fliCH7基因的反应程序:94 ℃预变性7 min,94 ℃变性1 min,56 ℃退火1 min,72 ℃延伸1 min,30 个循环;72 ℃延伸5 min。引物序列及扩增片段大小如表1所示。
表1 引物序列及扩增片段大小
Table 1 Primers sequences and corresponding amplifified fragment size
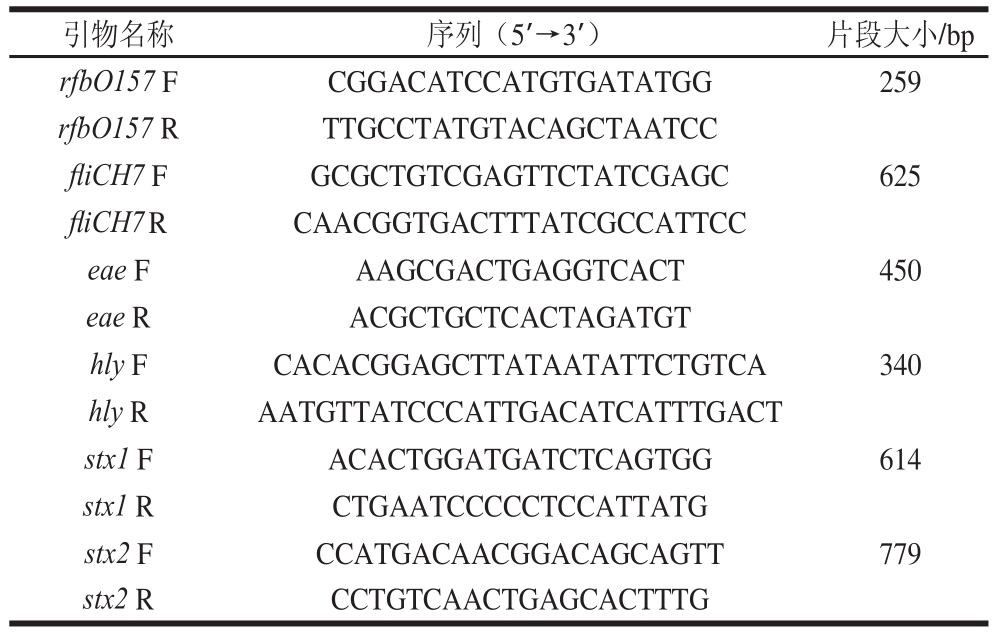
引物名称 序列(5’→3’) 片段大小/bp rfbO157 F CGGACATCCATGTGATATGG 259 rfbO157 R TTGCCTATGTACAGCTAATCC fliCH7 F GCGCTGTCGAGTTCTATCGAGC 625 fliCH7 R CAACGGTGACTTTATCGCCATTCC eae F AAGCGACTGAGGTCACT 450 eae R ACGCTGCTCACTAGATGT hly F CACACGGAGCTTATAATATTCTGTCA 340 hly R AATGTTATCCCATTGACATCATTTGACT stx1 F ACACTGGATGATCTCAGTGG 614 stx1 R CTGAATCCCCCTCCATTATG stx2 F CCATGACAACGGACAGCAGTT 779 stx2 R CCTGTCAACTGAGCACTTTG
1.3.2.2 生化鉴定
对O157特异基因rfbO157PCR反应为阳性的可疑菌株,采用北京陆桥公司的DBI-06大肠埃希氏菌O157:H7/NM干制生化鉴定试剂盒进行生化鉴定。
1.3.2.3 血清学鉴定
对O157特异基因rfbO157 PCR反应为阳性的可疑菌株,用O157和H7诊断血清作玻片凝集实验。对于H7因子血清不凝集者,穿刺接种半固体琼脂,检查动力,经连续传代3 次,动力实验均为阴性,确定为无动力株。
1.3.2.4 免疫胶体金快速检测
对O157特异基因rfbO157 PCR反应为阳性的可疑菌株,采用上海慧耘公司的Prajina大肠杆菌O157快速检测卡进行鉴定。
1.3.3 毒力基因检测
对前期收集的14 株及上述分离鉴定的菌株进行4 种毒力基因(stx1、stx2、eae、hly)多重PCR扩增,以大肠杆菌CICC21530为阳性对照,以大肠杆菌DH5α为阴性对照,PCR反应体系(50 μL):10×Buffer 5 μL、10 mmol/L dNTP 1 μL、25 mmol/L Mg2+5 μL、eae上下游引物各1.5 μL、hly上下游引物各0.2 μL、stx1上下游引物各0.8 μL、stx2上下游引物各2.2 μL、Taq DNA聚合酶0.25 μL,ddH2O补足。四重PCR反应程序:94 ℃预变性7 min,94 ℃变性40 s,56 ℃退火40 s,72 ℃延伸40 s,25 个循环;72 ℃延伸5 min。
1.3.4 产毒菌株耐酸性比较实验
根据毒力基因检测结果,选取毒力基因阳性菌株分别进行盐酸、乳酸耐 受性比较。
菌液制备:将本实验室分离鉴定的菌株及购买和收集的菌株经TSA划线培养,37 ℃培养24 h,挑取单菌落接种于3 mL TSB中,37 ℃培养18 h,再转接至100 mL TSB中,37 ℃培养18~20 h。
盐酸和乳酸应激处理:预实验发现,采用盐酸酸化的TSB(pH 3.5)处理菌液时,细菌存活率均较高,故进一步采用盐酸酸化的TSB(pH 2.0,模拟胃酸)、乳酸酸化的TSB(pH 3.5,模拟一些酸性食品)进行酸应激实验。
取活化2 次后的稳定期菌液1 0 m L(约9(lg(CFU/mL))),于4 ℃、9 000×g条件下离心5 min,用磷酸盐缓冲液(phosphate buffered saline,PBS)(pH 7.2)洗涤2 次,菌体沉淀分别重悬于等体积的盐酸酸化的TSB(pH 2.0)和乳酸酸化的TSB(pH 3.5)中,37 ℃应激反应2 h,立即于上述条件下离心,沉淀用PBS洗涤重悬(终止酸应激)[16],用TSA-YE进行培养计数。实验重复3 次,取平均值,按照下式进一步计算存活率。
1.4 数据处理
菌株存活数用lg(CFU/mL)表示,采用SPSS V17.0软件进行单因素方差分析,结果显著性使用Duncan’s多重比较检验,P<0.05表示存在显著性差异。酸应激后菌株存活率采用GraphPad Prism 7软件进行作图。
2 结果与分析
2.1 菌株初步鉴定
大肠杆菌O157:H7在科玛嘉显色培养基上呈浅红色至淡紫色,中心灰褐色。大肠菌群呈铁蓝色,变形杆菌等为无色至灰色。结果表明,309 份样品中存在疑似菌株的有8 份,其中7 份为牛粪样,1 份为牛肉样。分离纯化得到的疑似菌株进行下一步鉴定。
2.2 基因rfbO157和f l iCH7的PCR扩增
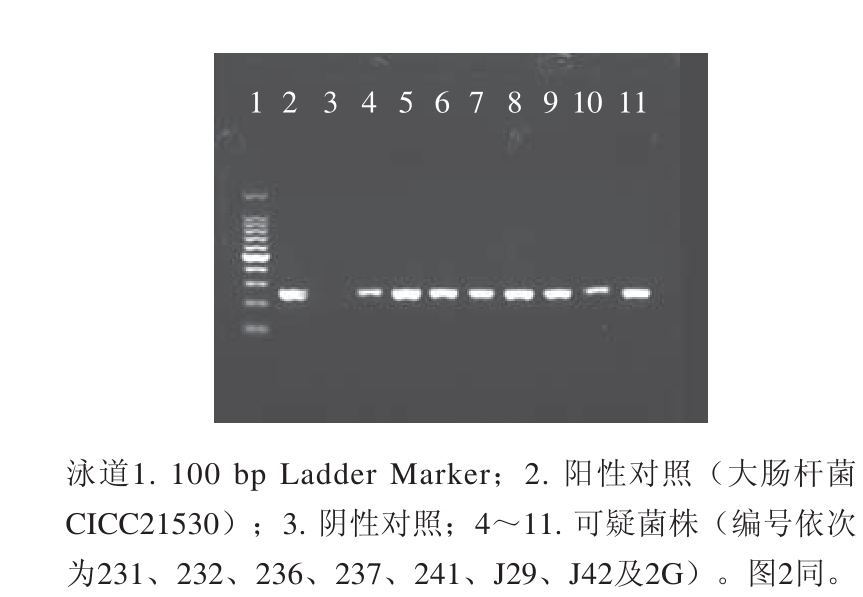
采用PCR方法对疑似菌株进行O型和H型鉴定,以大肠杆菌CICC21530为阳性对照菌株,结果如图1~2所示,8 株菌的rfbO157基因均为阳性,其中6 株fliCH7基因阳性,2 株fliCH7基因阴性(菌株J29和菌株2G)。表明8 株菌鉴定为大肠杆菌O157,其中6 株菌为大肠杆菌O157:H7。
2.3 生化鉴定结果
上述8 株菌株进一步用大肠杆菌O157:H7/NM干制生化鉴定试剂盒进行生化鉴定,结果表明:8 株菌株均能够发酵葡萄糖、乳糖、麦芽糖和甘露醇;发酵蔗糖,产酸不产气;M-R实验、吲哚实验阳性;VP实验、柠檬酸盐利用实验阴性;不产生硫化氢。以上生化结果与大肠杆菌O157的生化特性相符合。
2.4 血清学鉴定结果
对PCR鉴定得到的8 株菌进行血清学鉴定,以菌株CICC21530为阳性对照,结果表明,8 株菌均可与O157诊断血清发生凝集,其中有6 株可与H7诊断血清发生凝集,2 株与H7诊断血清不发生凝集(菌株J29和菌株2G),血清学实验结果与PCR鉴定结果一致。
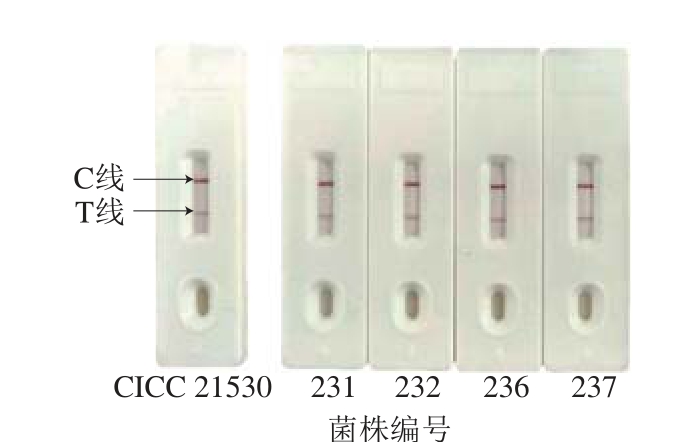
2.5 免疫胶体金快速检测结果
采用大肠杆菌O157胶体金快速检测卡对上述8 株菌株进一步进行检测,以菌株CICC21530为阳性对照。阳性结果:C线显色,T线肉眼可见;阴性结果:C线显色,T线不显色。结果表明,8 株菌均呈阳性,与rfbO157基因的PCR鉴定结果(图1)一致。部分菌株检测结果如图3所示。
综合以上实验结果,本研究检出大肠杆菌O157阳性的样品共8 份,阳性检出率为2.59%(8/309),其中牛粪阳性检出率为2.80%(7/250),牛肉阳性检出率为1.69%(1/59),牛粪阳性检 出率高于牛肉。陈雅君等[17]报道,粪便样中阳性检出率为0.67%,而牛乳和鲜肉中阳性检出率为0%。Uhitil等[18]也报道,在牛肉和牛肉制品中没有检测到大肠杆菌O157。然而,另一些研究报道,在南非和马来西亚的牛肉样中,大肠杆菌O157阳性检出率分别为74.5%和36.0%[19-20],显著高于本研究中牛肉样的阳性检出率。杨一群等[21]也报道,117 份食品样中有55 份为rfbO157基因阳性,即大肠杆菌O157阳性检出率高达47.01%。上述研究报道的差异与地区、牛群饲养管理、屠宰环境及屠宰工艺等有关。本研究的牛肉中检出大肠杆菌O157,提示应在加强防控大肠杆菌O157粪源污染的同时,更应高度关注生肉及相关食品中致病菌的安全监测。
2.6 毒力基因检测结果
表2 8 株分离菌株的PCR检测结果Table 2 PCR results of 8 isolates

注:+. 扩增出阳性条带,-. 未扩增出阳性条带。下同。
菌株编号 rfbO157 f l iCH7 stx1 stx2 eae hly 231++--++232++--++236++--++237++--++241++--++J42++--++J29+-----2G +-----
上述分离得到的8 株大肠杆菌O157,以菌株CICC21530为阳性对照,以菌株DH5α为阴性对照,采用多重PCR扩增4 种毒力基因。由图4和表2可知,8 株菌株均呈stx1和stx2基因阴性,6 株菌株呈eae和hly基因阳性,菌株J29和2G这2 株H7阴性的O157菌株4 种毒力基因均呈阴性。Sallam等[22]报道,在不同的研究中,大多数大肠杆菌O157:H7均携带eae基因,甚至有一些研究发现所有分离株均携带eae和hly基因[23-24]。本研究也表明,大多数分离株携带eae和hly基因,但均不携带stx基因,表明未分离到产志贺毒素大肠杆菌。Lorenz等[25]研究表明,hly基因对大肠杆菌O157:H7存在志贺毒素是一个很好的流行病学标记。因此,不能忽视携带eae和hly基因大肠杆菌O157:H7的污染。
表3 14 株菌株毒力基因的PCR检测结果
Table 3 PCR results of virulence genes of 14 collected strains
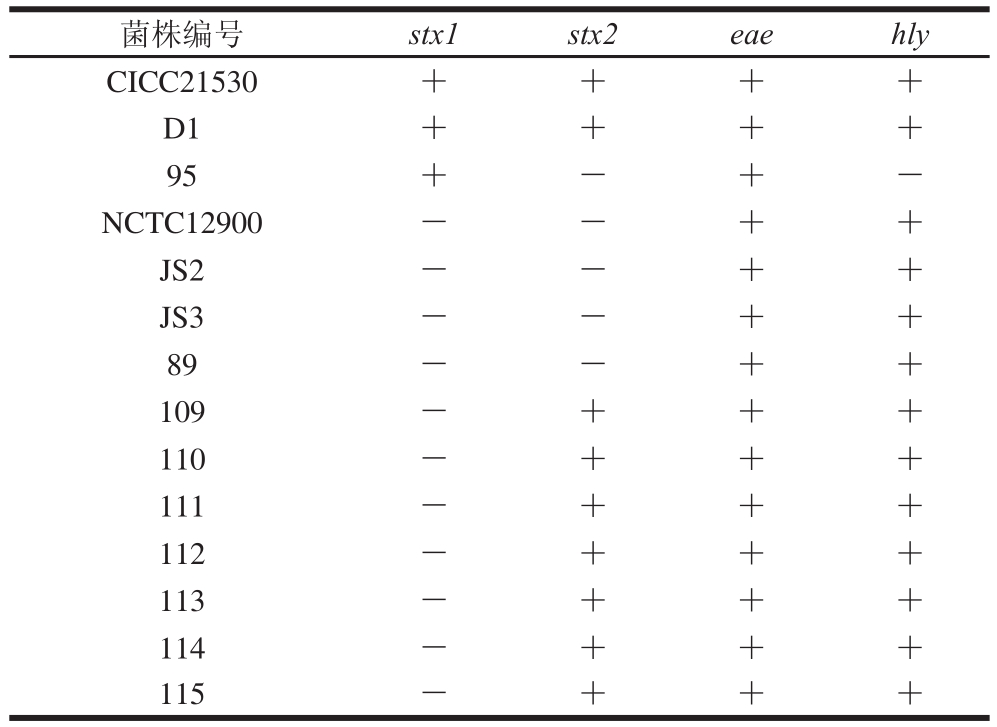
菌株编号 stx1 stx2 eae hly CICC21530 + + + +D1 + + + +95 +-+-NCTC12900--++JS2 --++JS3 --++89 --++109 -+++110 -+++111 -+++112 -+++113 -+++114 -+++115 -+++
对本实验室前期收集的14 株大肠杆菌O157也进行了4 种毒力基因的多重PCR扩增,由图4和表3可知,菌株CICC21530和D1的4 种毒力基因均呈阳性,菌株95的stx1和eae基因呈阳性,菌株NCTC12900、JS2、JS3、89的eae及hly基因呈阳性,其他7 株菌株的stx2、eae及hly基因呈阳性。
2.7 不同产毒菌株耐酸性比较实验结果
对上述分离鉴定的6 株产毒大肠杆菌O157:H7和前期收集的14 株产毒菌株,共20 株菌株进行盐酸(pH 2.0)和乳酸(pH 3.5)耐受实验。
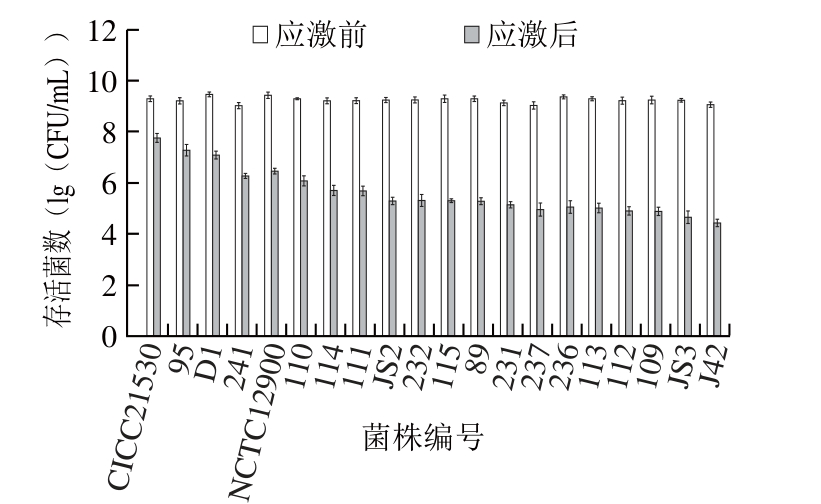
由图5可知,盐酸处理2 h后20 株菌株的存活菌数均显著下降(P<0.05),下降程度呈现明显菌株差异性,存活菌数下降1.52~4.62 (lg(CFU/mL)),从菌株CICC21530到菌株J42下降程度逐渐增大。进一步计算存活率,由图7可知,菌株CICC21530存活率最高(3.0%),菌株J42存活率最低。
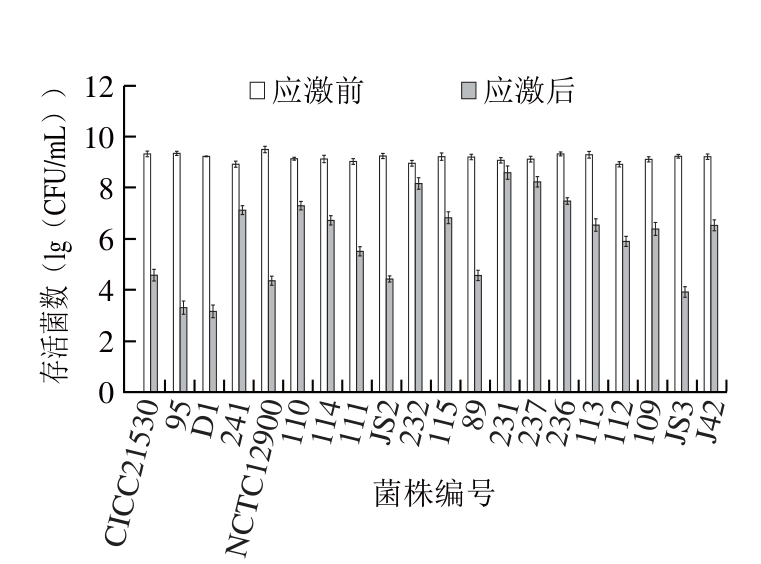
由图6~7可知,乳酸处理2 h后20 株菌株的存活菌数均显著下降(P<0.05),但与盐酸应激结果差异明显,从菌株CICC21530到菌株J42存活菌数下降程度呈现波动变化,其中菌株231下降程度最小(0.44 (lg(CFU/mL))),存活率最高(36.2%),菌株9 5下降程度最大(6.02 (lg(CFU/mL))),存活率最低。
综合图5~7可知,大肠杆菌O157:H7对盐酸应激和乳酸应激的耐受存活情况明显不同,例如,菌株241具有较高的盐酸和乳酸应激耐受能力,菌株CICC21530、95、D1耐受盐酸而不耐受乳酸,菌株231、232耐受乳酸而不耐盐酸,而菌株JS3既不耐受盐酸也不耐受乳酸。实验结果表明,大肠杆菌O157:H7对盐酸和乳酸的应激呈现明显的菌株差异,而同一菌株对盐酸、乳酸呈现明显的耐受差异,这应该与无机酸(盐酸)和有机酸(乳酸)诱导菌株产生的应激响应机制不同有关[7,12]。
3 结 论
从309 份牛粪及牛肉样品中分离鉴定大肠杆菌O157:H7,通过选择性培养增菌和平板分离、PCR检测、生化鉴定、血清学方法和免疫胶体金快速检测,共鉴定出8 株大肠杆菌O157,阳性检出率为2.59%。采用多重PCR检测毒力基因发现,8 株菌株均不携带stx1和stx2基因,6 株菌株呈eae及hly基因阳性。20 株产毒菌株经盐酸或乳酸处理后菌数均显著下降,但下降程度呈现明显的菌株差异性,而同一菌株对盐酸、乳酸呈现明显的耐受差异性。本实验结果提示,应加强环境和食品中大肠杆菌O157:H7的监测和控制,更应重视产毒菌株应激耐受差异的存在,以保证其风险评估和控制更加科学有效。
参考文献:
[1] MAGWEDERE K, DANG H A, MILLS E W, et al. Incidence of Shiga toxin-producing Escherichia coli strains in beef, pork,chicken, deer, boar, bison, and rabbit retail meat[J]. Journal of Veterinary Diagnostic Investigation, 2013, 25(2): 254-258.DOI:10.1177/1040638713477407.
[2] DEWEY-MATTIA D, MANIKONDA K, HALL A J, et al. Surveillance for foodborne disease outbreaks-United States, 2009-2015[J]. MMWR Surveillance Summaries, 2018, 67(10): 1-11. DOI:10.15585/mmwr.ss6710a1.
[3] MAJOWICZ S E, SCALLAN E, JONES-BITTON A, et al.Global incidence of human Shiga toxin-producing Escherichia coli infections and deaths: a systematic review and knowledge synthesis[J]. Foodborne Pathogens and Disease, 2014, 11(6): 447-455.DOI:10.1089/fpd.2013.1704.
[4] European Commission. Commission Regulation (EU) No 101/2013 of 4 February 2013: concerning the use of lactic acid to reduce microbiological surface contamination on bovine carcasses[S].
[5] YIN F G, ZHU Y, KOUTCHMA T, et al. Inactivation and potential reactivation of pathogenic Escherichia coli O157:H7 in apple juice following ultraviolet light exposure at three monochromatic wavelengths[J]. Food Microbiology, 2015, 5(46): 329-335.DOI:10.1016/j.fm.2014.08.015.
[6] 李小媚, 桂荣, 钟俊良, 等. 大肠杆菌O157:H7耐酸性及酸胁迫下菌体形态变化的初步研究[J]. 食品安全质量检测学报, 2018, 9(7):1501-1506. DOI:10.3969/j.issn.2095-0381.2018.07.006.
[7] WI S M, YOON J W. Acid resistance mechanisms in enterohemorrhagic Escherichia coli O157:H7[J]. Journal of Preventive Veterinary Medicine, 2018, 42(3): 124-132. DOI:10.13041/jpvm.2018.42.3.124.
[8] PAUDYAL R, BARNES R H, KARATZAS K A G. A novel approach in acidic disinfection through inhibition of acid resistance mechanisms;Maleic acid-mediated inhibition of glutamate decarboxylase activity enhances acid sensitivity of Listeria monocytogenes[J]. Food Microbiology, 2018, 69: 96-104. DOI:10.1016/j.fm.2017.07.013.
[9] MOHAN A, POHLMAN F W. Role of organic acids and peroxyacetic acid as antimicrobial intervention for controlling Escherichia coli O157:H7 on beef trimmings[J]. LWT-Food Science and Technology,2016, 65: 868-873. DOI:10.1016/j.lwt.2015.08.077.
[10] KIM C, BUSHLAIBI M, ALREFAEI R, et al. Inf l uence of prior pH and thermal stresses on thermal tolerance of foodborne pathogens[J].Food Science and Nutrition, 2019, 7(6): 2033-2042. DOI:10.1002/fsn3.1034.
[11] DIVYA K H, VARADARAI M C. Growth kinetics of a native toxigenic isolate of Yersinia enterocolitica CFR 2301 under the inf l uence ofincubation temperature, pH, sodium chloride and sodium nitrite[J]. Journal of Food Science and Technology-Mysore, 2015,52(11): 7014-7025. DOI:10.1007/s13197-015-1867-3.
[12] CHUNG H J, BANG W, DRAKE M A. Stress response of Escherichia coli[J]. Comprehensive Reviews in Food Science and Food Safety,2006, 5(3): 52-64. DOI:10.1111/j.1541-4337.2006.00002.x.
[13] QIN X, KLEIN E J, GALANAKIS E, et al. Real-time PCR assay for detection and differentiation of shiga toxin-producing Escherichia coli from clinical samples[J]. Journal of Clinical Microbiology, 2015,53(7): 2148-2153. DOI:10.1128/JCM.00115-15.
[14] 陈道利, 许彦梅, 罗霞, 等. PCR方法检测EHEC O157:H7的rfbO157、fliCH7、hlyA、eaeA、stx2及其变种基因[J]. 中国卫生检验杂志, 2005, 15(10): 1197-1200. DOI:10.3969/j.issn.1004-8685.2005.10.018.
[15] 中华人民共和国国家卫生和计划生育委员会, 国家食品药品监督管理总局. 食品安全国家标准 食品微生物学检验 大肠埃希氏菌O157:H7/NM检验: GB 4789.36—2016[S]. 北京: 中国标准出版社,2016.
[16] YOSHITOMI K J, ZAPATA R, JINNEMAN K C, et al. Recovery of E. coli O157 strains after exposure to acidification at pH 2[J]. Letters in Applied Microbiology, 2012, 54(6): 499-503. DOI:10.1111/j.1472-765X.2012.03250.x.
[17] 陈雅君, 王亚宾, 张莉娟, 等. 动物源性肠出血性大肠杆菌O157:H7及其3 个毒力基因的多重PCR快速检测研究[J]. 中国人兽共患病学报, 2013, 29(7): 686-691. DOI:10.3969/cjz.j.issn.1002-2694.2013.07.009.
[18] UHITIL S, JAKSIĆ S, PETRAK T, et al. Presence of Escherichia coli O157:H7 in ground beef and ground baby beef meat[J]. Journal of Food Protection, 2001, 64(6): 862-864. DOI:10.4315/0362-028X-64.6.862.
[19] VORSTER S M, GREEBE R P, NORTJE G L. Incidence of Staphylococcus aureus and Escherichia coli in ground beef, broilers and processed meats in pretoria, South Africa[J]. Journal of Food Protection, 1994, 57(4): 305-310. DOI:10.4315/0362-028X-57.4.305.
[20] RADU S, ABDUL M S, RUSUL G, et al. Detection of Escherichia coli O157:H7 in the beef marketed in Malaysia[J]. Applied and Environmental Microbiology, 1998, 64(3): 1153-1156.
[21] 杨一群, 陈洋, 杜文芳, 等. 食品中大肠杆菌O157:H7疑似菌株的分离和鉴定研究[J]. 中国酿造, 2014, 33(6): 46-49. DOI:10.11882/j.issn.0254-5071.2014.06.011.
[22] SALLAM K I, MOHAMMED M A, AHDY A M, et al. Prevalence,genetic characterization and virulence genes of sorbitol-fermenting Escherichia coli O157:H- and E. coli O157:H7 isolated from retail beef[J]. International Journal of Food Microbiology, 2013, 165(3):295-301. DOI:10.1016/j.ijfoodmicro.2013.05.024.
[23] DONG Pengcheng, ZHU Lixian, MAO Yanwei, et al. Prevalence and characterization of Escherichia coli O157:H7 from samples along the production line in Chinese beef-processing plants[J]. Food Control,2015, 54(1): 39-46. DOI:10.1016/j.foodcont.2015.01.038.
[24] AYAZ N D, GENCAY Y E, EROL I. Prevalence and molecular characterization of sorbitol fermenting and non-fermenting Escherichia coli O157:H7+/H7- isolated from cattle at slaughterhouse and slaughterhouse wastewater[J]. International Journal of Food Microbiology, 2014, 174: 31-38. DOI:10.1016/j.ijfoodmicro.2014.01.002.
[25] LORENZ S C, SON I, MAOUNOUNEN-LAASRI A, et al. Prevalence of hemolysin genes and comparison of ehxA subtype patterns in shiga toxin-producing Escherichia coli (STEC) and non-STEC strains from clinical, food, and animal sources[J]. Applied and Environmental Microbiology, 2013, 79(20): 6301-6311. DOI:10.1128/AEM.02200-13.
Isolation and Identification of Escherichia coli O157:H7 from Cattle and Comparison of Their Acid Tolerance
FU Wenjing, JI Saisai, YU Jinlong, ZHANG Su, XU Yuanyuan, JIANG Yun*
(School of Food Science and Pharmaceutical Engineering, Nanjing Normal University, Nanjing 210023, China)
Abstract:To investigate the variability of the tolerance of Shiga toxin-producing Escherichia coli O157:H7 to hydrochloric acid and lactic acid, suspected colonies w ere isolated from 309 fecal and beef samples and identified in this study. The isolates and another 14 strains collected for this study were detected for 4 major virulence genes (eae, hly, stx1, stx2) by multiplex polymerase chain reaction, and each of the Shiga toxin-producing strains with virulence genes was separately exposed to hydrochloric acid and lactic acid stress. The results showed that 8 isolates of E. coli O157 were identified and the prevalence of positive samples was 2.59%. The results of virulence genes showed that the stx1 and stx2 genes were detected in none of the 8 strains and the eae and hly genes were detected in 6 of them. The results of acid tolerance showed that the survival rates of 20 Shiga toxin-producing strains including the 6 strains and the other 14 above were all significantly decreased (P < 0.05) after treatment with hydrochloric acid or lactic acid for 2 h. However, this effect varied between strains,and the tolerance of the same strain to different acid treatments was different.
Keywords: Escherichia coli O157:H7; isolation and identification; acid tolerance; inter-strain variation; Shiga toxinpr oducing strains
收稿日期:2019-06-19
基金项目:国家自然科学基金面上项目(31671915);江苏省“六大人才高峰”项目(NY-080)
第一作者简介:付文静(1993—)(ORCID: 0000-0002-9339-0962),女,硕士研究生,研究方向为动物源食品质量安全。E-mail: 1356781676@qq.com
*通信作者简介:江芸(1971—)(ORCID: 0000-0002-1195-1843),女,教授,博士,研究方向为动物源食品质量安全。E-mail: jiangyun@njnu.edu.cn
DOI:10.7506/rlyj1001-8123-20190619-139
中图分类号:TS201.3
文献标志码:A
文章编号:1001-8123(2019)07-0001-06
引文格式:付文静, 姬赛赛, 禹金龙, 等. 牛源性大肠杆菌O157:H7的分离鉴 定及其耐酸性比较研究[J]. 肉类研究, 2019, 33(7): 1-6.DOI:10.7506/rlyj1001-81 23-20190619-139. http://www.rlyj.net.cn
FU Wenjing, JI Saisai, YU J inlong, et al. Isolation and ientification of Escherichia coli O157:H7 from cattle and comparison of their acid tolerance[J]. Meat Research, 2019, 33(7): 1-6. DOI:10.7506/rlyj1001-8123-20190619-139. http://www.rlyj.net.cn