鳕鱼隶属脊椎动物门(Vertebrata)、真骨鱼纲(Division Teleostei)、鳕形目(Gadiformes),是生活在海洋底层和深海中下层的冷水鱼类[1]。鳕鱼是经济价值极高的海洋鱼类,具有高蛋白、低脂肪、富含多不饱和脂肪酸、牛磺酸及多种维生素的特点,品质优于其他深海鱼类[2-3]。鳕鱼属包括大西洋鳕鱼(Gadus morhua)、格陵兰鳕鱼(Gadus ogac)和太平洋鳕鱼(Gadus macrocephalus)3 种。阿拉斯加狭鳕鱼(Theragra chalcogramma)属于狭鳕属,与鳕鱼属亲缘关系较近,在SN/T 3589.7—2013《出口食品中常见鱼类及其制品的鉴伪方法 第7部分:鳕鱼成分检测实时荧光PCR法》[4]中将其归为鳕鱼类,但阿拉斯加狭鳕鱼的市售价格远低于大西洋鳕鱼。白姑鱼(Argyrosomus argentatus)为鲈形目(Perciformes)鱼类,经加工处理后外观与大西洋鳕鱼难以区分。由于生态环境变化和过度捕捞,大西洋鳕鱼在市场上较为稀缺[5],而阿拉斯加狭鳕鱼和白姑鱼年产量相对较高,价格低廉。在商业利益驱动下,错误标签、掺假销售等现象时有发生[6-7],以廉价的阿拉斯加狭鳕鱼或白姑鱼替换大西洋鳕鱼,不仅严重损害消费者利益,而且增加了食品安全风险。
鳕鱼物种传统的鉴定方法是通过观察生物形态、研究遗传特点等鉴定物种类别[8]。而商品鳕鱼通常经过去头、冷冻切片等加工后进行销售,鳕鱼形态学特征缺失,外形鉴定方法难以达到对鳕鱼或鳕鱼加工产品真假或掺假鉴别的要求。基于核酸分子对物种进行鉴定的方法打破了传统鉴定方法的局限性[9],DNA序列不随物种加工过程发生变化,在水产品及其加工产品物种鉴定中得到广泛应用。采用环介导等温扩增(loop-mediated isothermal amplification,LAMP)[10-11]、DNA条形码[12-14]、 多重聚合酶链式反应(polymerase chain reaction,PCR)[15-16]、 微卫星标记[16]等技术可以鉴定鲑科鱼[10]、海参[13]、 鳖[14]、鳟鱼[15]、鳙鱼[16]等物种。
自Hebert等[17]应用线粒体细胞色素c氧化酶亚基Ⅰ(cytochrome c oxidase subunit Ⅰ,COⅠ)基因进行物种鉴定以来,DNA鉴定技术在食品掺假、物种鉴定方面起到重要作用。毕潇潇等[18]针对太平洋鳕鱼、阿拉斯加狭鳕鱼、远东宽突鳕和蓝鳕的COⅠ基因片段进行PCR扩增,得到长度均为601 bp的产物;万超[19]、孙晓飞[20]等均针对COⅠ基因设计特异性引物,建立太平洋鳕鱼和大西洋鳕鱼的SYBR Green荧光PCR检测方法;Fernandes等[21]通过对COⅠ基因区域的分析,开发了一种能够更快速、有效分辨4 种鳕鱼的实时定量PCR检测方法。
限制性内切酶与PCR相结合的技术常用于区分亲缘关系较近的物种,陈双雅等[22]扩增细胞色素b(cytochrome b,Cyt b)基因464 bp片段,并对PCR产物采用Dde Ⅰ、Hae Ⅲ、Nla Ⅲ限制性内切酶进行酶切,建立横纹东方鲀、黄鳍东方鲀、菲律宾叉鼻鲀、怀氏兔头鲀及月兔头鲀5 种河豚的物种鉴定方法;Sumathi等[23]将16S rRNA基因的PCR产物采用Sdu Ⅰ、Bci Ⅵ、Sau3 AⅠ限制性内切酶进行酶切,鉴别5 个石斑鱼品种。
目前,国内外对鳕形目鱼类DNA特异性的研究较少,为了更加有效确保鳕鱼产品质量,建立一种实用性强、准确性高的鳕鱼DNA特异性检测方法是必要的。本研究选取鳕鱼线粒体COⅠ基因作为鉴定片段进行比对分析,大西洋鳕鱼和阿拉斯加狭鳕鱼COⅠ基因序列匹配度高达96.91%,设计鳕鱼种间特异性引物PCR难以直接鉴别大西洋鳕鱼和阿拉斯加狭鳕鱼,需在此基础上结合限制性内切酶酶切技术对鳕鱼进行进一步物种鉴定。旨在考察利用COⅠ基因鉴别鳕鱼的可行性,以建立一种有效、快速鉴别真假鳕鱼的方法,为鳕鱼及其产品物种防伪及保护的分子鉴定进行有益探索,为海产品贸易的商业打假提供技术支撑。
1 材料与方法
1.1 材料与试剂
大西洋鳕鱼、阿拉斯加狭鳕鱼(原产于冰岛)、白姑鱼(台州海域捕捞的野生白姑鱼)购于市场专营店。冷冻鱼肉样品置于-20 ℃冰箱贮藏备用。
DNA提取所需混合裂解液(含STE缓冲液(pH 8.0,由1 0 m m o l/L 三羟甲基氨基甲盐酸(Tr i s-H C l)(pH 8.0)、100 mmol/L NaCl、1 mmol/L乙二胺四乙酸(ethylenediamine tetraacetic acid,EDTA)(pH 8.0)组成)和20 g/100 mL十二烷基磺酸钠(sodium dodecyl sulfonate,SDS),其中STE缓冲液、SDS溶液体积比为25∶1)、酚-氯仿-异戊醇(25∶24∶1,V/V)、氯仿-异戊醇(24∶1,V/V) 杭州诺扬生物技术有限公司;脱氧核苷三磷酸(deoxy-ribonucleoside triphosphate,dNTPs)溶液(10 mmol/L)、Taq DNA聚合酶、ddH2O、MgCl2、琼脂糖、蛋白酶K(12 mg/mL)、无水乙醇、BstE Ⅱ限制性内切酶(B300155-0001)、1×TE(Tris-EDTA)缓冲溶液 生工生物工程(上海)有限公司。
1.2 仪器与设备
T960 PCR仪 杭州晶格仪器有限公司;核酸电泳仪 美国Bio-Rad公司;Tanon-3500 10T5553-825凝胶成像 系统 上海天能科技有限公司;ND2000微量核酸蛋白 分析仪、PICO 17台式离心机 美国Thermo公司;HH系列数显恒温水浴锅 江苏省金坛市科析仪器有限公司;XH-C漩涡混合器 江苏省金坛市白塔新宝仪器厂。
1.3 方法
1.3.1 DNA提取
在参考酚-氯仿抽提法提取DNA[24-25]的基础上,优化反应体系、合并试剂并简化步骤[26],按下列方法提取DNA。
称取约100 mg鱼肉,粉碎后装于2 mL离心管中,加入550 μL混合裂解液和15 μL蛋白酶K(12 mg/mL), 漩涡振荡数秒,置于56 ℃水浴锅水浴30 min;取上层清液于新管中,加入5 0 0 μ L 酚-氯仿-异戊醇 (25∶24∶1,V/V),漩涡振荡数秒,10 000 r/min离心4 min;吸取上清液于新管中,加入500 μL氯仿-异戊醇(24∶1,V/V),漩涡振荡数秒,10 000 r/min离心3 min;吸取上清液于新管中,加入50 μL氯化钠溶液(5 mol/L)和1 000 μL无水乙醇,漩涡振荡数秒,10 000 r/min离心3 min;弃去液体,加入600 μL体积分数75%乙醇,漩涡振荡数秒,10 000 r/min离心3 min;弃去液体,开盖将离心管反扣在吸水纸上静置数分钟;加入100 μL 1×TE缓冲液,静置片刻使DNA溶解;使用 ND-2000微量核酸蛋白分析仪测定提取的DNA浓度与纯度,记录并标记后置于-20 ℃冰箱保存。
1.3.2 PCR引物设计
从NCBI数据库下载多个大西洋鳕鱼、阿拉斯加狭鳕鱼、白姑鱼的DNA线粒体基因全序列,利用DNAman软件进行比对分析,从中选择COⅠ基因用于后续引物设计。以大西洋鳕鱼(X99772)、阿拉斯加狭鳕鱼(AB182300)、白姑鱼(HQ890946)的COⅠ基因序列进行比对。依据物种间的特异性区段,使用DNAman软件设计得到多组引物,结合特异性引物的基本要求和实验可行性筛选并经人工矫正得到的最终特异性引物如表1所示,大西洋鳕鱼、阿拉斯加狭鳕鱼扩增产物序列理论长度为579 bp,白姑鱼扩增产物序列理论长度为399 bp。
表 1 PCR扩增特异性引物
Table 1 specific primer sequences used for PCR amplification

扩增物种 引物名称 引物序列(5’-3’)大西洋鳕鱼、阿拉斯加狭鳕鱼codF1 CTCCATCTTTCCTGCTCCT codR1 CGTCCATCCCAACTGTAAAC白姑鱼 argF2 GAATAGTAGGCACAGCCCTG argR2 CCAGAATAGAAGAGACACCTGC
50 μL PCR扩增体系组成如表2所示。PCR管中体系10 000 r/min离心1 min,使体系沉降,减少气泡,有利于扩增。PCR仪循环程序设置为:95 ℃变性30 s,52 ℃退火40 s,72 ℃延伸30 s,共35 个循环,最后72 ℃延伸10 min。收集扩增产物进行电泳凝胶检测和酶切备用。
表 2 50 μL PCR扩增体系组成
Table 2 Composition of 50 μL PCR amplification system

成分 加入量/μL上游引物F(10 μmol/L) 2下游引物R(10 μmol/L) 2 10×PCR Buffer 5 dNTPs(10 mmol/L) 1 Taq DNA聚合酶 1模板DNA 3 Mg2+(100 mmol/L) 3 ddH2O 33总体积 50
1.3.3 限制性内切酶酶切
PCR产物用BstE Ⅱ限制性内切酶进行酶切,30 μL酶切体系组成:10 μL PCR产物、16 μL ddH2O、2 μL Buffer、2 μL BstE Ⅱ限制性内切酶原液(10 U/μL)。酶切条件:37 ℃酶切处理1 h,酶切产物65 ℃灭活30 min后进行凝胶电泳检测。
1.3.4 PCR产物测序与比对
取PCR产物送至生工生物工程(上海)有限公司测序,测序结果与GenBank核酸数据库选择设计出的COⅠ基因序列进行比较分析。
2 结果与分析
2.1 不同鱼肉样品COⅠ基因序列的PCR扩增结果及分析
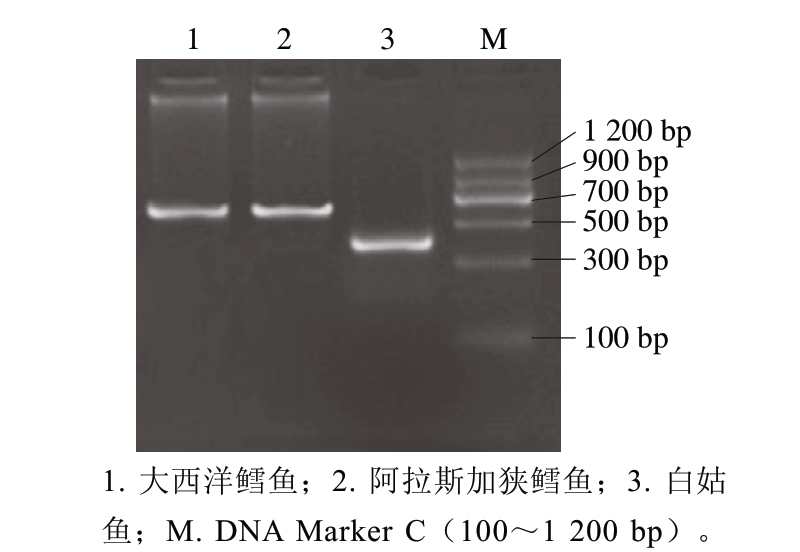
图 1 不同鱼肉样品COⅠ基因序列的PCR扩增结果
Fig. 1 PCR amplification results of COⅠ gene sequence from different fish species
由图1可知,大西洋鳕鱼、阿拉斯加狭鳕鱼与白姑鱼COⅠ基因序列的PCR扩增产物长度与理论大致相符,经测序得到的结果和实验设计预期结果一致。因此可以依据COⅠ基因序列的PCR产物条带有效区别鳕鱼的优劣及其掺假物种白姑鱼。
2.2 不同鱼肉样品COⅠ基因序列PCR扩增产物的酶切

图 2 不同鱼肉样品COⅠ基因目标片段的引物和酶切位点
Fig. 2 Primers and restriction sites for target fragments of COⅠ genes
为了进一步鉴别阿拉斯加狭鳕鱼和大西洋鳕鱼,在阿拉斯加峡鳕鱼COⅠ基因序列的PCR扩增产物片段中寻找BstE Ⅱ限制性内切酶(5’-G/GTNACC-3’)酶切位点,通过酶切分析对产物进行进一步区分。由图2可知,阿拉斯加峡鳕鱼COⅠ基因在676~682 bp位置的序列与酶切位点一致,可以被酶切为361 bp和214 bp 2 个片段,而大西洋鳕鱼在681 bp位点上的碱基为T,与BstE Ⅱ识别的靶序列不同,不能被酶切。
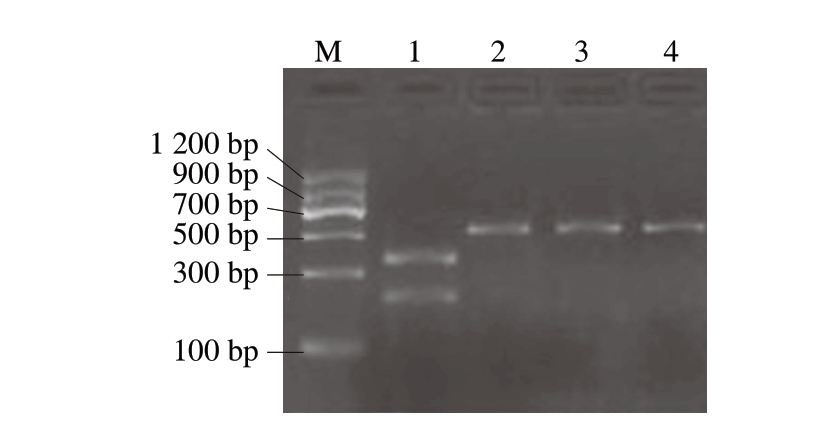
图 3 不同鱼肉样品COⅠ基因序列PCR扩增产物酶切片段的 PCR扩增结果
Fig. 3 PCR amplification results of restriction enzyme digestion fragments of PCR amplified products of COⅠ gene from different fish species
M. DNA Marker C(100~1 200 bp);1. 阿拉斯加狭鳕鱼COⅠ基因序列PCR产物酶切片段;2. 阿拉斯加狭鳕鱼COⅠ基因序列PCR产物;3. 大西洋鳕鱼COⅠ基因序列PCR产物酶切片段;4. 大西洋鳕鱼COⅠ基因序列PCR产物。
由图3可知:大西洋鳕鱼COⅠ基因序列PCR扩增产物无酶切位点,酶切处理后产物的条带长度仍约为579 bp;阿拉斯加狭鳕鱼COⅠ基因序列PCR扩增产物经过酶切处理后,产物包括长度约为361、218 bp的2 个片段,说明在PCR基础上运用酶切技术能够区别大西洋鳕鱼及阿拉斯加狭鳕鱼。
3 讨 论
中国的食品标签受到众多法律、法规和标准的制约。依据强制性国家标准GB 7718—2011《预包装食品标签通则》,产品名称和成分表是预包装产品鱼类信息的主要来源。国家质量监督检验检疫总局关于修改《食品标识管理规定》的决定(总局2009年第123号令)[27] 涉及国内生产(分装)食品的标识标注和管理。2015年10月1日生效的《中国食品安全法》修订版进一步严苛了食品安全监管,然而大多数海鲜标签和可追溯性的具体标准都是非强制性标准,仍然缺乏关于渔业产品标签的具体规定和海产品贸易名称的正式参考清单[28]。在中国市场上新推出的海洋渔业产品中,鳕鱼是最受欢迎的产品之一,鳕鱼的掺伪问题也广泛引起关注,如含有蜡油质的油鱼(Pseudogyrincheilus procheilus)、低价的阿拉斯加狭鳕鱼、白姑鱼或其他与鳕鱼种属相关但来历不明的物种但因标记不明晰进入市场,对鳕鱼或其加工制品进行掺伪的行为在世界各国均已有报道[29-30],这些行为增加了鳕鱼及其制品含有过敏源或有毒有害物质的风险。
在食品贸易全球化时代,海产品通常经过复杂的物流网到达销售地,难以追溯原产地,依据加工海产品的残留特征也难以进行形态鉴定[28]。为了支持海产品标准文件的可追溯性,已经提出许多基于DNA的鉴定方法。例如,基于680 bp COⅠ基因区域的DNA条形码技术[31]是最常用的方法之一[17-21,32-34]。COⅠ基因较容易被通用引物扩增,能够获得绝大多数动物的5’端序列,具有稳健性[35-36]。其次,COⅠ基因相较于其他线粒体基因具有更大范围的系统发育信号[31]。COⅠ基因相较于编码核糖体(12S、16S)DNA的线粒体基因不易出现插入和缺失[31],并且与其他蛋白质编码基因一样,其第3位核苷酸显示出高碱基取代发生率,导致分子进化速率加快,这种进化速率优势使其不仅可以用于区分密切相关的物种,还可以用于区分单一物种内的系统地理群[37-38]。不同品种鳕鱼与其易混淆鱼类COⅠ基因序列的相似度在78.1%~80.6%之间[6]。王敏等[39]以COⅠ基因为目标基因,应用DNA条形码技术鉴别77 份深圳批发市场和超市零售鱼肉制品的来源,结果表明,产品“错贴”率高达36.36%。Cawthom等[40]利用DNA条形码技术研究南非市场的鱼类,发现在批发商和零售商中贴错标签的比例差别巨大。虽然通过DNA条形码技术可以对鱼的种类进行鉴定,但是检测用引物是通用引物,对于市场上的部分加工制品,对混合DNA进行鉴定需要将多个PCR产物纯化后与载体连接,转化培养后挑选单克隆菌落,再经过测序达到鉴定目的,过程繁琐且存在极大的漏检可能 性[41]。本研究设计2 组PCR引物鉴别白姑鱼,结合内切酶方法,能够直接根据电泳图上条带长度的差异判定PCR产物能否被限制性酶切,达到鉴别鳕鱼的目的,即使对混合DNA的复杂样品也能实现成分的有效鉴定。
SN/T 3589.7—2013详述了利用实时荧光PCR法对出口食品中鳕鱼成分进行检测的方法,可以用于太平洋鳕鱼、大西洋鳕鱼、黑线鳕、青绿鳕、蓝鳕、青鳕、狭鳕、南非无须鳕、阿根廷无须鳕及北太平洋无须鳕等鳕鱼及其制品的定性检测。现有鳕鱼标准检测方法不能够覆盖整个鳕形目[41],且所述方法的PCR产物片段较短,长度大约为80 bp,实验过程需严格要求无DNA酶污染,并防止反应产物的交叉污染。目前,鱼肉制品的鉴定技术主要还包括近红外光谱技术、简单重复序列(simple sequence repeats,SSR)标记(微卫星标记)技术、扩增片段长度多态性(amplified fragment length polymorphism,AFLP)技术等[42-43]。近红外光谱技术能够较为快速地鉴别鱼肉制品,然而需要应用建模工具,在应用方面具有局限性[6];SSR标记技术在杂合子和纯合子方面具有优势,但SSR标记的等位基因数目较多,在DNA指纹识别上具有难度[42-43];操作简便的AFLP技术的谱带错配率和高成本是其限制性因素[43]。Dooley等[44] 应用PCR-限制性片段长度多态性(restriction fragment length polymorphism,RFLP)和基因芯片技术,基于Cyt b 基因对鉴定10 种不同英国鳕鱼产品,利用Dde Ⅰ限制酶区分了8 个鱼种,当与Nla Ⅲ和Hae Ⅲ 2 种酶组合时,产生了所有10 个鱼种的特定谱,说明PCR与RFLP技术联用在鱼种鉴定方面具有有效性。本研究在对样品进行鱼种鉴定时,应用PCR结合限制性内切酶对特定片段的特定位点进行识别切割,通过电泳图的条带大小进行鉴定,大部分鉴定结果均与目的结果相吻合,说明本研究中的方法对于初步鉴别大西洋鳕鱼、阿拉斯加狭鳕鱼和白姑鱼具有实际意义,无需复杂昂贵的检测仪器,是海产品物种防伪及保护分子鉴定方面的有益探索。在稳定本研究结果和灵敏度的基础上,未来可探求PCR技术与基因芯片技术、时间温度梯度电泳等相结合的联用技术,以期实现对成分单一或复杂的鳕鱼肉制品的更准确、快速鉴别[45-46]。
4 结 论
结合PCR扩增技术和限制性内切酶酶切技术设计了一种可以有效鉴别大西洋鳕鱼、阿拉斯加狭鳕鱼和白姑鱼的方法,通过对待测鱼种进行DNA提取,对其进行PCR扩增实验,实现了大西洋鳕鱼和阿拉斯加狭鳕鱼2 种鳕鱼与白姑鱼的鉴别;在此基础上运用限制性内切酶技术,结果表明,阿拉斯加狭鳕鱼PCR产物被酶切成长度约为361 bp和218 bp的两部分,从而可以有效将其与大西洋鳕鱼进行区分。该方法特异性高、简便实用、检测时间短,可满足市场监管和检验的适用性要求,具有较大的推广价值和应用前景,对维持鳕鱼市场健康发展及维护广大消费者利益具有重要意义。
[1] 孟庆闻, 苏锦祥. 缪学祖鱼类分类学[M]. 北京: 中国农业出版社, 1985: 380-407.
[2] 邵胜荣, 郭利萍. 鳕鱼肝油中维生素A、D含量检测方法的探讨[J]. 食品科技, 2013(11): 276-279.
[3] 于琴芳, 邓放明. 鲢鱼小黄鱼鳕鱼和海鳗肌肉中营养成分分析及 评价[J]. 农产品加工(学刊), 2012(9): 11-14. DOI:10.3969/j.issn.1671-9646(X).2012.09.003.
[4] 中国检验检疫科学研究院, 中华人民共和国上海出入境检验检疫局, 中华人民共和国山东出入境检验检疫局. 出口食品中常见鱼类及其制品的鉴伪方法 第7部分: 鳕鱼成分检测实时荧光PCR法: SN/T3589.7—2013[S]. 北京: 中国标准出版社, 2013.
[5] GONZ LEZ-IRUSTA J M, WRIGHT P J. Spawning grounds of Atlantic cod (Gadus morhua) in the north sea[J]. ICES Journal of Marine Science, 2015, 73(2): 304-315. DOI:10.1093/icesjms/fsv180.
LEZ-IRUSTA J M, WRIGHT P J. Spawning grounds of Atlantic cod (Gadus morhua) in the north sea[J]. ICES Journal of Marine Science, 2015, 73(2): 304-315. DOI:10.1093/icesjms/fsv180.
[6] 李新光, 王璐, 赵峰, 等. DNA条形码技术在鱼肉及其制品鉴别中的应用[J]. 食品科学, 2013, 34(18): 337-342. DOI:10.7506/spkx1002-6630-201318069.
[7] 石蕊寒, 南汇珠, 丁红田, 等. DNA条形码技术鉴别市售鱼肉制品真伪[J]. 肉类研究, 2018, 32(2): 40-45. DOI:10.7506/rlyj1001-8123-201802007.
[8] 高天翔, 武云飞, 张秀梅, 等. 四种鳕鱼的形态学研究[J]. 中国海洋大学学报(自然科学版), 2002, 32(6): 884-890. DOI:10.3969/j.issn.1672-5174.2002.06.023
[9] YANG Fan, DING Fei, CHEN Hon, et al. DNA barcoding for the identification and authentication of animal species in traditional medicine[J]. Evidence-Based Complementary and Alternative Medicine, 2018(7): 1-18. DOI:10.1155/2018/5160254.
[10] 冯俊丽, 叶剑, 孟璐, 等. 环介导等温扩增技术快速检测大西洋鲑的研究[J]. 核农学报, 2017, 31(5): 868-875. DOI:10.11869/j.issn.100-8551.2017.05.0868.
[11] SAULL J, DUGGAN C, HOBBS G, et al. The detection of Atlantic cod (Gadus morhua) using loop mediated isothermal amplification in conjunction with a simplified DNA extraction process[J]. Food Control, 2016, 59: 306-313. DOI:10.1016/j.foodcont.2015.05.038.
[12] 徐春燕, 沈长春, 蔡建堤, 等. 基于COⅠ基因的福建近海部分仔稚鱼DNA条形码分析[J]. 中国水产科学, 2017, 24(6): 1176-1183.
[13] 律迎春. 基于DNA条形码的分子生物学方法鉴定海参种类的研究[D]. 青岛: 中国海洋大学, 2012: 29. DOI:10.7666/d.y2157861.
[14] 唐伟, 朱治任, 汪财生, 等. 基于COⅠ基因的DNA条形码在鳖科动物鉴定上的应用[J]. 江苏农业科学, 2017, 45(6): 30-36. DOI:10.15889/j.issn.1002-1302.2017.06.006.
[15] RASMUSSEN HELLBERG R S, MORRISSEY M T, HANNER R H. A multiplex PCR method for the identification of commercially important salmon and trout species (Oncorhynchus and Salmo) in north America[J]. Journal of Food Science, 2010, 75(7): C595-C606. DOI:10.1111/j.1750-3841.2010.01752.x.
[16] 耿波, 孙效文, 梁利群, 等. 利用17 个微卫星标记分析鳙鱼的遗传多样性[J]. 遗传, 2006, 28(6): 683-688. DOI:10.3321/j.issn:0253-9772.2006.06.009.
[17] HEBERT P D N, RATNASINGHAM S, DE WAARD J R. Barcoding animal life: cytochrome coxidase subunit 1 divergences among closely related species[J]. Proceedings of the Royal Society B: Biological Sciences, 2003, 270(Suppl 1): S96-S99. DOI:10.1098/rsbl.2003.0025.
[18] 毕潇潇, 高天翔, 肖永双, 等. 4 种鳕鱼线粒体16S rRNA、COI和Cytb基因片段序列的比较研究[J]. 南方水产科学, 2009, 5(3): 46-52. DOI:10.3969/j.issn.1673-2227.2009.03.008.
[19] 万超, 蒋丹, 吕泉, 等. 太平洋无须鳕鱼及其制品的PCR鉴定[J]. 食品安全质量检测学报, 2017, 8(1): 71-75.
[20] 孙晓飞, 蒋丹, 万超, 等. 建立一种快速鉴定大西洋鳕鱼及其制品的SYBR Green荧光PCR方法[J]. 东北师大学报(自然科学版), 2017, 49(3): 127-130. DOI:10.16163/j.cnki.22-1123/n.2017.03.026.
[21] FEMANDES T J R, COSTA J, OLIVEIRA M B P P, et al. DNA barcoding coupled to HRM analysis as a new and simple tool for the authentication of Gadidae fish species[J]. Food Chemistry, 2017, 230: 49-57. DOI:10.1016/j.foodchem.2017.03.015.
[22] 陈双雅, 陈文炳, 张津, 等. 应用PCR-RFLP和芯片生物分析系统鉴别河豚鱼品种[J]. 食品科学, 2012, 33(22): 200-202.
[23] SUMATHI G, JEYASEKARAN G, JEYA SHAKILA R, et al. Molecular identification of grouper species using PCR-RFLP technique[J]. Food Control, 2015, 51: 300-306. DOI:10.1016/j.foodcont.2014.11.026.
[24] 王继英, 俞英, 冯利霞, 等. 利用改进的酚-氯仿法从猪毛囊中提取基因组DNA[J]. 遗传, 2010, 32(7): 752-756. DOI:10.3724/SP.J.1005.2010.00752.
[25] 姚琳, 李青娇, 江艳华, 等. 水产加工品DNA提取方法的比较[J]. 中国渔业质量与标准, 2013, 3(2): 80-88.
[26] 冯涛. 动物源性成分交叉引物等温扩增快速检测方法的研究和 建立[D]. 杭州: 中国计量大学, 2016: 15-18.
[27] 国家质量监督检验检疫总局. 国家质量监督检验检疫总局关于修改<食品标识管理规定>的决定(总局2009年第123号令)[EB/OL]. (2009-11-12) [2019-08-02]. http://www.aqsiq.gov.cn/xxgk_13386/jlgg_12538/zjgg/2009/200911/t20091112_238391.htm.
[28] XIONG X, D’AMICO P, GUARDONE L, et al. The uncertainty of seafood labeling in China: a case study on cod, salmon and tuna[J]. Marine Policy, 2016, 68: 123-135. DOI:10.1016/j.marpol.2016.02.024.
[29] XIONG X, GUARDONE L, GIUST A, et al. DNA barcoding reveals chaotic labeling and misrepresentation of cod (鳕, Xue) products sold on the Chinese market[J]. Food Control, 2016, 60: 519-532. DOI:10.1016/j.foodcont.2015.08.028.
[30] DALMA J, VIEITES J M, ESPINEIRA M. Detection of the causal agents of Keriorrhea (Lepidocybium falvobrunneum and Ruvettus pretiosus) by means of real time PCR[J]. Food Chemistry, 2014, 174: 326-329. DOI:10.1016/j.foodchem.2014.11.070.
[31] HEBERT P D N, CYWINSKA A, BALL S L, et al. Biological identifications through DNA barcodes[J]. Proceedings of the Royal Society B, 2003, 270: 313-321. DOI:10.1098/rspb.2002.2218.
[32] AMANI A, GUARDONE L, LA CASTELLANA R, et al. DNA barcoding reveals commercial and health issues in ethnic seafood sold on the Italian market[J]. Food Control, 2015, 55: 206-214. DOI:10.1016/j.foodcont.2015.02.030.
[33] CAEVALHO D C, PALHARES R M, DRUMMOND M G, et al. DNA barcoding identification of commercialized seafood in south Brazil: a governmental regulatory forensic program[J]. Food Control, 2015, 50: 784-788. DOI:10.1016/j.foodcont.2014.10.025.
[34] CAWTHOM D M, DUNCAN J, KASTEM C, et al. Fish species substitution and misnaming in south Africa: an economic, safety and sustainability conundrum revisited[J]. Food Chemistry, 2015, 185: 165-181. DOI:10.1016/j.foodchem.2015.03.113.
[35] FOLMER O, BLACK M, HOEH W, et al. DNA primers for amplification of mitochondrial cytochrome coxidase subunit I from diverse metazoan invertebrates[J]. Molecular. Marine. Biology and. Biotechnolog, 3(5): 294-299. DOI:10.4028/www.scientific.net/DDF.7.460.
[36] ZHANG D X, HEWITT G M. Assessment of the universality and utility of a set of conserved mitochondrial COⅠ primers in insects[J]. Insect Molecular Biology, 1997, 6(2): 143-150. DOI:10.1111/j.1365-2583.1997.tb00082.x.
[37] WARES J P, CUNNINGHAM C W. Phylogeography and historical ecology of the North Atlantic Intertidal[J]. Evolution, 2001, 55(12): 2455-2469. DOI:10.1111/j.0014-3820.2001.tb00760.x.
[38] 池宇, 王诗迪, 张春田. 基于线粒体COI基因的双翅目昆虫研究进展[J]. 昆虫分类学报, 2010, 32(增1): 71-77.
[39] 王敏 , 刘荭 , 黄海, 等. DNA条形码技术在深圳鱼肉制品鉴定中的应用[J]. 食品科学, 2015, 36(20): 247-251. DOI:10.7506/spkx1002-6630-201520048.
[40] CAWTHOM D M, STEINMAN H A, WITTHUHN R C. DNA barcoding reveals a high incidence of fish species misrepresentation and substitution on the south African market[J]. Food Research International, 2012, 46(1): 30-40. DOI:10.1016/j.foodres.2011.11.011.
[41] 林霖, 陈国培, 何永盛, 等. 鳕鱼产品属性鉴别及细鳞壮鳕检测方法建立[J]. 食品工业, 2017(4): 208-212.
[42] TELETCHEA F. Molecular identification methods of fish species: reassessment and possible applications[J]. Reviews in Fish Biology and Fisheries, 2009, 19(3): 265-293. DOI:10.1007/s11160-009-9107-4.
[43] 吴潇, 潘玉春, 唐雪明. 肉制品的DNA溯源技术[J]. 猪业科学, 2009, 26(3): 105-106.
[44] DOOLEY J J, SAGE H D, CLARKE M A L, et al. Fish species identification using PCR-RFLP analysis and lab-on-a-chip capillary electrophoresis: application to detect white fish species in food products and an interlaboratory study[J]. Journal of Agricultural and Food Chemistry, 2005, 53(9): 3348-3357. DOI:10.1021/jf047917s.
[45] COLOMBO F, CHESSA S, CATTANEO P. Polymerase chain reaction products (PCR) on “DNA barcode zone” resolved by temporal temperature gradient electophoresis: a tool for species identification of mixed meat specimens-a technical note on preliminary results[J]. Food Control, 2011, 22(8): 1471-1472. DOI:10.1016/j.foodcont.2011.03.007.
[46] HAIDER N, NABULS I, SAFADI B. Identification of meat species by PCR-RFLP of the mitochondrial COI gene[J]. Meat Science, 2012, 90(2): 490-493. DOI:10.1016/j.meatsci.2011.09.013.