克氏原螯虾(Procambarus clarkia)俗称小龙虾,是一种重要的淡水经济虾类,肉质鲜嫩、肥美,广泛分布在有浅滩的地方。自2000年以来,小龙虾餐饮业蓬勃发展,但与之相关的食品安全问题屡见不鲜。2010年,南京23 人因食用小龙虾患横纹肌溶解症(Haff病)[1]。2011年,300余人于瑞昌参加“瑞昌市万人龙虾节”,随后出现副溶血性弧菌群体性食物中毒[2]。与其他水产品相似,小龙虾营养丰富且水分活度高,此外,生鲜小龙虾自身微生物污染严重,在其运输、贮藏过程中易腐败 变质[3-5]。李兵兵等[6]发现,淮安地区所产的生鲜小龙虾带有副溶血性弧菌、金黄色葡萄球菌、沙门氏菌等致病菌。张生元[7]、朱若林[8]等从小龙虾中分离到条件致病菌布氏柠檬酸杆菌。
传统培养基鉴定法是将样品处理稀释液涂布在各类选择性培养基上以获得菌种,纯化分离后再分别提取DNA,最后通过BLAST分析确定菌株类别,但该方法存在明显不足,凭肉眼筛选菌株时,人为主观意识较强,容易遗漏部分细菌。且自然界中可经人工培养的微生物仅约1%,传统培养基鉴定法无法培养出样品中所有细菌[9-10]。相比之下,宏基因组测序可以同时对几十万到几百万条DNA进行测序,能够对样品中所含微生物的基因组进行全面分析[11]。于美娟等[12]采用传统培养基法和宏基因组测序技术确定传统固态发酵鱼冷藏期间的优势腐败菌,结果表明,宏基因组测序检测到的菌属更具有多样性。小龙虾属于变温动物,生存高度依赖水体,不同地区小龙虾所处的生存环境、摄入的食物可能不相同,因此对小龙虾菌群的研究不能仅从某一单因素出发,需综合考虑多种因素[13]。本研究选择泗洪地区的小龙虾为代表,利用传统培养和宏基因组测序方法对生鲜小龙虾整体、小龙虾表面以及小龙虾尾肉的细菌菌群进行分析,找出初始优势菌群,系统分析其潜在的食品安全风险。
1 材料与方法
1.1 材料与试剂
小龙虾(体质量25~35 g),来源于江苏泗洪地区,冷鲜包装快递至实验室后进行样品处理。
平板计数琼脂(plate count agar,PCA)、甘露醇氯化钠琼脂(mannitol salt agar,MSA)、结晶紫中性红胆盐葡萄糖琼脂(violet red bile glucose agar,VRBGA)、营养肉汤、十二烷基硫酸盐胰蛋白酶(lauryl sulfate tryptose,LST)肉汤、脑心浸液(brain heart infusion,BHI) 肉汤 北京奥博星生物技术制造有限责任公司;假单胞菌琼脂基础培养基(CN琼脂) 山东青岛海博生物技术有限公司。
1.2 方法
1.2.1 样品处理
在不进行预清洗的条件下,对小龙虾样品的处理分为3 种:1)以小龙虾整体为样品:称取鲜活小龙虾50 g,打碎后称取10 g置于90 mL无菌生理盐水中,均质30 min后,梯度稀释;2)以小龙虾表面为样品:选取10 只大小均等、质量约180 g的鲜活小龙虾,置于225 mL无菌生理盐水中,均质30 min后,取出小龙虾,保留生理盐水并进行梯度稀释;3)以小龙虾尾肉(带虾线)为样品:无菌剥取10 g小龙虾虾仁,剪碎后置于90 mL无菌生理盐水中,均质30 min后,梯度稀释。
1.2.2 菌数测定及优势腐败菌分离鉴定
按照GB 4789.2—2016《食品安全国家标准 食品微生物学检验 菌落总数测定》进行小龙虾样品菌落总数测定。分别取1.2.1节中合适梯度的稀释液100 µL涂布于PCA(菌落总数计数)、VRBGA(肠杆菌菌落计数)、MSA(葡萄球菌菌落计数)、CN琼脂(假单胞菌菌落计数)平板上,30 ℃恒温培养24 h。
将各类平板上的特征菌落分离纯化,在对应的培养基上划线2 次,获得单菌落后,挑取单菌落于5 mL对应的液体培养基中,30 ℃振荡培养至600 nm波长处的光密度(optical density,OD600 nm)为1左右后收集菌液[14]。将一部分菌液与50%甘油按体积比1∶1混合后放入-80 ℃冰箱中保存;另一部分菌液 10 000 r/min离心2~3 min后弃上清。用100 μL无菌超纯水将沉淀重悬后,100 ℃水浴10 min;再将其10 000 r/min 离心1 m i n,收集上清液备用;取2 μ L 上清液,加入2×Master Mix 20 μL、无菌超纯水14 μL、引物27F(5’-AGAGTTTGATCMTGGCTCAG-3’)与1492R(3’-GGTTACCTTGTTACGACTT-5’)各2 μL,进行16S rDNA的聚合酶链式反应(polymerase chain reaction,PCR) 扩增,取5 μL扩增产物进行1%琼脂糖凝胶电泳,将电泳条带清晰的扩增产物送至生工生物工程(上海)股份有限公司测序[15]。
1.2.3 样品总DNA提取
取1.2.1节中的样品均质液,4 ℃、2 000 r/min离心10 min后,取上清液;然后4 ℃、12 000 r/min离心10 min后,弃上清液,收集沉淀。将提取得到的DNA样品进行琼脂糖凝胶电泳质量检测后,置于-80 ℃保存备用。
1.2.4 MiSep测序和数据分析
将DNA样品送至北京奥维森基因科技有限公司,利用Illumina MiSeq PE300宏基因组测序平台测序。微生物多样性检测选取细菌16S rDNA V3~V4区,该区扩增引物为338F(5’-ACTCCTACGGGAGGCAGCAG-3’)和806R(5’-GGACTACNNGGGTATCTAAT-3’)[16-17]。通过Illumina MiSeq平台进行Paired-end测序,下机数据经过QIIME(v1.8.0)软件过滤、拼接,过滤双端序列数据,收集有效序列并矫正序列方向[18]。根据Barcodes归类各处理组序列信息,聚类为用于物种分类的操作分类单元(operational taxonomic unit,OTU),OTU相似性设置为97%[19]。对比Silva数据库,得到每个OTU对应的物种分类信息。再利用Mothur软件(version 1.31.2)进行Alpha多样性分析[20]。在门、纲、目、科、属等分类水平上对微生物进行群落结构统计,汇总分析新鲜泗洪小龙虾不同部位的初始微生物群落。
1.3 数据处理
所有实验重复测定3 次,采用SPSS 16.0软件进行方差分析。
2 结果与分析
2.1 泗洪小龙虾不同部位的初始菌相分析

图 1 泗洪小龙虾不同部位的微生物数量
Fig. 1 Total number of bacteria in different parts of fresh crawfish
由图1可知,泗洪小龙虾不同部位的优势腐败菌均为肠杆菌。小龙虾整体样品在PCA、VRBGA、MSA和CN琼脂平板上生长的菌落数分别为8.4、7.3、7.2、 7.1 (lg(CFU/g)),小龙虾表面样品分别为7.1、6.7、6.7、6.2 (lg(CFU/g)),小龙虾尾肉样品分别为7.0、6.4、6.1、6.0 (lg(CFU/g))。泗洪小龙虾不同部位的菌落数有一定差别,尾肉样品的菌落数较低。一方面可能是由于小龙虾体表外骨骼中的几丁质具有致密结构,能有效阻拦微生物入侵[21],还可能是由于通过去除虾头和外壳减少了一定数量的微生物[22]。
庄秋丽等[23]发现,刀额新对虾的初始菌落数为2~3 (lg(CFU/g)),段伟文等[24]通过菌落计数发现,凡纳滨对虾的初始菌落数大于4.0 (lg(CFU/g))。初始菌落数的差异可能与虾的品种、养殖环境、实验预处理方式等因素有关。
2.2 泗洪小龙虾不同部位优势腐败菌的分离与鉴定
通过平板划线法,根据4 种平板培养基上菌株的形态,分离得63 株菌。将每株菌进行16S rDNA扩增,对扩增产物进行琼脂糖凝胶电泳检测。在2 000 bp与1 000 bp之间出现特异性条带即为成功扩增样品,对获得特异性条带的PCR产物进行测序分析。将测序所得序列进行NCBI BLAST分析,选取同源性98%以上的菌株序列,确定菌株种属[15],构建系统进化发育树(图2)。
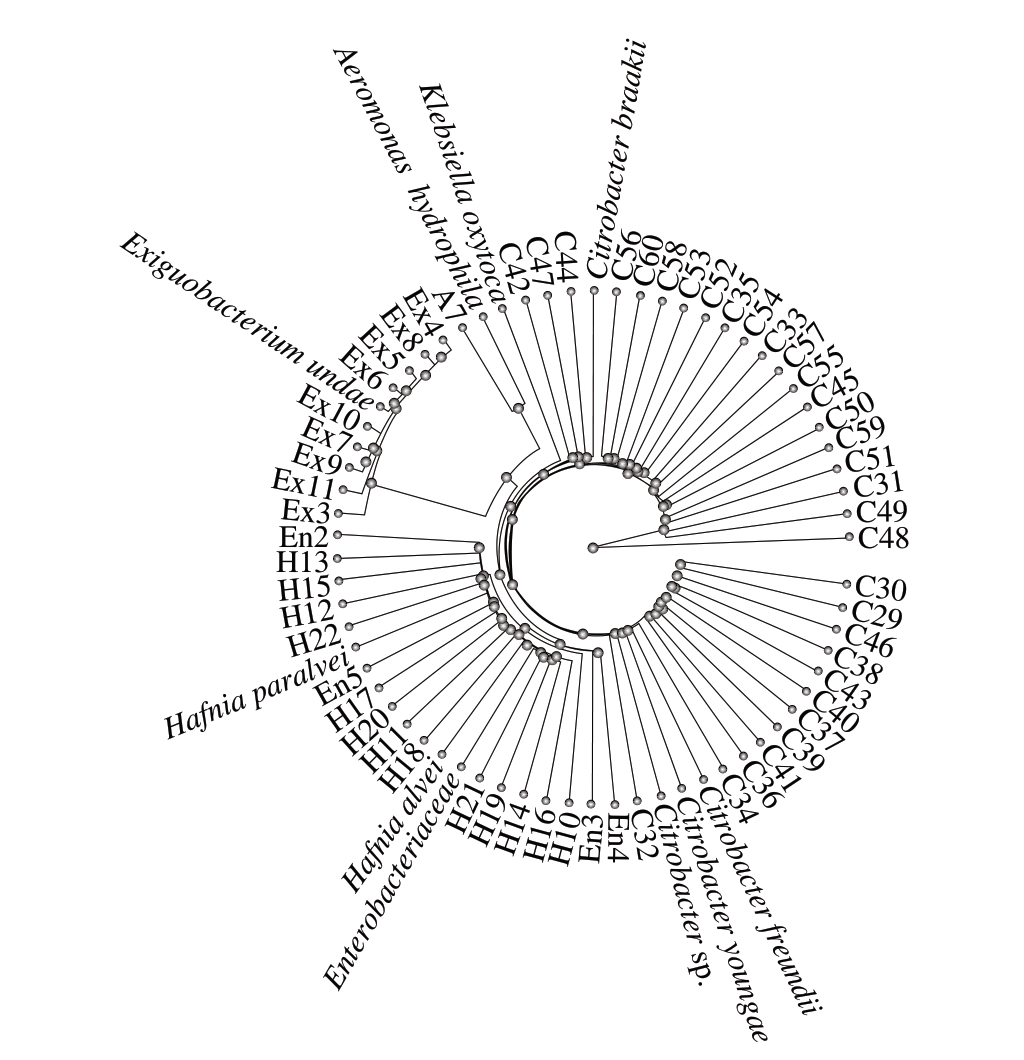
图 2 基于16S rDNA构建的63 株腐败菌的进化发育树
Fig. 2 Phylogenetic tree for 63 strains of spoilage bacteria based on 16S rDNA sequences
由表1可知,泗洪小龙虾不同部位分离所得63 株菌中大多数菌株均属于肠杆菌科,其中32 株菌为柠檬酸杆菌属。柠檬酸杆菌在自然界中普遍存在,易引起水产动物感染发病[25]。曹荣等[26]发现,凡纳滨对虾的主要初始菌群为鞘脂杆菌属(Sphingobacterium)、金黄杆菌属(Chryseobacterium)和寡养单胞菌属(Stenotrophomonas)。Jaaskelainen等[27]发现,三文鱼和金枪鱼中的主要腐败菌分别为发光杆菌属(Photobacterium)和假单胞菌属(Pseudomonas)。 Zhang Jingbin等[28]发现,不动杆菌(Acinetobacter)和假单胞菌(Pseudomonas)是新鲜草鱼片中的主要腐败菌。水产品中主要腐败菌的差异可能与生长环境和物种有关。
表 1 泗洪小龙虾不同部位分离所得菌株的鉴定结果
Table 1 Identification of strains isolated from different parts of fresh crawfish

小龙虾部位 菌株鉴定结果整体副蜂房哈夫尼菌(Hafnia paralvei),H10~H12微小杆菌(Exiguobacterium artemiae),Ex1~Ex8柠檬酸杆菌(Citrobacter sp.),C29~C36表面产气肠杆菌(Enterobacter aerogenes),En2~En5蜂房哈夫尼菌(Hafnia alvei),H13~H18气单胞菌(Aeromonas sp.),A7柠檬酸杆菌(Citrobacter sp.),C37~C45微小杆菌(Exiguobacterium artemiae),Ex9~Ex11产酸克雷伯氏菌(Klebsiella oxytoca),K14尾肉 蜂房哈夫尼菌(Hafnia alvei),H19~H22柠檬酸杆菌(Citrobacter sp.),C46~C60
2.3 泗洪小龙虾不同部位的宏基因组测序结果及OTU分析

图 3 有效序列分布
Fig. 3 Sequence distribution
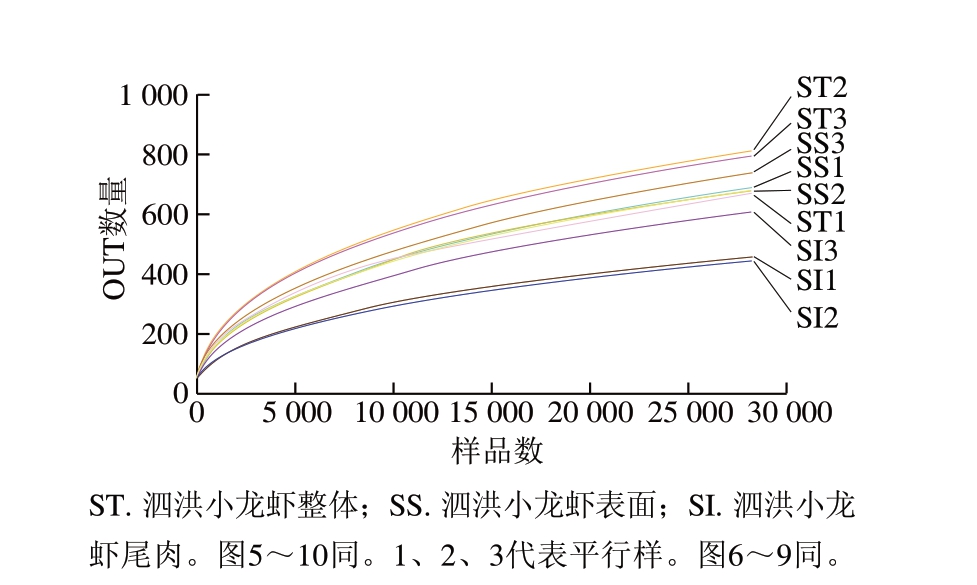
图 4 样品稀释曲线图
Fig. 4 Rarefaction curves for samples
用Trimmomatic及FLASH软件优化通过Illumina MiSeq宏基因组测序获得的新鲜小龙虾不同部位样品总菌的原始序列数据,9 个小龙虾样品共得到674 567 条有效序列。由图3可知,674 567 条有效序列中,序列的平均长度大部分集中在200~260区间内。由图4可知,随着样品数的增加,OTU数量增加,当样品数大于20 000时,OTU曲线趋于平缓,说明测序数据量较为合理,且测序数据量足够大,基本可以反映样品中所有的微生物多样性信息。
2.4 泗洪小龙虾不同部位菌群的Alpha多样性指数分析
Alpha多样性是指某个特定生态系统或区域内的多样性,对样品进行Alpha多样性指数(包括Chao1、Observed_species、PD_whole_tree及Shannon指数)分析可以反映样品中微生物群落的丰度和均匀度。Chao1和Observed_species指数可以反映样品中微生物群落的丰富度,主要反映样品的测序量是否充足;Shannon指数可以反映样品中微生物群落的多样性,PD_whole_tree指数可以反映样品中的微生物群落对进化历史保存的差异[15-16]。
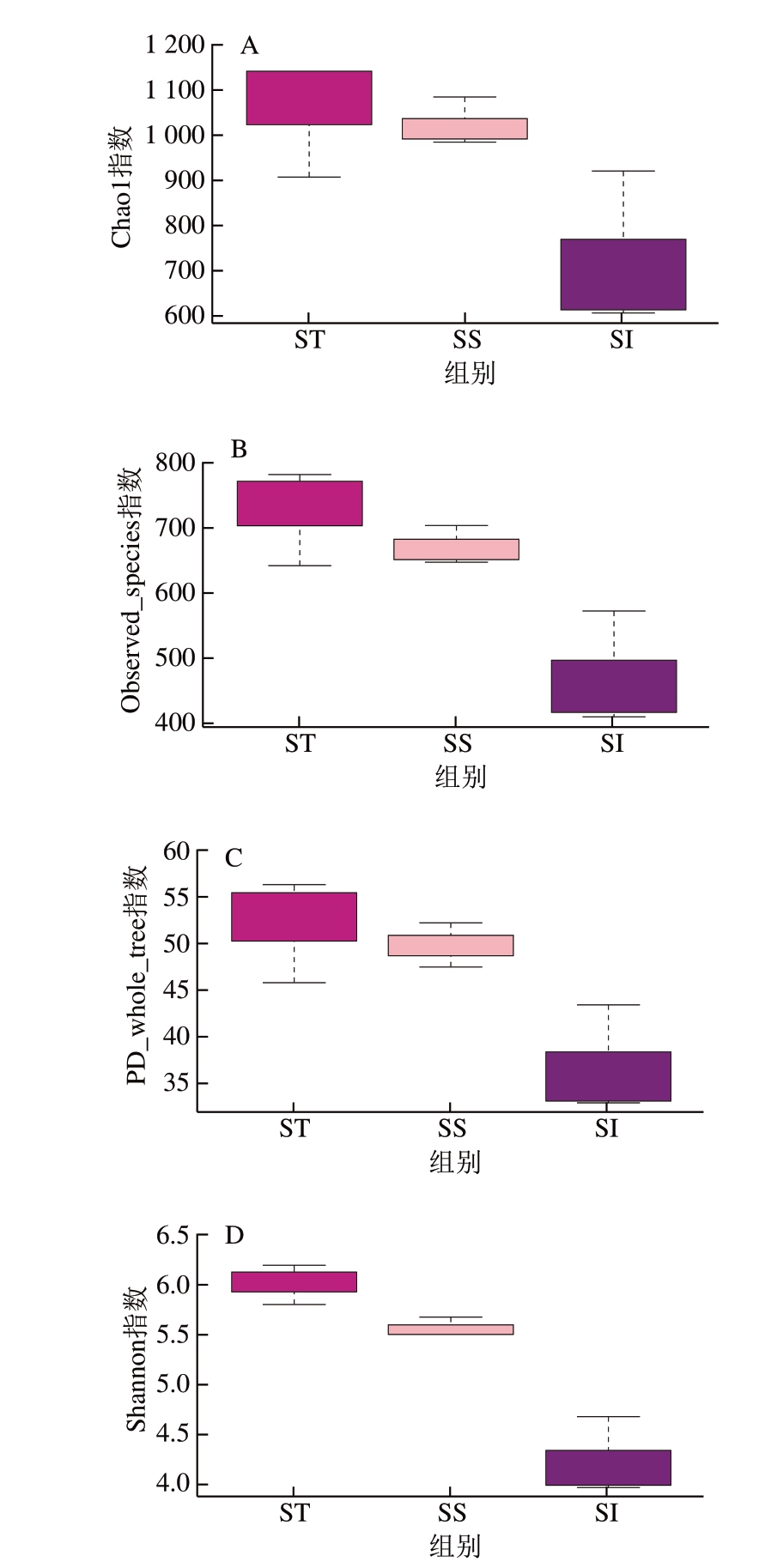
图 5 泗洪小龙虾不同部位菌群Alpha多样性指数箱图
Fig. 5 Box plots for alpha diversity indexes of microflora in different parts of crayfish
由图5可知,3 个部位样品的Chao1和Observed_species 指数均较高,说明样品的测序量较为充足。另外,泗洪小龙虾整体的PD_whole_tree及Shannon指数均最高,说明该批次小龙虾菌群多样性较高,小龙虾尾肉的PD_whole_tree 及Shannon指数与小龙虾整体、小龙虾表面相比较低,而小龙虾与外界直接接触的部位主要是体表、消化器官及肠道,小龙虾的消化器官及肠道是重要的免疫器官,能有效阻止病原微生物的侵入[21],尾肉样品中的菌群主要为小龙虾的肠道菌群,说明该批次小龙虾的肠道菌群多样性较低,这可能与小龙虾自身的免疫机制及所处饲养环境有关。
2.5 泗洪小龙虾不同部位的菌群组成和聚类分析
为了得到每个OTU对应的物种分类信息,采用核糖体数据库项目(ribosomal database project,RDP)Classifier算法对OTU代表序列进行比对分析,并在科、属的水平上注释各样品中菌群的物种信息。
2.5.1 基于科分类水平的分析
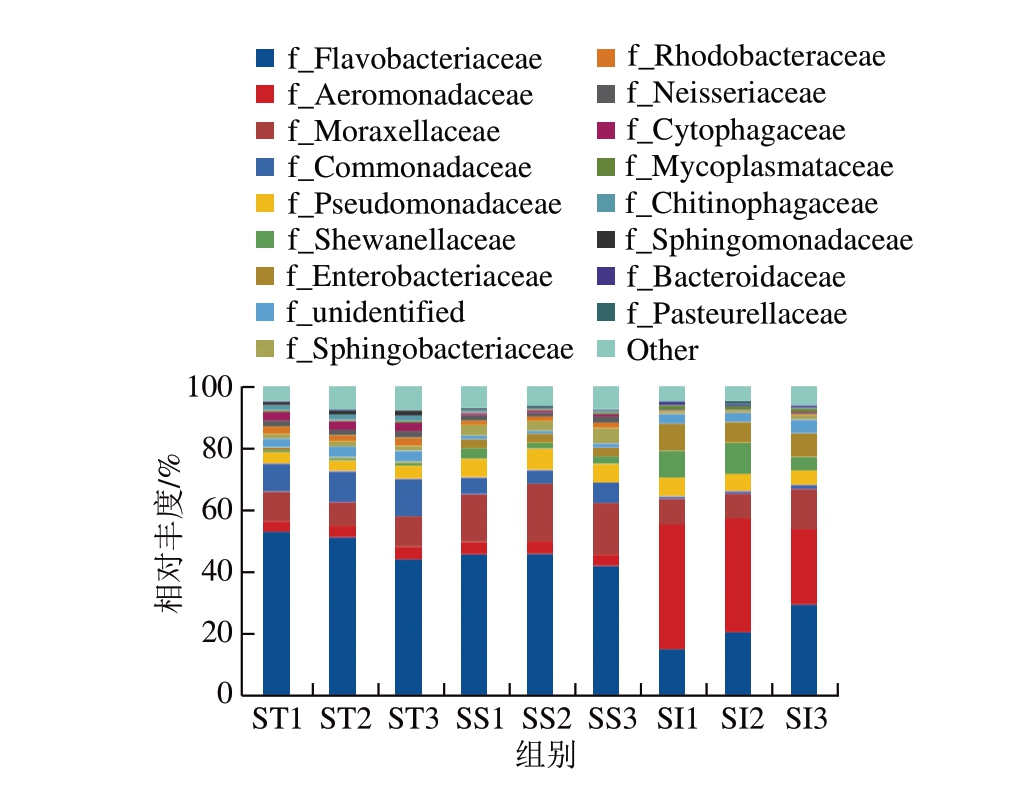
图 6 基于科水平的物种组成分析柱状图
Fig. 6 Bacterial community distribution at the family level
由图6可知,泗洪小龙虾不同部位的菌群组成差异明显。黄杆菌科(Flavobacteriaceae)是泗洪小龙虾整体和表面携带菌群中的主要菌科,而莫拉氏菌科(Moraxellaceae)在表面样品中的相对丰度与丛毛单胞菌科(Comamonadaceae)在整体样品中的相对丰度均超过10%。泗洪小龙虾尾肉中的主要菌群为气单胞菌科(Aeromonadaceae),黄杆菌科(Flavobacteriacea)的相对丰度低于气单胞菌科(Aeromonadaceae)。此外,所有样品中相对丰度高于5%的菌科还有希瓦氏菌科(Shewanellaceae)、肠杆菌科(Enterobacteriaceae)和假单胞菌科(Pseudomonadaceae)。
2.5.2 基于属分类水平的分析

图 7 基于属水平的物种组成分析柱状图
Fig. 7 Bacterial community distribution at the genus level
由图7可知,泗洪小龙虾不同部位的菌群多样性较高,主要有气单胞菌属(Aeromonas)、黄杆菌属(Flavobacterium)、不动杆菌属(Acinetobacter)和金黄杆菌属(Chryseobacterium),3 个部位样品菌群多样性类似,但比例稍有不同。与传统培养基鉴定法分离到的菌属有较大差别,表明宏基因组测序法在一定程度上弥补了传统培养基鉴定法的局限性。其中,泗洪小龙虾整体样品中的主要菌属为黄杆菌属(Flavobacterium)、不动杆菌属(Acinetobacter)、金黄杆菌属(Chryseobacterium)、假单胞菌属(Pseudomonas)和气单胞菌属(Aeromonas),表面样品中的主要菌属为黄杆菌属(Flavobacterium)、不动杆菌属(Acinetobacter)、金黄杆菌属(Chryseobacterium)和假单胞菌属(Pseudomonas),尾肉样品中的主要菌属为气单胞菌属(Aeromonas)、黄杆菌属(Flavobacterium)、不动杆菌属(Acinetobacter)、希瓦氏菌属(Shewanella)和假单胞菌属(Pseudomonas)。
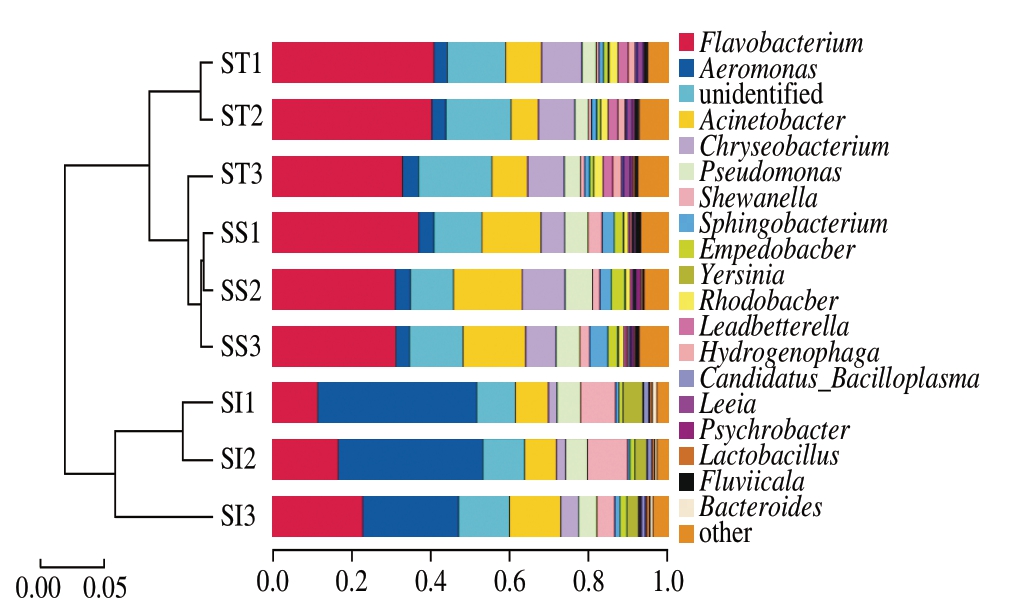
图 8 样本聚类柱状图
Fig. 8 Sample clustering histogram
根据属水平上的物种组成和相对丰度,通过UPGMA方法对各样品进行聚类分析。由图8可知,泗洪小龙虾3 个部位的样品在一个分支上,说明3 个部位样品的菌种组成和相对丰度类似。
2.6 泗洪小龙虾不同部位菌群变化的热图分析
通过可以用颜色变化反映原始数据信息或二维矩阵的热图,将9 个样品的数据简单聚合,直观地用颜色深浅来表示数据大小。不同丰度的物种分块聚焦,通过彩虹色梯度及颜色深浅可以反映全部样品在各分类水平上群落组成的差异性和相似性[29-30]。
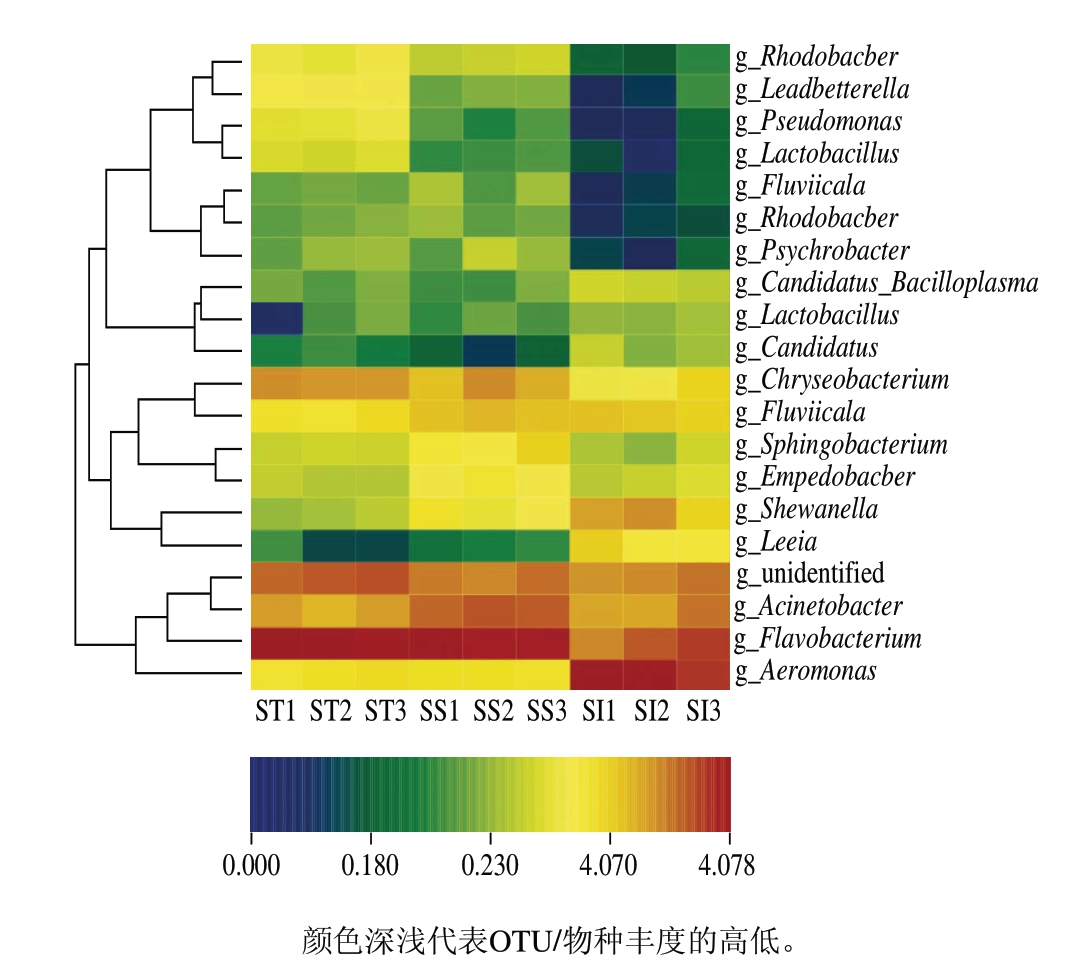
图 9 OUT及其属分类学水平热图
Fig. 9 OUT and heatmap at texonomic levels
由图9 可知,泗洪小龙虾不同部位的优势菌群,如黄杆菌属(Flavobacterium)、不动杆菌属(Acinetobacter)、金黄杆菌属(Chryseobacterium)和假单胞菌属(Pseudomona)等的比例大致相同,但泗洪小龙虾整体与表面的优势菌群类似,泗洪小龙虾尾肉与整体、表面的优势菌群有一定差别。例如,泗洪小龙虾整体和表面中的黄杆菌属(Flavobacterium)和金黄杆菌属(Chryseobacterium)丰度较尾肉高,尾肉中的气单胞菌属(Aeromonas)丰度较整体和表面高。小龙虾养殖场的水体环境中存在着一定量微生物[31],而小龙虾表面中的微生物主要来自小龙虾直接接触的水体环境,饲养环境对小龙虾整体的初始菌群影响较大。
2.7 泗洪小龙虾不同部位菌群的主成分分析
通过主成分分析可以进一步反映不同样品的菌群物种差异。横、纵坐标轴分别表示样品中微生物群落结构组成差异的两大主成分,横轴表示第1主成分,纵轴表示第2主成分,百分数代表主成分对样品差异的贡献率[23]。
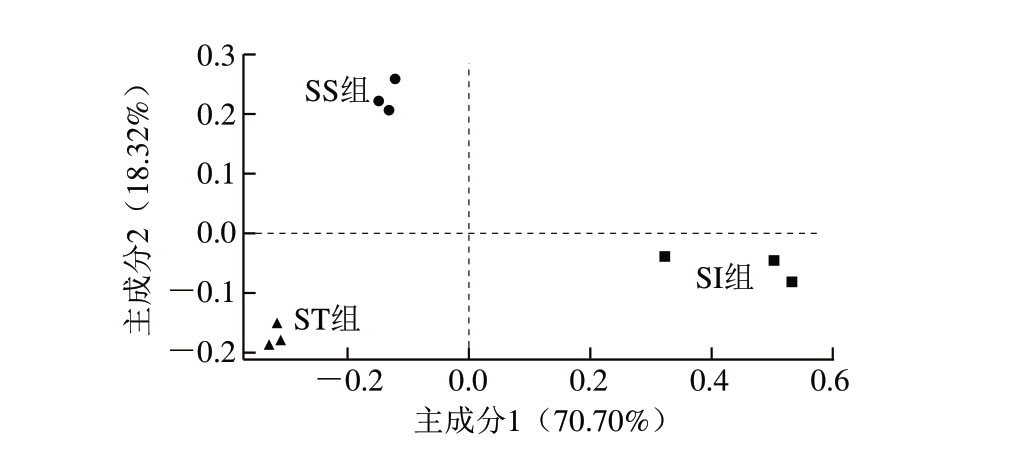
图 10 泗洪小龙虾不同部位菌群的主成分分析
Fig. 10 Principal component analysis of bacterial flora in different parts of fresh crawfish
由图10可知,第1主成分与第2主成分的贡献率分别为70.70%和18.32%,说明2 个主成分能反映所有样品中微生物群落结构的组成差异。泗洪小龙虾样品中微生物群落结构组成越相似,图中样品之间的距离越近。通过纵向和横向观察均发现,泗洪小龙虾尾肉(带虾线)样品与小龙虾整体样品、表面样品之间的距离较远,小龙虾整体样品与表面样品之间的距离较近,与Alpha多样性指数分析结果一致。
3 结 论
以泗洪小龙虾的不同部位为研究对象,运用Illumina MiSeq宏基因组测序方法分析新鲜小龙虾不同部位的菌群组成,结果表明,新鲜小龙虾不同部位所携带的菌群多样性有差异。传统培养基鉴定法分离所得3 个部位小龙虾中的主要细菌为柠檬酸杆菌属(Citrobacter)、克雷伯氏菌属(Klebsiella)、蜂房哈夫尼菌属(Hafnia alvei)和气单胞菌属(Aeromonas)。宏基因组测序结果表明,小龙虾3 个部位的初始菌群组成有一定差别:新鲜小龙虾整体菌群以黄杆菌属(Flavobacterium)、不动细菌属(Acinetobacter)、金黄杆菌属(Chryseobacterium)为主,与小龙虾表面的微生物组成类似;小龙虾尾肉的菌群与整体、表面差别较大,主要由气单胞菌属(Aeromonas)、黄杆菌属(Flavobacterium)、假单胞菌属(Pseudomonas)和希瓦氏菌属(Shewanella)组成。针对不同部位菌群多样性差异,本研究建议对小龙虾的养殖环境进行严格管控,必要时可着重清洗小龙虾表面、去除尾部肠道,以控制小龙虾的初始菌落数。
已有研究表明,柠檬酸杆菌属和气单胞菌属是小龙虾的主要细菌性病原,对小龙虾养殖业有较大 威胁[7,8,32]。而希瓦氏菌属、假单胞菌属和不动细菌属均为水产品等富含蛋白质食品中的常见腐败菌,其中希瓦氏菌属的产硫能力极强,有一定的嗜冷性,低温条件下可迅速繁殖,不利于小龙虾的低温贮藏[33]。本研究为评估小龙虾食品安全质量风险、检测小龙虾质量提供了扎实的理论依据。
[1] 韩丽岚, 徐荣靖. 南京“小龙虾事件”调查处理分析[J]. 中国卫生监督杂志, 2012, 19(1): 75-78. DOI:1007-6131(2012)01-075-04.
[2] 孙吉昌, 游兴勇, 刘成伟, 等. 一起由副溶血性弧菌致群体性食物中毒的调查报告[J]. 中国食品卫生杂志, 2012, 24(1): 89-91. DOI:10.13590/j.cjfh.2012.01.009.
[3] 倪治明. 浙北地区餐饮业小龙虾重点危害因子调查及风险评估[D]. 杭州: 浙江大学, 2013: 4-15.
[4] KAMADIA V V, SCHILLING M W, MARSHALL D L. Cooking and packaging methods affect consumer acceptability and shelflife of ready-to-eat gulf brown shrimp[J]. Journal of Aquatic Food Product Technology, 2013, 22(2): 146-159. DOI:10.1080/10498850.2011.632709.
[5] 王焕庆, 李学英, 杨宪时, 等. 高水分烤虾贮藏过程中的品质变化和菌相分析[J]. 食品与机械, 2012, 28(2): 165-169. DOI:10.3969/j.issn.1003-5788.2012.02.043.
[6] 李兵兵, 刘纯成, 侯海燕, 等. 淮安地区小龙虾及其外环境中致病菌分布规律和耐药性分析[J]. 食品安全质量检测学报, 2016, 7(9): 3530-3534.
[7] 张生元, 郑瑞珠, 艾桃山, 等. 克氏原螯虾源布氏柠檬酸杆菌的分离鉴定及药敏试验[J]. 江苏农业科学, 2018, 46(12): 129-131. DOI:10.15889/j.issn.1002-1302.2018.12.031.
[8] 朱若林, 杨彩桥, 蒋书东, 等. 克氏原螯虾布氏柠檬酸杆菌的分离鉴定及药敏分析[J]. 安徽农业大学学报, 2018, 45(4): 617-620. DOI:10.13610/j.cnki.1672-352x.20180825.029.
[9] DONG Linlin, XU Jiang, ZHANG Lianjuan, et al. High-throughput sequencing technology reveals that continuous cropping of American ginseng results in changes in the microbial community in arable soil[J]. Chinese Medicine, 2017, 12(1): 18. DOI:10.1186/s13020-017-0139-8.
[10] DE MUINCK E J, TROSVIK P, GILFILLAN G D, et al. A novel ultra high-throughput 16S rRNA gene amplicon sequencing librarypreparation method for the Illumina HiSeq platform[J]. Microbiome, 2017, 5(1): 68. DOI:10.1186/s40168-017-0279-1.
[11] 许超. 养殖大鲵肠道菌群结构及肠道细菌药敏性研究[D]. 重庆: 西南大学, 2017: 32-49.
[12] 于美娟, 谭欢, 马美湖, 等. 传统固态发酵鱼中细菌群落多样性与品质特征分析[J]. 食品科学, 2017, 38(8): 86-95. DOI:10.7506/spkx1002-6630-201708015.
[13] YUN J H, ROH S W, WHON T W, et al. Insect gut bacterial diversity determined by environmental habitat, diet, developmental stage, and phylogeny of host[J]. Applied and Environmental Microbiology, 2014, 80(17): 5254-5264. DOI:10.1128/AEM.01226-14.
[14] 郭锦锦, 芦起, 冯蕾, 等. 青蒿琥酯对铜绿假单胞菌生物膜形成及结构的影响[J]. 基因组学与应用生物学, 2018, 37(8): 3733-3739. DOI:10.13417/j.gab.037.003733.
[15] PARLAPANI F F, MEZITI A, KORMAS K A, et al. Indigenous and spoilage microbiota of farmed sea bream stored in ice identified by phenotypic and 16S rRNA gene analysis[J]. Food Microbiology, 2013, 33(1): 85-89. DOI:10.1016/j.fm.2012. 09.001.
[16] 温冬玲, 成淑君, 刘悦, 等. 高通量测序分析不同增菌温度下冷鲜鸡肉细菌的群落多样性[J]. 食品科学, 2018, 39(24): 156-161. DOI:10.7506/spkx1002-6630-201824024.
[17] 高乾坤, 焦琳舒, 杜贺超, 等. 高通量测序分析不同产地带鱼冷藏时微生物群落多样性[J]. 食品科学, 2018, 39(18): 127-132. DOI:10.7506/spkx1002-6630-201818020.
[18] ANDREEVSKAYA M, JOHANSSON P, LAINE P, et al. Genome sequence and transcriptome analysis of meat-spoilage-associated lactic acid bacterium Lactococcus piscium MKFS47[J]. Applied and Environmental Microbiology, 2015, 81(11): 3800-3811. DOI:10.1128/AEM.00320-15.
[19] EDGAR R C. UPARSE: highly accurate OTU sequences from microbial amplicon reads[J]. Nature Methods, 2013, 10(10): 996-999. DOI:10.1038/NMETH.2604.
[20] LU Xiaoming, LU Pengzhen. Characterization of bacterial communities in sediments receiving various wastewater effluents with high-throughput sequencing analysis[J]. Microbial Ecology, 2014, 67(3): 612-623. DOI:10.1007/s00248-014-0370-0.
[21] 刘振兴. 嗜水气单胞菌及环境因子对克氏原螯虾免疫相关指标的影响[D]. 武汉: 华中农业大学, 2011: 1-7.
[22] 邓晓影, 张宾, 汤贺, 等. 基于高通量测序的南美白对虾中微生物群落分析[J]. 食品科学, 2018, 39(24): 149-155. DOI:10.7506/spkx1002-6630-201824023.
[23] 庄秋丽, 林建城, 洪辉集, 等. 溶菌酶复合保鲜剂对刀额新对虾的保鲜作用[J]. 食品工业, 2019, 40(5): 37-41.
[24] 段伟文, 全沁果, 章雪琴, 等. 静电场结合冰温技术对凡纳滨对虾贮藏期品质的影响[J]. 食品与机械, 2018, 34(12): 101-107. DOI:10.13652/j.issn.1003-5788.2018.12.022.
[25] DOMINGOS M O, FRANZOLIN M R, DOS ANJOS M T, et al. The influence of environmental bacteria in freshwater stingray woundhealing[J]. Toxicon, 2011, 58(2): 147-153. DOI:10.1016/j.toxicon. 2011.04.016.
[26] 曹荣, 林瑞环, 赵玲, 等. 基于高通量测序技术分析凡纳滨对虾(Litopenaeus vannamei)冷藏过程菌群结构变化[J]. 食品安全质量检测学报, 2019, 10(8): 2127-2131.
[27] JAASKELAINEN E, JAKOBSEN L M A, HULTMAN J, et al. Metabolomics and bacterial diversity of packaged yellowfin tuna (Thunnus albacares) and salmon (Salmo salar) show fish speciesspecific spoilage development during chilled storage[J]. International Journal of Food Microbiology, 2019, 293: 44-52. DOI:10.1016/j.ijfoodmicro.2018.12.021.
[28] ZHANG Jingbin, LI Yan, LIU Xiaochang, et al. Characterization of the microbial composition and quality of lightly salted grass carp (Ctenopharyngodon idellus) fillets with vacuum or modified atmosphere packaging[J]. International Journal of Food Microbiology, 2019, 293: 87-93. DOI:10.1016/j.ijfoodmicro.2018.12.022.
[29] 李丽, 蒋景龙. 基于高通量测序的西洋参根际土壤细菌群落分析[J]. 中药材, 2019, 42(1): 7-12. DOI:10.13863/j.issn1001-4454.2019.01.002.
[30] 李恒, 陈功, 伍亚龙, 等. 高通量测序方法研究传统四川泡菜母水中微生物群落的动态变化[J]. 食品科学, 2018, 39(24): 131-138. DOI:10.7506/spkx1002-6630-201824020.
[31] 张胜金戈, 刘小燕, 文志安, 等. 淡水小龙虾细菌性疾病的诊断与防治措施[J]. 中国水产, 2017(11): 88-90.
[32] 彭博文, 杨移斌, 艾晓辉, 等. 克氏原螯虾源维氏气单胞菌分离鉴定及药敏特性研究[J]. 海洋湖沼通报, 2018(4): 108-114. DOI:10.13984/j.cnki.cn37-1141.2018.04.014.
[33] 于红雷, 史国萃, 常晶, 等. 腐胺对冷藏凡纳滨对虾优势腐败菌生长代谢的影响研究[J]. 食品科技, 2019, 44(3):131-135. DOI:10.13684/j.cnki.spkj.2019.03.023.