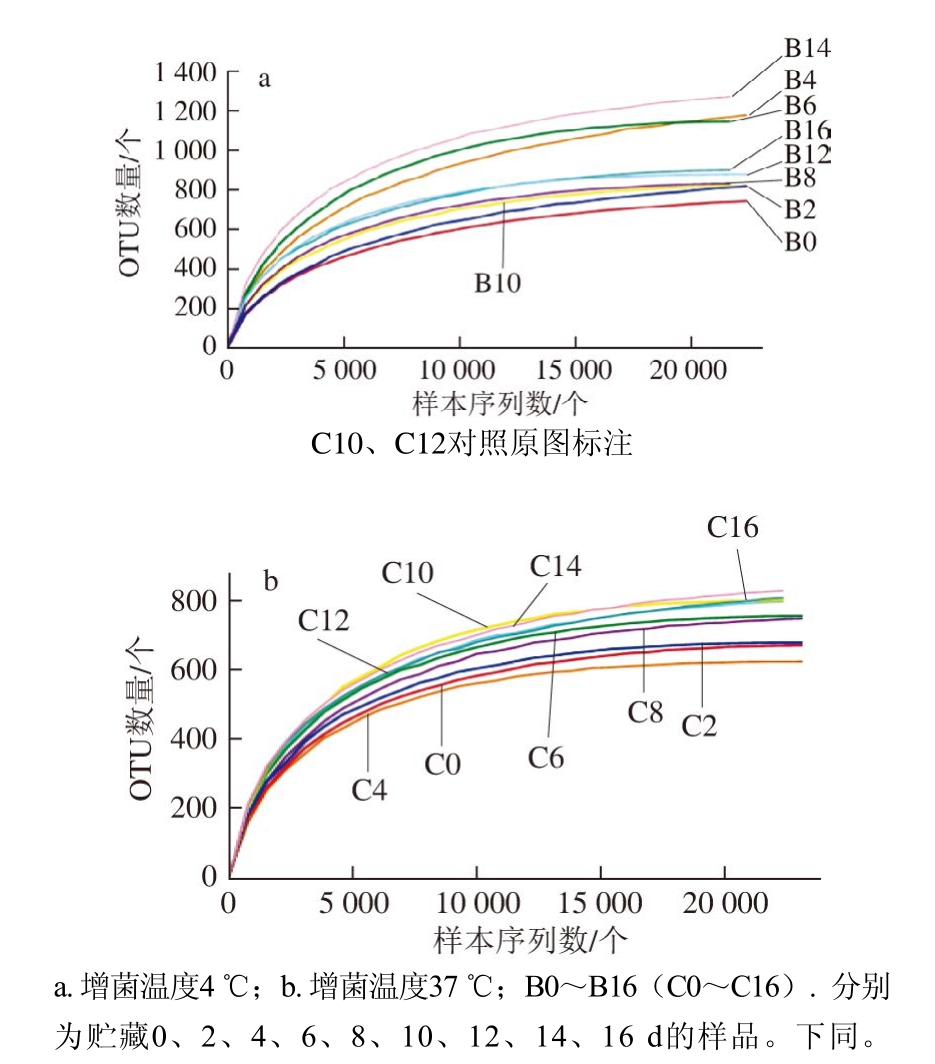
图 1 不同增菌温度下各样品的稀释性曲线
Fig. 1 Rarefaction curves for each sample under different enrichment temperatures
蔡教英1,王小玉1,姚丽锋1,游淑珠1,丁 琦1,陈海光2,*,朱楚雯2
(1.珠海检验检疫技术中心,广东 珠海 519015;2.仲恺农业工程学院轻工食品学院,广东 广州 510225)
摘 要:采用Ion Torrent PGM高通量测序技术,从分子水平上对冰鲜鸽肉贮藏过程中的细菌在4 ℃与37 ℃ 2 种增菌温度下的细菌群落结构、丰度及演替规律进行深入研究。结果表明:在整个贮藏过程中,4 ℃与37 ℃增菌处理的结果表明:在门水平上,冰鲜鸽肉中的微生物菌群均以变形菌门、厚壁菌门、拟杆菌门为优势菌门,其中变形菌门占比达78%;在科水平上,4 ℃增菌条件下,假单胞菌科、莫拉氏菌科、假诺卡氏科及肠杆菌科为优势菌科,37℃增菌条件下,气单胞菌科、动球菌科、肠杆菌科及假单胞菌科为优势菌科,占比最多的为假单胞菌科。2 种增菌温度下细菌群落的多样性及菌群结构变化反映了冰鲜鸽肉潜在的安全风险,可用于冰鲜鸽肉冷藏过程或贮藏过程中温度失控情况下的质量管理。
关键词:冰鲜鸽;微生物菌群;16S rDNA;聚合酶链式反应;测序
“一鸽胜九鸡”的俚语说明鸽子的营养成分非常丰富,比鸡更胜一筹。粤港澳等地区素来有食用乳鸽的饮食习惯,烹调方法多种多样,市场对鸽肉的需求也越来越大。但近年来,禽流感疫情严重影响了整个家禽产业的发展,传统的活禽销售模式对消费者及养殖户都潜在巨大危害。随着2014年以来广东省倡导的“集中屠宰、冷链配送、生鲜上市”的家禽屠宰政策的实施,改变禽类食品供应模式,以冰鲜替代活禽供应,对建立防控人感染禽流感的长效机制和促进产业转型升级均具有重要意义。
鸽肉营养物质丰富且水分活度高,这使得鸽肉在生产、贮藏、运输及销售等过程中易受微生物的污染,且在不同贮藏条件下,冰鲜肉的微生物多样性存在一定差异[1]。冰鲜鸽是在乳鸽屠宰后将卫生检验合格的乳鸽胴体迅速冷却至0~4 ℃,并在此温度下对动物胴体进行加工、贮运和销售[2]。冰鲜鸽肉在0~4 ℃低温条件下贮藏,能够有效抑制肉中微生物的生长和酶活性,降低细菌总数,减少有害毒素的产生,使冰鲜肉中的致病菌、生物毒素受到一定程度的抑制,延缓肉类腐败变质[3]。目前,关于冰鲜肉类的腐败微生物研究大部分集中在鸡肉[4-6]、猪肉[7]、虾[8]、牛肉[9]、羊肉、鸭肉、鱼[10-11]、贝等。张莉等[12]采用转录间隔区序列(internal transcribed spacer,ITS)测序,大部分研究采用16S rDNA测序。孙彦雨等[13]应用16S rDNA的变性梯度凝胶电泳指纹图谱技术对冰鲜鸡肉的微生物多样性进行研究。唐文静等[14]使用16S rDNA分子鉴定对冷藏海鲈鱼的优势腐败菌进行研究。岑璐伽等[15]使用聚合酶链式反应(polymerase chain reaction,PCR)-变性梯度凝胶电泳分析冷却牦牛肉贮藏过程中的优势菌。
近年来,随着测序技术的发展,可以进行大规模平行测序,且能客观反映样本中DNA的丰度和鉴定出传统培养条件下不可生长细菌的高通量测序技术应用越来越广泛,而且较传统测序法成本低廉[16]。与传统方法相比,细菌的高通量测序技术在微生物群落研究方面的优势越来越突出,目前已广泛应用于医学[17]、环境[18-20]、昆虫学[21-23]、中药[24-25]及植物[26-27]等多个方面。且近几年来在食品领域的应用发展迅速,已广泛应用于乳制品[15]、发酵类食品[16,28]、肉制品[4-7]、水产品[8,10-11]中的细菌群落多样性研究。
本研究采用高通量测序技术对冰鲜鸽肉中的腐败菌进行研究,分析不同增菌温度下冰鲜鸽肉中微生物菌群的菌相构成情况、贮藏过程中各种微生物菌群数量的变化特点以及贮藏过程中肉质新鲜度的变化规律。为了提高DNA的得率,需对细菌进行增菌处理。目前的国家强制性标准中测定菌落总数时的培养温度为(36±1) ℃,但是该条件下培养时的菌落总数测定对象只包括嗜温性需氧微生物。对于低温贮藏冷鲜肉来说,嗜冷菌也可能是优势菌群,如果能对嗜冷菌和嗜温菌进行同时检测,综合评价会更合理。因此,本研究选取4 ℃和37 ℃ 2 种增菌温度,综合分析得出冰鲜鸽肉的微生物消长规律,了解其菌相变化和品质变化的规律,以期为冰鲜鸽的保鲜控制技术提供理论基础。
冰鲜鸽肉由珠海市金同农副食品有限公司提供,随机选取18 只当天屠宰的冰鲜鸽密封包装后2 h内用冰盒运送至实验室。在无菌环境下将鸽肉随机分为9 组,每组2等份,分别收集每只冰鲜鸽的鸽肉作为样品,每份25 g,经取样袋密封包装后放置于4 ℃冰箱贮藏。
Q5413细菌基因组DNA提取试剂盒 德国Tiangen公司;10855-021 LB肉汤 美国Termo Fisher Scientific公司;M0491L扩增聚合酶 美国NEB公司;P7589荧光定量染料、75510-019琼脂糖凝胶试剂、AM9870琼脂糖凝胶电泳缓冲液 美国Invitrogen公司;DL2000核酸Marker 日本TaKaRa公司。
2100生物分析仪 美国Agilent公司;KB400恒温培养箱 德国Binder公司;Minispin小型高速离心机德国Eppendorf公司;LogicA2生物安全柜 美国Labconco公司;Synergy2全波长酶标仪 美国Biotek公司。
1.3.1 细菌总DNA提取
以购买当天为0 d,作为对照组,其余为实验组。在无菌环境下,分别取贮藏不同时间(0、2、4、6、8、10、12、14、16 d)的冰鲜鸽肉25 g装入拍打袋,加入225 mL无菌生理盐水,用拍击式均质器拍打后,移取1 mL菌液加入40 mL的LB培养基中,于4 ℃条件下增菌7 d,同时取1 mL菌液加入40 mL的LB培养基中,于37 ℃条件下增菌24 h。分别取1 mL增菌液至EP管内,采用Tangen细菌基因组DNA提取试剂盒按照相应操作步骤进行。
1.3.2 16S rDNA V3-V4区PCR扩增
引物序列:上游5’-A C T C C T A C G G G A GGCAGCA-3’,下游5’-GGACTACHVGGGTWTCTAAT-3’。对细菌16S rDNA基因V3-V4可变区进行PCR扩增。反应体系(25 μL):2×Mix 12.5 μL、引物(10 μmol/L)1 μL、DNA模板(30 ng)1 μL、ddH2O 10.5 μL。98 ℃预变性45 s;98 ℃变性15 s,57 ℃退火30 s,72 ℃延伸1 min,循环20 次;72 ℃末端延伸10 min,4 ℃条件下保温。
1.3.3 测序文库制备及数据分析
高通量测序文库的构建和Ion Torrent PGM高通量测序的工作委托上海伯豪生物公司进行。按97%相似性标准进行操作分类单元(operational taxonomic unit,OTU)聚类,然后利用RDP-classifier及Silva数据库进行物种注释和分类学分析。基于OTU的分析结果,采用对样本序列进行随机抽样的方法,计算常用的Alpha多样性指数,包括超Ⅰ指数、Ace指数、香农指数(Shannon index)和辛普森指数(Simpson index),并作出相对应的稀释曲线。超Ⅰ指数和Ace指数用于评价微生物菌群丰富度,其数值越高,表明群落物种丰富度越高;香农指数和辛普森指数反映物种多样性,香农指数与群落多样性呈正相关,辛普森指数则相反,其值越大,群落多样性越低。通过多变量统计学方法的主成分分析(principal component analysis,PCA),可以直观显示不同样品群落间物种组成的相似性及差异性[14]。
为避免在高通量测序过程中由于引入错误或不可靠碱基等不可控因素造成的测序错误,减少对下游生物信息分析造成的不良影响,需要对测序得到的原始数据进行优化处理。通过利用QIIME软件对测序所得原始序列数进行过滤,去除低质量序列片段、错误序列、连接后引物序列以及无法与数据库中序列信息比对一致的序列后,得到最终的有效序列。本次测序所得到的有效序列均可以达到后续微生物多样性分析的要求。稀释性曲线是通过对序列进行随机抽样,以抽到的序列数与它们所代表的物种数来构建的曲线。根据曲线走势可以比较测序样本的物种丰富度,同时说明测序数据的合理性以及测序量能否涉及所有菌群。
18 个样品经过测序后得到的下机序列总数为589 542 个,其中有效序列总数达420 154 个,每个样品的有效序列百分比均达60%以上。说明本次测序所得到的有效序列可以达到后续微生物多样性分析的要求。

图 1 不同增菌温度下各样品的稀释性曲线
Fig. 1 Rarefaction curves for each sample under different enrichment temperatures
利用R语言工具绘制曲线图,由图1可知,样品在4 ℃增菌温度下得到的OTU数量普遍比37 ℃多,说明在4 ℃增菌温度下微生物的多样性比37 ℃时更丰富。随着测序量的不断增加,每个样品的稀释性曲线都有趋向平缓的趋势,说明本研究的检测取样合理,测序量基本反映出样品中绝大部分的物种信息,本次实验数据具有很大的实际价值。
根据97%相似性水平下的OTU信息,采用Alpha多样性指数对样品微生物物种的丰富度和多样性进行评估。
表 1 样品的Alpha多样性指数
Table 1 Alpha diversity indexs for samples
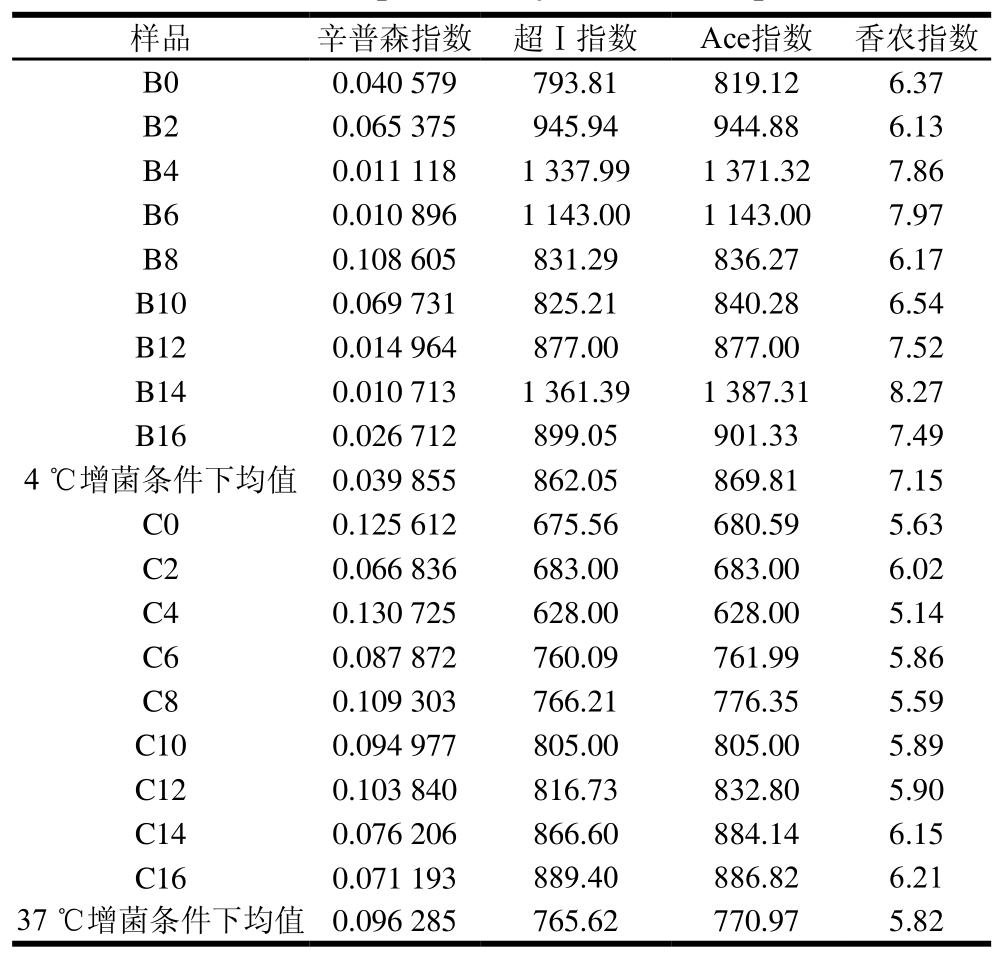
由表1可知,样品的辛普森指数、超Ⅰ指数、Ace指数及香农指数在4 ℃增菌温度下的均值分别为0.039 855、862.05、869.81及7.15,在37 ℃增菌温度下的均值分别为0.096 285、765.62、770.97及5.82,除辛普森指数外,4 ℃增菌温度下的数值普遍高于37 ℃,表明在4 ℃增菌温度下获得的微生物群落多样性与丰富度比37 ℃高。其中超Ⅰ指数和Ace指数最大的样品为B14,其次为B4,最小的为样品C4,说明4 ℃增菌温度下贮藏14 d的冰鲜鸽肉物种丰富度达到最高。这可能是由于冰鲜鸽肉此时已腐败,腐败菌含量迅速增加,占主要优势;而贮藏4 d时的物种丰富度也比较高,可能是由于此时冰鲜鸽肉中营养成分含量较高,细菌迅速生长繁殖,甚至达到细菌的对数生长期。香农指数最大的样品为B14,最小的为C4。辛普森指数最小的样品为B14,最大的为C4。上述结果表明,微生物群落多样性最高的为4 ℃条件下贮藏14 d的冰鲜鸽肉样品,最低的为37 ℃条件下藏贮4 d的冰鲜鸽肉样品。
采用RDP-classifier对各样品中的物种数目进行物种注释可以鉴定出冰鲜鸽肉贮藏过程中的细菌分属于10 门、19 纲、28 目、39 科、53 属。本研究就门、科分类学水平做具体分析。
2.3.1 基于门分类水平上的分析
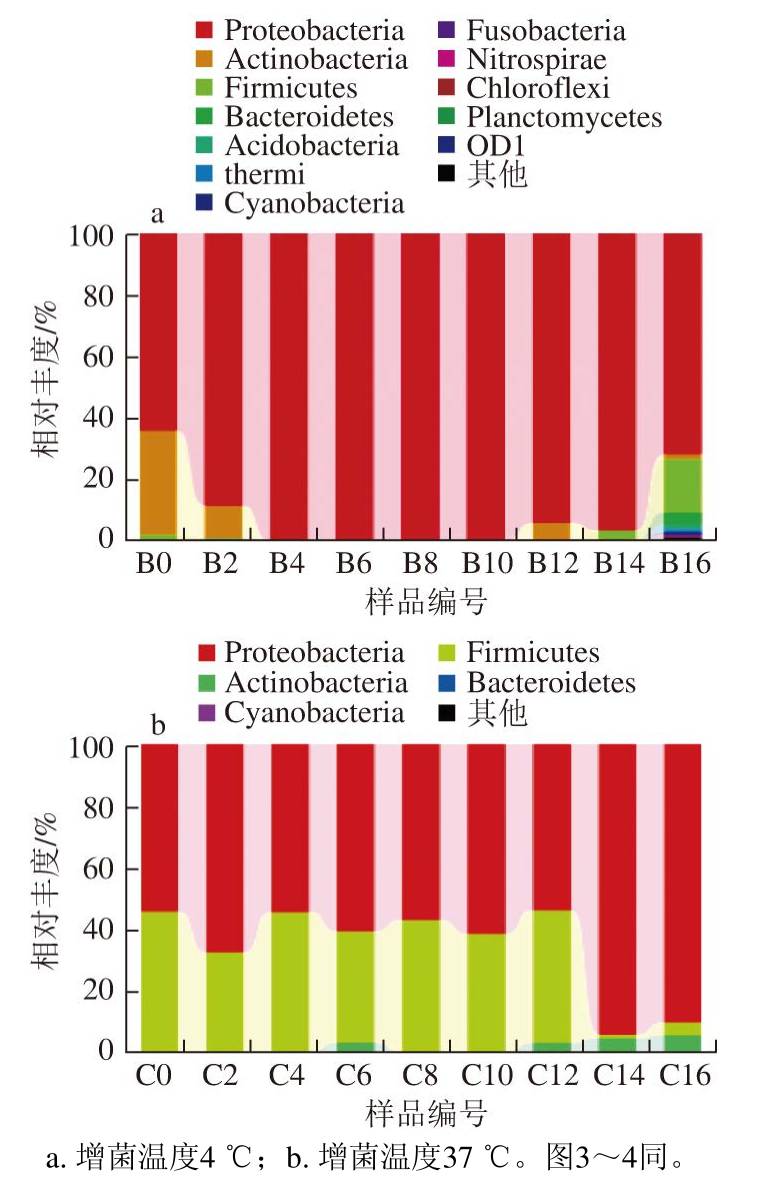
图 2 基于门分类水平上的样品菌群分布图
Fig. 2 Bacterial community distribution at the phylum level
由图2可知,不同贮藏时间的冰鲜鸽肉微生物菌群在4 ℃和37 ℃增菌温度下的菌相主要由变形菌门(Proteobacteria)、厚壁菌门(Firmicutes)和放线菌门(Actinobacteria)三大菌门组成。根据样品在门水平上的菌群组成结果可以看出,4 ℃增菌温度下变形菌门的总丰度为90.6%,放线菌门为5.8%,厚壁菌门为2.6%,37 ℃增菌温度下变形菌门的丰度为66.1%,厚壁菌门为32.1%,放线菌门为1.8%。这说明不同贮藏时间的冰鲜鸽肉在不同增菌温度下的微生物菌群组成均以变形菌门为优势菌门,且4 ℃增菌温度下尤为明显。4 ℃增菌温度下变形菌门的生长趋势为由低到高,贮藏10 d时达到峰值,并在16 d时降至71.9%;放线菌门总体呈下降趋势。而在37 ℃增菌温度下,变形菌门的生长趋势为由低到高,在14 d时达到峰值,厚壁菌门与变形菌门的生长呈相反的趋势。除了这些主要菌门外,还存在一些丰度较低的拟杆菌门(Bacteroidetes)、酸杆菌门(Acidobacteria)、蓝藻细菌门(Cyanobacteria)、梭杆菌门(Fusobacteria)、硝化螺旋菌门(Nitrospirae)、绿弯菌门(Chloroflexi)、浮霉菌门(Planctomycetes)以及其他未能分类的菌门,有的所占百分比不足0.01%,图中显示不明显。这也说明冰鲜鸽肉在贮藏过程中的微生物多样性丰富、丰度比较集中的特点。
2.3.2 基于科分类水平上的分析
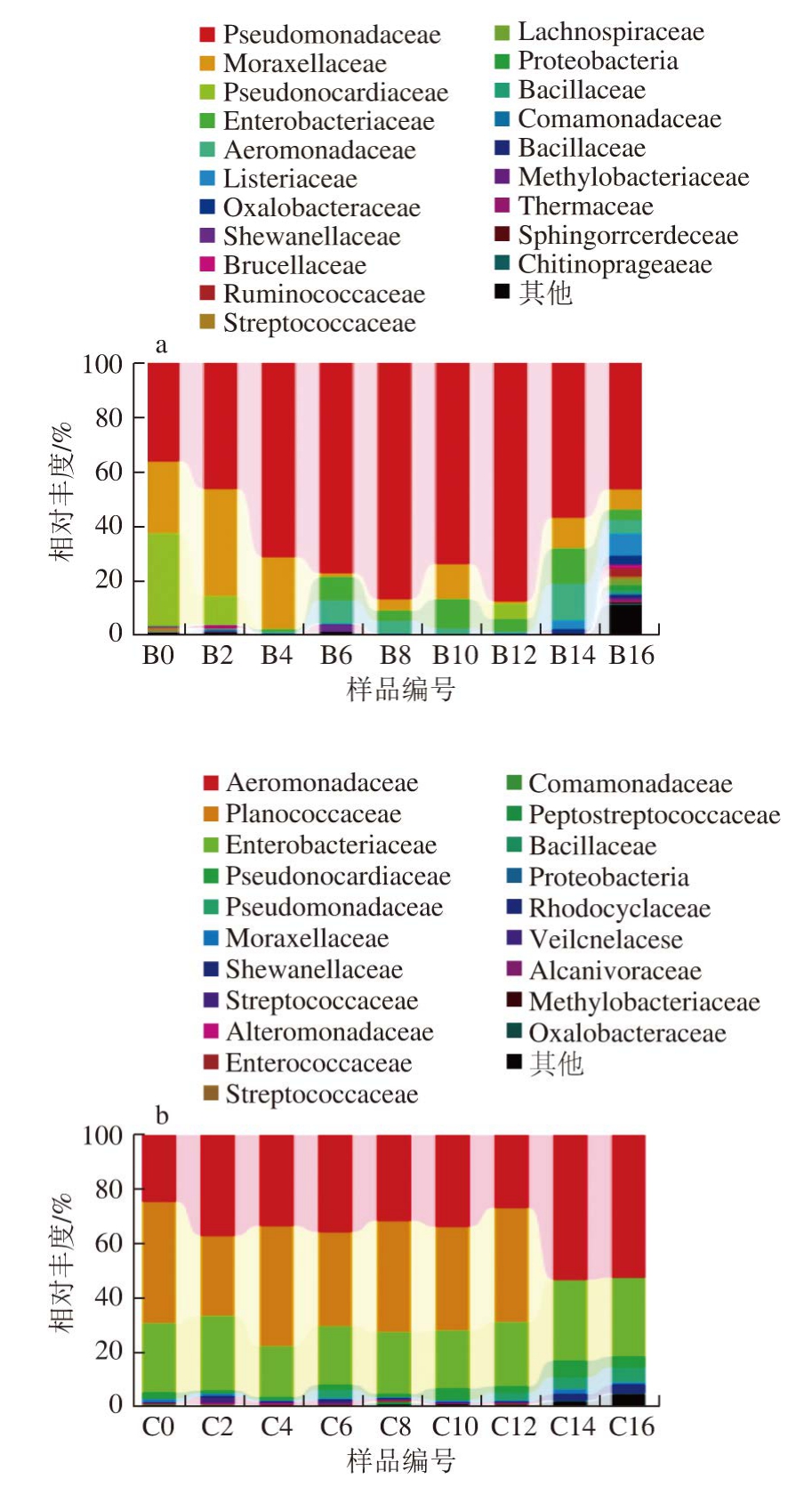
图 3 基于科分类水平上的样品菌群分布图
Fig. 3 Bacterial community distribution at the family level
由图3可知,不同贮藏时间的冰鲜鸽肉在4 ℃和37 ℃增菌温度下总共分离得到39 个不同的科。在门水平上占主要优势的变形菌门(Proteobacteria)在科的分类水平上主要包括假单胞菌科(Pseudomonadaceae)、莫拉氏菌科(Moraxellaceae)、肠杆菌科(Enterobacteriaceae)、气单胞菌科(Aeromonadaceae)、草酸杆菌科(Oxalobacteraceae)、希万氏菌科(Shewanellaceae)、丛毛单胞菌科(Comamonadaceae)、甲基杆菌科(Methylobacteriaceae)、交替单胞菌科(Alteromonadaceae)、布鲁氏菌科(Brucellaceae)、红环菌科(Rhodocyclaceae)、食碱菌科(Alcanivoraceae)、柄杆菌科(Caulobacteraceae)及慢生根瘤菌科(Bradyrhizobiaceae)等。厚壁菌门(Firmicutes)在科水平上鉴定到的主要包括李斯特氏菌科(Listeriaceae)、疣微菌科(Ruminococcaceae)、链球菌科(Streptococcaceae)、毛螺菌科(Lachnospiraceae)、芽孢杆菌科(Bacillaceae)、消化链球菌科(Peptostreptococcaceae)、动球菌科(Planococcaceae)、肠球菌科(Enterococcaceae)及葡萄乳球菌科(Staphylococcaceae)等。放线菌门(Actinobacteria)在科水平上鉴定到的主要有假诺卡氏科(Pseudonocardiaceae)、微球菌科(Micrococcaceae)、微杆菌科(Microbacteriaceae)、棒状杆菌科(Corynebacteriaceae)、红椿菌科(Coriobacteriaceae)、段杆菌科(Brevibacteriaceae)及双歧杆菌科(Brevibacteriaceae)等。
在4 ℃和37 ℃增菌温度下,微生物菌群种类和丰度大小存在较大的差异性。在4 ℃增菌条件下,冰鲜鸽肉中的微生物菌群主要以假单胞菌科(Pseudomonadaceae)、莫拉氏菌科(Moraxellaceae)、假诺卡氏科(Pseudonocardiaceae)、肠杆菌科(Enterobacteriaceae)和气单胞菌科(Aeromonadaceae)为主,其中假单胞菌科占比达64.6%,在4 ℃贮藏过程中呈先增长后下降的趋势,贮藏16 d时达到峰值87.3%。假单胞菌科是冰鲜鸽肉贮藏过程中的主要优势菌群,与目前大部分研究结果一致[15-16,29]。其次是莫拉氏菌科和假诺卡氏科,分别占14.5%和5.6%。在37 ℃增菌条件下,冰鲜鸽肉中的微生物菌群主要以气单胞菌科(Aeromonadaceae)、动球菌科(Planococcaceae)、肠杆菌科(Enterobacteriaceae)、假单胞菌科(Pseudomonadaceae)和假诺卡氏科(Pseudonocardiaceae)为主,其中气单胞菌科、动球菌科和肠杆菌科占比分别为36.7%、30.5%和24.4%。还有一部分菌群未能鉴定到具体菌科,有待进一步研究。
莫拉氏菌科(Moraxellaceae)和假单胞菌科(Pseudomonadaceae)在4 ℃增菌条件下的菌群丰度明显高于37 ℃。莫拉氏菌科和假单胞菌科是典型嗜冷菌,是冰鲜鸽肉冷藏贮藏期间的优势腐败菌,这与原林等[30]的研究结果一致,说明莫拉氏菌科和假单胞菌科是冷冻肉制品、冷冻水产品等的优势腐败微生物。气单胞菌科、动球菌科和肠杆菌科在37 ℃增菌条件下的菌群丰度明显高于4 ℃,且37 ℃增菌条件下气单胞菌科、假单胞菌科和假诺卡氏科所占的比例总体上呈增加趋势,在贮藏16 d时达到最高,说明这几类菌科是影响冰鲜鸽肉腐败变质的优势菌群。
综合以上分析发现,低温贮藏冰鲜鸽肉的主要菌群不仅包括嗜冷菌,还包括一些嗜温菌。虽然样本的贮藏条件为0~4 ℃,但是样本中仍然存在一些嗜温菌,生长温度在30~37 ℃,如动杆菌科、肠杆菌科、气单胞菌科等。因此冰鲜鸽肉在贮藏过程中一定要控制好温度,温度波动范围太大会直接导致嗜温菌等腐败菌大量繁殖,促使冰鲜鸽肉腐败。结合4 ℃低温和37 ℃中温2 种增菌温度分析,可以较全面地反映冰鲜鸽肉在冷藏贮藏过程中污染的细菌群落结构的演替规律。
依照不同贮藏时间将冰鲜鸽肉样品分为9 组,每组由同一贮藏时间的4 ℃和37 ℃ 2 种增菌温度的样品组成。PCA图中,横轴表示第1主成分(PC1),纵轴表示第2主成分(PC2),图中样品之间位置越接近,表示样品群落结构组成越相似。
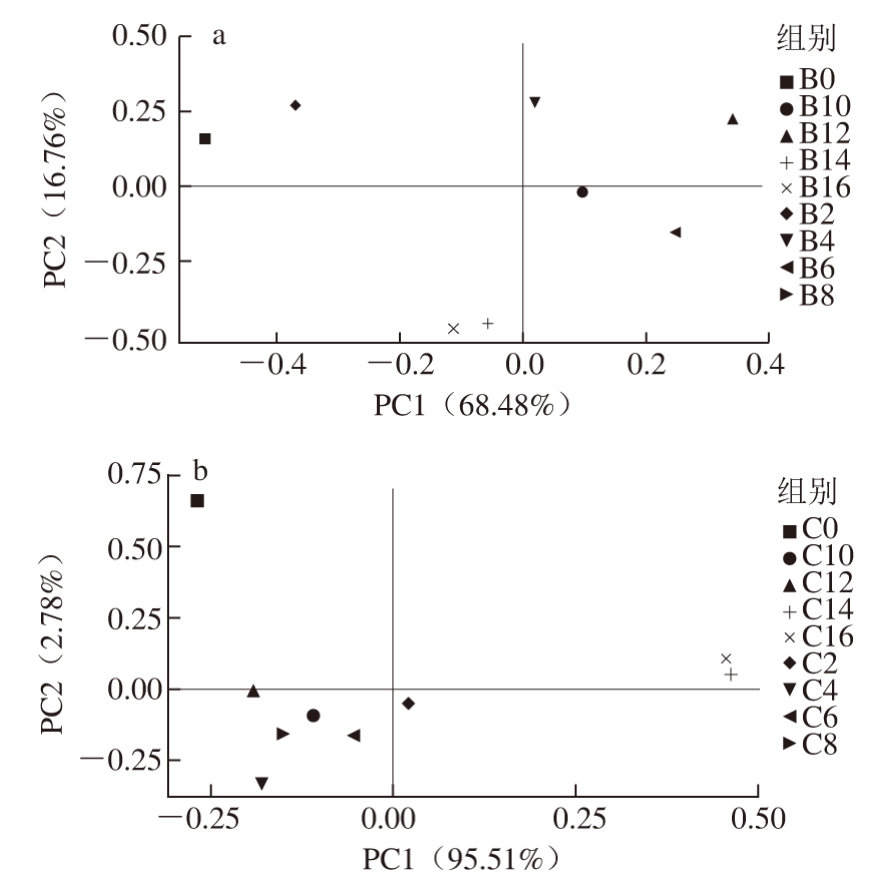
图 4 样品细菌群落的PCA
Fig. 4 PCA of bacterial communities
由图4可知,在4 ℃增菌条件下,PC1与PC2的贡献率分别为68.48%和16.76%,在37 ℃增菌条件下,PC1与PC2的贡献率分别为95.51%和2.78%,说明2 个主成分是解释样品微生物群落结构组成差异的主要因子。在4 ℃与37 ℃贮藏条件下,不同贮藏时间的各个样品能明显地分离,表明各个样品间的微生物群落结构组成具有一定差异。在PC1方向上,4 ℃和37 ℃增菌条件下的样品能够显著地分布在2 个区域,说明增菌温度在PC1水平上对样品菌群结构变化的影响显著;在PC2方向上,不同贮藏时间的样品区分明显,说明贮藏时间在PC2水平上对样品菌群结构变化的影响显著。同一增菌温度下,贮藏前期(0~4 d)的样品与贮藏中期(6~10 d)、贮藏后期(10~16 d)的样品之间分布距离较大,这主要是由于冰鲜鸽肉在贮藏前期仍处于鲜肉与次鲜肉状态,而在贮藏中期开始加速腐败,直到贮藏后期完全腐败,进而导致冰鲜鸽肉群落结构组成发生较大变化。然而,2 种增菌温度下,处于贮藏前期、中期、后期的各个样品间距离较大,说明同一时期的样品群落结构组成发生变化。
对冰鲜鸽肉在4 ℃和37 ℃ 2 种增菌温度下的细菌群落多样性进行研究,结果表明:在门水平上,4 ℃增菌温度下变形菌门的丰度为90.6%,放线菌门为5.8%,厚壁菌门为2.6%;37 ℃增菌温度下变形菌门的丰度为66.1%,厚壁菌门为32.1%,放线菌门为1.8%。其中,变形菌门从贮藏初期到贮藏后期的生长趋势由低至高。在科水平上,在4 ℃增菌条件下,冰鲜鸽肉中的微生物菌群主要以假单胞菌科、莫拉氏菌科、假诺卡氏科、肠杆菌科和气单胞菌科为主,其中假单胞菌科占比达64.6%,是冰鲜鸽肉贮藏过程中的主要优势菌群,生长趋势也是随着时间的延长而增长。在37 ℃增菌条件下,冰鲜鸽肉中的微生物菌群主要以气单胞菌科、动球菌科、肠杆菌科、假单胞菌科和假诺卡氏科为主,其中气单胞菌科、动球菌科和肠杆菌科的占比分别为36.7%、30.5%和24.4%。贮藏前期,假单胞菌科、莫拉氏菌科为主要优势菌科,但到后期莫拉氏菌科占比逐渐减少,而假诺卡氏科的占比逐渐增加。以上研究结果能够帮助对冰鲜鸽肉低温贮藏中的微生物菌群变化有较为全面的认识,要对冰鲜鸽肉进行全面质量管理的深入研究,基于门的水平,可以以变形菌门、厚壁菌门、拟杆菌门等优势菌门为重点,基于科的水平,可以以假单胞菌科、莫拉氏菌科、假诺卡氏科、肠杆菌科和气单胞菌科为重点。本研究结果对冰鲜鸽肉贮藏过程中微生物生长的抑制和延长冰鲜鸽肉货架期方面的深入研究具有意义。
参考文献:
[1] 王森, 王雪梅, 周永昌, 等. 安全、高品质的冷鲜肉[J]. 肉类工业,2011(5): 50-51. DOI:10.3969/j.issn.1008-5467.2011.05.021.
[2] 梁雅妍, 陈益填. 中国鸽肉产品出口面临的挑战与对策分析[J]. 养禽与禽病防治, 2017(4): 39-42.
[3] 尹德凤, 张莉, 张大文, 等. 生鲜肉类产品中腐败细菌研究[J]. 农产品质量与安全, 2018(3): 21-29.
[4] 吴海虹, 刘朏, 刘芳, 等. 低温贮藏对冷鲜鸡腐败菌菌群变化的影响[J]. 现代食品科技, 2016, 32(4): 177-181. DOI:10.13982/j.mfst.1673-9078.2016.4.029.
[5] 吴文锦, 汪兰, 李新, 等. 冰鲜鸡肉保鲜技术的研究[J]. 食品工业,2015, 36(8): 91-95.
[6] 高磊, 谢晶, 叶藻, 等. 冷鲜鸡腿肉中优势腐败菌的分离鉴定及腐败能力研究[J]. 食品与发酵工业, 2015, 41(8): 48-53. DOI:10.13995/j.cnki.11-1802/ts.201508009.
[7] 黄永震, 王周利, 姜艳芬, 等. 高通量转录组测序技术在猪肉品质研究中的应用进展[J]. 中国畜牧杂志, 2018, 54(2): 8-14.
[8] 朱亚珠, 翁佩芳, 刘娜, 等. 中国毛虾贮藏过程细菌群落多样性分析[J].核农学报, 2017, 31(9): 1759-1766.
[9] 范晓攀, 王娉, 陈颖, 等. 肉类调理食品中细菌多样性的分析[J]. 现代食品科技, 2017, 33(1): 237-242.
[10] 杨超, 车有, 宋存江. 生鲜家畜肉类与水产鱼虾类物流过程中微生物菌相变化的研究进展[J]. 食品科学, 2015, 36(23): 307-313.
[11] 蓝蔚青, 张皖君, 谢晶, 等. 气调包装对鲳鱼冷藏期间主要微生物多样性的影响[J]. 中国食品学报, 2017, 17(2): 152-159. DOI:10.16429/j.1009-7848.2017.02.021.
[12] 张莉, 林佳宁, 张远, 等. ITS高通量测序研究黄海微型真核浮游生物多样性及分布特征[J]. 环境科学, 2018, 39(5): 2368-2379.DOI:10.13227/j.hjkx.201710055.
[13] 孙彦雨, 周光宏, 徐幸莲. 冰鲜鸡肉贮藏过程中微生物菌相变化分析[J]. 食品科学, 2011, 32(11): 146-151.
[14] 唐文静, 王楚文, 柳云龙, 等. 冷藏海鲈鱼优势腐败菌的筛选和鉴定[J].食品科学, 2016, 37(3): 170-174. DOI:10.7506/spkx1002-6630-201603031.
[15] 岑璐伽, 唐善虎, 郝小倩, 等. 冷却牦牛肉贮藏过程中优势菌的PCR-变性梯度凝胶电泳分析[J]. 肉类研究, 2012, 26(1): 36-40.
[16] 闫雪, 姚卫蓉, 钱和. 国内外食品微生物快速检测技术应用进展[J].食品科学, 2005, 26(6): 269-272.
[17] XU Sai, LU Wenjing, LIU Yanting, et al. Structure and diversity of bacterial communities in two large sanitary landfills in China as revealed by high-throughput sequencing (MiSeq)[J]. Waste Management, 2017, 63: 41-48. DOI:10.1016/j.wasman.2016.07.047.
[18] 张艺洁, 邵惠芳, 张珂, 等. 基于高通量测序研究施肥对连作植烟土壤环境及微生物的影响[J]. 中国农业科技导报, 2018, 20(5): 16-25.DOI:10.13304/j.nykjdb.2017.0348.
[19] 杨岳, 魏杰. 高通量测序分析蒙古白丽蘑蘑菇圈子实体下土壤真菌多样性[J]. 分子植物育种, 2018(10): 3412-3422. DOI:10.13271/j.mpb.016.003412.
[20] 苗菁, 宗万松, 吕敏, 等. 基于高通量测序技术研究土壤细菌对盐生植被演替的响应[J]. 土壤通报, 2018(3): 602-608. DOI:10.19336/j.cnki.trtb.2018.03.15.
[21] 郑天瑶, 宿树兰, 蔡红蝶, 等. 基于16S rDNA测序技术分析不同龄期蚕沙中菌群多样性[J]. 中国中药杂志, 2017, 42(16): 3098-3105.DOI:10.19540/j.cnki.cjcmm.20170714.005.
[22] XIANG Yang, WU Xiaoqin, ZHOU Aidong. Bacterial diversity and community structure in the pine wood nematode Bursaphelenchus xylophilus and B. mucronatus with different virulence by highthroughput sequencing of the 16S rDNA[J]. PLoS One, 2015, 10(9):e0137386. DOI:10.1371/journal.pone.0137386.
[23] 杨晓晴, 王正亮, 王天召, 等. 基于16S rRNA高通量测序的灰飞虱体内细菌群落结构及多样性分析[J]. 昆虫学报, 2018, 61(2): 200-208.DOI:10.16380/j.kcxb.2018.02.006.
[24] 刘蓬蓬, 陈江宁, 孟莉, 等. 基于Illumina MiSeq高通量测序分析黄芪内生细菌多样性[J]. 中草药, 2018(11): 2640-2645.
[25] 陈艳红, 邢晓科, 郭顺星. 北京地区手参内生真菌的区系组成分析[J]. 菌物学报, 2018, 37(1): 35-42. DOI:10.13346/j.mycosystema.170214.
[26] 陈泽斌, 高熹, 胡昳, 等. Illumina MiSeq高通量测序分析花生仁内生细菌多样性[J]. 昆明学院学报, 2018(3): 86-89. DOI:10.14091/j.cnki.kmxyxb.2018.03.017.
[27] 王春蕾, 方志军, 许艳蕊, 等. 基于高通量测序技术分析使它隆对玉米根系内生菌多样性的影响[J]. 作物杂志, 2018(1): 160-165.DOI:10.16035/j.issn.1001-7283.2018.01.026.
[28] 关统伟, 向慧平, 王鹏昊, 等. 基于高通量测序的郫县豆瓣不同发酵期细菌群落结构及其动态演替[J]. 食品科学, 2018, 39(4): 106-111.DOI:10.7506/spkx1002-6630-201804016.
[29] 张赫宇, 杨波, 罗瑞明, 等. 高通量测序分析冷鲜滩羊肉储藏过程中的细菌群落多样性[J]. 食品工业科技, 2016, 37(13): 177-182.DOI:10.13386/j.issn1002-0306.2016.13.027.
[30] 原林, 丁长河. 冷藏食品中的常见微生物及其防控[J]. 粮食与食品工业, 2016, 23(6): 29-33.
Diversity of Microbial Flora of Chilled Pigeon Meat during Storage
CAI Jiaoying1, WANG Xiaoyu1, YAO Lifeng1, YOU Shuzhu1, DING Qi1, CHEN Haiguang2,*, ZHU Chuwen2
(1.Technical Center of Zhuhai Entry-Exit Inspection and Quarantine Bureau, Zhuhai 519015, China;2.College of Light Industry and Food Technology, Zhongkai University of Agriculture and Engineering, Guangzhou 510225, China)
Abstract:The microbial community diversity, abundance and succession in chilled pigeon during storage were studied at the molecular level through high-throughput sequencing using the Ion Torrent Personal Genome Machine (PGM). Bacterial enrichment culture was conducted at 4 or 37 ℃. The results showed that Proteobacteria (78%), Firmicutes and Bacteroidetes were identif i ed as the dominant bacteria at the phylum level duing the whole storage period for both culture temperatures. At the family level, Pseudomonadaceae, Moraxellaceae, Pseudonocardiaceae and Enterobacteriaceae were the dominant bacteria for 4 ℃ enrichment temperature, while Aeromonadaceae, Planococcaceae, Enterobacteriaceae and Pseudomonadaceae were dominant when they were enriched at 37 ℃. Pseudomonadaceae was the most dominant family. The changes in bacterial community diversity and structure could ref l ect the potential safety risk of chilled pigeon and therefore provide a theoretical basis for the effective implementation of chilled pigeon quality and safety monitoring when temperature is out of control during storage.
Keywords:chilled pigeon; microf l ora; 16S rDNA; polymerase chain reaction; sequencing
Meat Research, 2018, 32(9): 41-46. DOI:10.7506/rlyj1001-8123-201809008. http://www.rlyj.pub
CAI Jiaoying, WANG Xiaoyu, YAO Lifeng, et al. Diversity of microbial fl ora of chilled pigeon meat during storage[J].
引文格式:蔡教英, 王小玉, 姚丽锋, 等. 冰鲜鸽肉贮藏过程中的微生物菌群多样性[J]. 肉类研究, 2018, 32(9): 41-46. DOI:10.7506/rlyj1001-8123-201809008. http://www.rlyj.pub
文章编号:1001-8123(2018)09-0041-06
文献标志码:A
中图分类号:TS251.1
DOI:10.7506/rlyj1001-8123-201809008
*通信作者简介:陈海光(1965—),男,教授,硕士,研究方向为农产品加工及贮藏。E-mail:chg1508@163.com
第一作者简介:蔡教英(1987—),女,工程师,硕士,研究方向为微生物学与分子生物学。E-mail:492487491@qq.com
基金项目:广州市科技计划项目(201604020050);国家质量监督检验检疫总局科技计划项目(2017IK134);海关总署科研计划项目(2018IK046)
收稿日期:2018-05-14