
图1 腊肉样品中细菌的DGGE分析结果
Fig.1 DGGE fingerprint of bacteria in Chinese bacon samples
董 蕴1,王玉荣1,王 尧1,廖 华2,赵慧君1,郭 壮1,3,*
(1.湖北文理学院食品科学技术学院,鄂西北传统发酵食品研究所,湖北 襄阳 441053;2.恩施市农业局,湖北 恩施 445000;3.恩施市公共检验检测中心,湖北 恩施 445000)
摘 要:以采集自湖北省恩施地区的5 个腊肉样品为研究对象,采用聚合酶链式反应-变性梯度凝胶电泳(polymerase chain reaction-denatured gradient gel electrophoresis,PCR-DGGE)与MiSeq高通量测序技术相结合的方法对其中所含微生物的多样性进行解析。PCR-DGGE结果表明,腊肉样品中的细菌主要为葡萄球菌属(Staphylococcus)、嗜冷杆菌属(Psychrobacter)、乳酸杆菌属(Lactobacillus)和假交替单胞菌属(Pseudoalteromonas);MiSeq高通量测序结果表明,腊肉中的细菌门主要为硬壁菌门(Firmicutes)和变形菌门(Proteobacteria),其平均相对含量分别为54.05%和44.28%,细菌属主要为葡萄球菌属(Staphylococcus)、嗜冷杆菌属(Psychrobacter)、假交替单胞菌属(Pseudoalteromonas)、环丝菌属(Brochothrix)、科贝特氏菌属(Cobetia)和不动细菌属(Acinetobacter),其平均相对含量分别为40.18%、24.02%、9.37%、8.53%、4.71%和2.31%;在分类操作单元(operational taxonomic units,OTU)水平上,发现252 个核心OTU,累计平均相对含量高达74.22%。由此可见,PCR-DGGE和MiSeq高通量测序的结果综合分析得出恩施地区腊肉中的细菌主要为葡萄球菌属(Staphylococcus)、嗜冷杆菌属(Psychrobacter)和假交替单胞菌属(Pseudoalteromonas)。
关键词:腊肉;细菌;变性梯度凝胶电泳;MiSeq高通量测序
腊肉主要是以鲜猪肉或冷冻猪肉为原料,添加食盐等腌制后经烘烤、晾晒或熏制而成的传统非即食性发酵肉制品,我国湖北、湖南、四川、云南、广东和黑龙江等多地区一直以来均有制作和食用腊肉的习惯[1-2]。在腊肉的整个发酵与贮藏过程中,微生物与腊肉中的蛋白质和脂肪等营养物质的反应不仅赋予腊肉色泽鲜明、风味独特的品质,同时也有可能使成品受到杂菌污染[3-4]。因此,采用合适的技术对腊肉制品中的微生物种类及多样性进行解析是极为必要的。
刘晓蓉等[5]采用纯培养手段从自然发酵腊肉中分离出28 株菌株,经鉴定其中有4 株隶属于乳酸杆菌属;刘书亮等[6]采用分离纯化和生理生化鉴定相结合的方法从12 份传统腌腊肉制品中分离出戊糖乳杆菌(Lactobacillus pentosus)、食品乳杆菌(Lactobacillus alimentarius)、干酪乳杆菌(Lactobacillus casei)、弯曲乳杆菌(Lactobacillus curvatus)和清酒乳杆菌(Lactobacillus sake)等菌株;陈竞适等[7]以湘西腊肉为研究对象,采用稀释平板计数法对其微生物结构进行解析,发现酵母菌、霉菌和微球菌是其主要微生物群落;冯秀娟等[8]以湖南传统腊肉为原料,采用传统微生物学方法对发酵过程中微生物的生长情况进行研究,发现乳酸菌和葡萄球菌为其中的优势菌;王海燕等[9]采用倾注平板和平板划线法从传统湖南腊肉中分离出289 株葡萄球菌;李福荣等[10]采用纯培养手段对信阳传统发酵腊肉中的细菌进行研究,发现其优势细菌为葡萄球菌和微球菌。以上研究均采用以纯培养技术为基础的传统微生物学手段对腊肉中的微生物进行分析,发掘了大量的微生物菌种资源,但该方法具有一定的局限性,不能鉴定出不可培养的微生物,不能全面、准确地反映样品的微生物信息[11]。变性梯度凝胶电泳(denatured gradient gel electrophoresis,DGGE)技术能够应用于食品中微生物群落结构的研究,具有操作简单易行、灵敏度高、可检测到1 个核苷酸水平的差异等特点[12],目前在猪肉[13]、鸭肉[14-15]和鸡肉[16]冷藏过程中的微生物多样性变化及发酵肉制品[17]和屠宰场废水[18]的微生物多样性解析中有着广泛的应用。较之DGGE技术,MiSeq高通量测序技术具有通量高、能够实现样品间平行分析的优点[19],目前在肉制品发酵用菌株基因组测序[20-21]、鸡肉制品[22]和火腿肠[23]腐败微生物鉴定及食用肉制品对肠道菌群多样性的影响方面[24]有着广泛的应用。DGGE和MiSeq高通量测序技术相结合,亦应用于牛肉等肉制品的细菌多样性研究中[25]。目前,关于恩施地区腊肉细菌多样性评价的研究还较少,将DGGE和MiSeq高通量测序技术相结合,用于腊肉制品微生物多样性解析的研究也较少。
本研究采用免培养、结果直观可靠的聚合酶链式反应(polymerase chain reaction,PCR)-DGGE技术与MiSeq高通量测序技术相结合的方法对采集自恩施地区农家自制腊肉的细菌多样性进行解析,以期为该地区腊肉品质的保障及微生物菌种资源发掘提供一定的参考。
腊肉:湖北省恩施土家苗族自治州农家自制,5 份样品分别编号为LR1、LR2、LR3、LR4和LR5。
聚丙烯酰胺、尿素、N,N-亚甲基二丙烯酰胺、过硫酸铵、冰醋酸、甲醛和硝酸银(均为分析纯) 国药集团化学试剂有限公司;QIAGEN DNeasy mericon Food Kit提取试剂盒 德国Qiagen公司;5×FastPfu Buffer、10×PCR Buffer、dNTPs Mix、DNA聚合酶(5 U/μL)、pMD18-T载体 宝生物工程(大连)有限公司;6×Loading buffer、DL2000、DL15000 DNA Marker 宝日医生物技术(北京)有限公司;2×PCR mix 南京诺唯赞生物科技有限公司;DGGE扩增引物ALL-GC-V3F(5’-CGCC CGGGGCGCGCCCCGGGCGGCCCGGGGGCACC GGGGGCCTACGGGAGGCAGCAG-3’)/ALL-V3R(5’-ATTACCGCGGCTGCTGG-3’)和高通量测序引物338F(5’-ACTCCTACGGGAGGCAGCA-3’)/806R(5’-GGACTACHVGGGT-3’) 武汉天一辉远生物科技有限公司;Axygen清洁试剂盒 北京科博汇智生物科技发展有限公司;大肠杆菌(E. coli)Top10 本实验室保藏。
HBM-400B拍击式无菌均质器 天津市恒奥科技发展有限公司;5810R台式高速冷冻离心机 德国Eppendorf公司;ND-2000C微量紫外分光光度计美国Nano Drop公司;DYY-12电泳仪 北京六一仪器厂;VeritiTM96孔梯度PCR扩增仪 美国AB公司;PowerPacTMBasic稳压仪、DCodeTMSystem、UVPCDS 8000凝胶成像分析系统 美国Bio-Rad公司;MiSeq PE300高通量测序平台 美国Illumina公司。
1.3.1 样品前处理及宏基因组DNA提取
参照GB/T 4789.17—2003《食品卫生微生物检验 肉与肉制品检验》进行取样,加入灭菌水225 mL混匀后300 r/min条件下离心10 min,取上清液;然后将上清液在10 000 r/min条件下离心10 min,保留沉淀[26]。按照QIAGEN DNeasy mericon Food Kit试剂盒使用说明中的方法提取样品总DNA,并用微量紫外分光光度计检测其浓度和纯度。
1.3.2 细菌PCR-DGGE指纹图谱分析
以提取的DNA为模板进行PCR扩增,扩增体系及反应参数参照文献[27]中的方法并略作改动:10×PCR Buffer 5 μL、dNTP mix(2.5 mmol/L)4 μL、ALL-GCV3F(10 μmol/L)和ALL-V3R(10 μmol/L)各1 μL、rTaq酶(5 U/μL)0.4 μL、DNA模板1 μL,用无菌水将体系补齐至50 μL,混合均匀后置于PCR仪中94 ℃预变性5 min、94 ℃变性30 s、55 ℃退火1 min、72 ℃延伸90 s,循环30 次;然后72 ℃完全延伸10 min,扩增产物用1.0%的琼脂糖凝胶电泳进行检测。取10 μL扩增产物,置于质量分数8%的聚丙烯酰胺凝胶(丙烯酰胺∶甲叉双丙烯酰胺体积比37.5∶1)(变性剂范围35%~52%)中进行检测,电泳条件为:0.5×TAE缓冲溶液,恒温60 ℃,120 V运行78 min后80 V维持13 h。电泳结束后将银染法染色的凝胶扫描成像,挑选优势条带切胶回收,然后使用不带GC夹子的引物进行扩增,清洁,连接pMD18-T载体并转化大肠杆菌Top10,挑选2 株单菌落进行阳性克隆鉴定,并将菌液送至测序公司测序[28]。序列置于NCBI数据库进行同源性比对。
1.3.3 细菌MiSeq高通量测序分析
参照沈馨等[29]的方法进行PCR扩增。20 μL体系:5×FastPfu Buffer 4 μL、dNTP mix(2.5 mmol/L)2 μL、引物338F(5 μmol/L)和806R(5 μmol/L)各0.8 μL、rTaq(5 U/μL)0.4 μL、DNA模板10 ng,剩余体积用无菌水补齐;反应参数为:95 ℃、3 min;95 ℃、30 s;55 ℃、30 s;72 ℃、45 s;30 次循环;72 ℃、10 min。扩增合格的产物寄往上海美吉生物医药科技有限公司测序。序列下机后需进行拼接和质量控制,参照郭壮等[30]的方法删除不合格序列后,再根据各序列标签将序列划分至各样本中。利用QIIME数据分析平台[31]划分操作分类单元(operational taxonomic units,OTU),再从每个OTU中挑选代表序列,于Greengenes[32]和RDP(Ribosomal Database Project)[33]数据库中进行比对,确定其微生物分类水平,并计算各样品α多样性。
DGGE条带序列使用BioEdit 7.0.9软件(美国Tom Hall公司)进行拼接和引物切除,然后将处理好的序列置于NCBI数据库(https://blast.ncbi.nlm.nih.gov/Blast.cgi)进行比对;使用OriginPro 2017软件(美国OriginLab公司)绘图。

图1 腊肉样品中细菌的DGGE分析结果
Fig.1 DGGE fingerprint of bacteria in Chinese bacon samples
由图1可知,经DGGE后不同样品泳道中出现数量、亮度不一的条带,从中共挑选出9 条优势条带,其中条带7、8为所有样品的共有条带,条带1、2、3、5为部分样品共有条带,而条带4、6分别为样品LR2和LR3的特有条带。
表1 腊肉样品细菌DGGE优势条带的比对分析结果
Table 1 Sequence alignment of domain DGGE bands of Chinese bacon samples
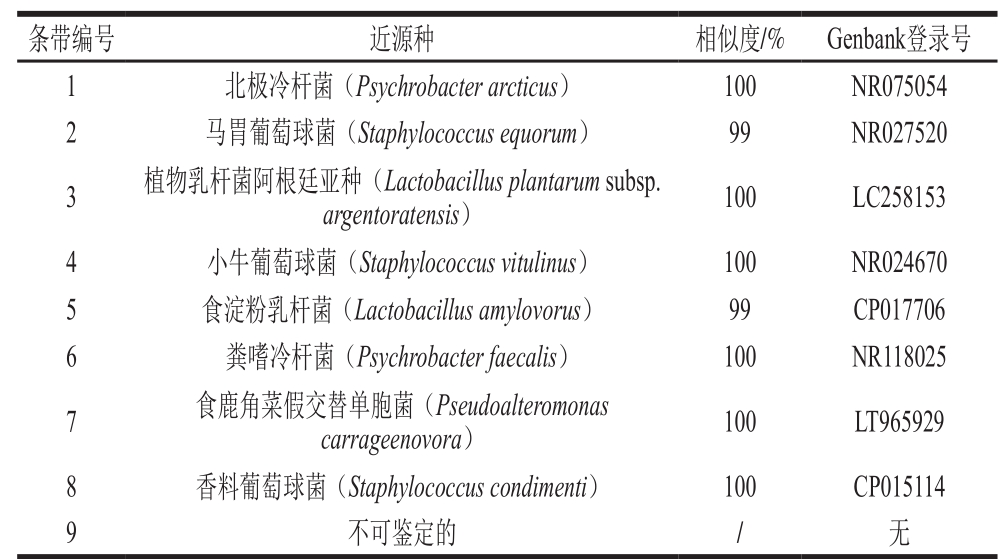
由表1可知,各优势条带与其近源种的相似度均在99%以上。经比对,条带2、4和8分别为马胃葡萄球菌(Staphylococcus equorum)、小牛葡萄球菌(Staphylococcus vitulinus)和香料葡萄球菌(Staphylococcus condimenti),均隶属于葡萄球菌属(Staphylococcus);条带1和6分别为北极冷杆菌(Psychrobacter arcticus)和粪嗜冷杆菌(Psychrobacter faecalis),均隶属于嗜冷杆菌属(Psychrobacter);条带3和5分别为植物乳杆菌阿根廷亚种(Lactobacillus plantarum subsp. argentoratensis)和食淀粉乳杆菌(Lactobacillus amylovorus),均隶属于乳酸杆菌属(Lactobacillus);条带7为食鹿角菜假交替单胞菌(Pseudoalteromonas carrageenovora),隶属于假交替单胞菌属(Pseudoalteromonas)。结合图1分析可知,腊肉中的细菌主要为葡萄球菌属(Staphylococcus)、嗜冷杆菌属(Psychrobacter)、乳酸杆菌属(Lactobacillus)和假交替单胞菌属(Pseudoalteromonas),且样品中均含有香料葡萄球菌(Staphylococcus condimenti)和食鹿角菜假交替单胞菌(Pseudoalteromonas carrageenovora),而样品LR2和LR3中的特有细菌分别为小牛葡萄球菌(Staphylococcus vitulinus)和粪嗜冷杆菌(Psychrobacter faecalis)。全拓等[34]的研究结果与本研究相同,他们通过采用纯培养的方法对川渝两地腊肉的优势菌多样性进行解析发现,葡萄球菌是腊肉产品货架期内的优势菌。
采用MiSeq高通量测序技术从5 个样品中检测到的序列在100%相似度下可划分为103 157 条,在97%的相似度下又可将这些序列划分至8 876 个OTU中,从各OTU中挑选代表性序列,与数据库进行比对可判定其微生物学分类定位。
表2 样品序列微生物分类地位统计
Table 2 Number of identifiable units at different taxonomical levels

注:*. 均在测序量为33 210 条序列时计算得到。
由表2可知,不同样本中检测到的细菌条带数量有所差异。经比对,样品中各微生物分类水平数量亦不相同,样品LR4中条带数量和OTU最多,而样品LR3中微生物的门、纲、目、科、属各级数量均较其他样品多。采用α多样性分析发现,在条带数量为33 210 条时,超Ⅰ指数和香农指数最大的为样品LR4,最小的为LR1,说明样品LR4中细菌总数及丰富度最高,而LR1中最低,这与DGGE分析结果一致。进一步在门水平分析样品中的细菌信息。
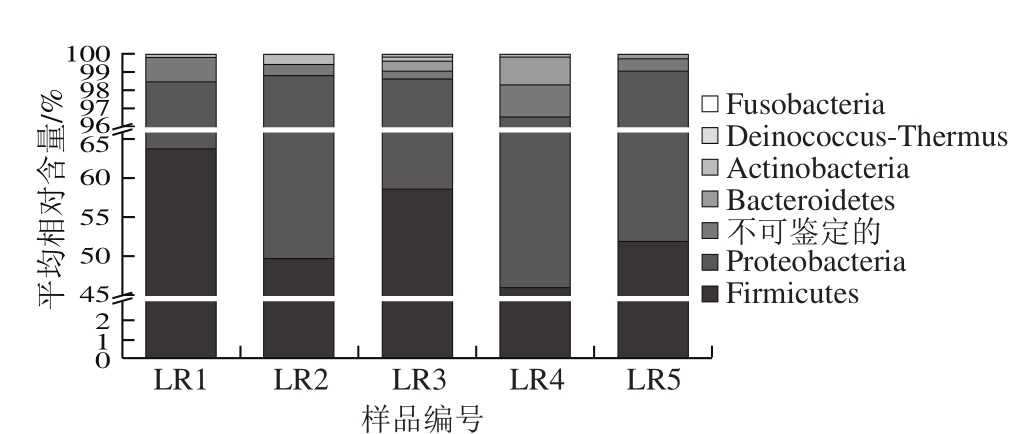
图2 腊肉样品细菌门水平的平均相对含量分析
Fig.2 Comparative analysis of the average relative contents of bacterial phyla in Chinese bacon samples
由图2可知,从5 个腊肉样品中检测出硬壁菌门(Firmicutes)、放线菌门(Actinobacteria)、拟杆菌门(Bacteroidetes)、异常球菌-栖热菌门(Deinococcus-Thermus)、梭杆菌门(Fusobacteria)和变形菌门(Proteobacteria)6 个细菌门,其中平均相对含量大于1.0%的为硬壁菌门(Firmicutes)和变形菌门(Proteobacteria),且二者的累积平均相对含量高达99.11%。异常球菌-栖热菌门为样品LR3的特有细菌门,平均相对含量仅为0.60%;梭杆菌门为样品LR4中的特有细菌门,平均相对含量仅为0.41%。
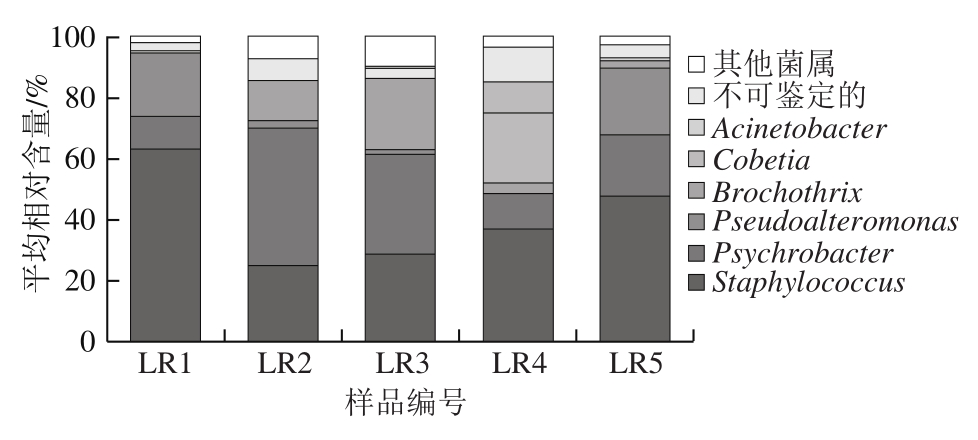
图3 腊肉样品中优势细菌属的平均相对含量分析
Fig.3 Comparative analysis of the average relative contents of dominant bacterial genera in Chinese bacon samples
进一步对99 个细菌属中平均相对含量大于1.0%的细菌属进行分析。由图3可知,腊肉中平均相对含量大于1.0%的细菌属分别为葡萄球菌属(Staphylococcus,40.18%)、嗜冷杆菌属(Psychrobacter,24.02%)、假交替单胞菌属(Pseudoalteromonas,9.37%)、环丝菌属(Brochothrix,8.53%)、科贝特氏菌属(Cobetia,4.71%)和不动细菌属(Acinetobacter,2.31%),仅有5.54%的序列不能鉴定到属水平。采用MiSeq高通量测序技术亦检测出乳酸菌属(Lactobacillus),其平均相对含量为0.27%,但PCR-DGGE未检测出隶属于科贝特氏菌属(Cobetia)和不动细菌属(Acinetobacter)等菌属的细菌,这可能与其扩增所用引物或这些菌属的丰度等因素有关。
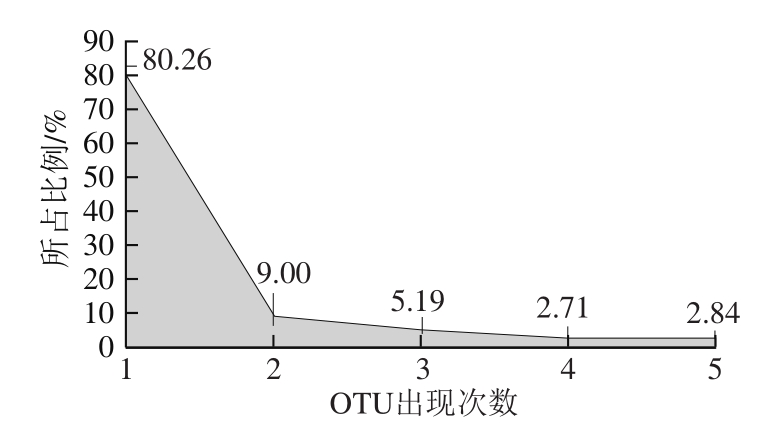
图4 腊肉样品中OTU出现次数的统计分析
Fig.4 Distribution of OTU as a function of their prevalence in Chinese bacon samples
表2表明不同样品中OTU数量亦不相同。由图4可知:仅出现1 次的OTU,即样品中特有OTU占总OTU数的80.26%,其所包含的序列有16 520 条,占总序列数的7.36%;出现5 次的OTU,即5 个样品中共有的OTU仅占OTU总数的2.84%,但其所包含的序列有166 546 条,占OTU总数的74.22%,说明腊肉样品细菌菌群中部分为某些样品中特有的,但数量较少,含量最多的主要为共有菌群。
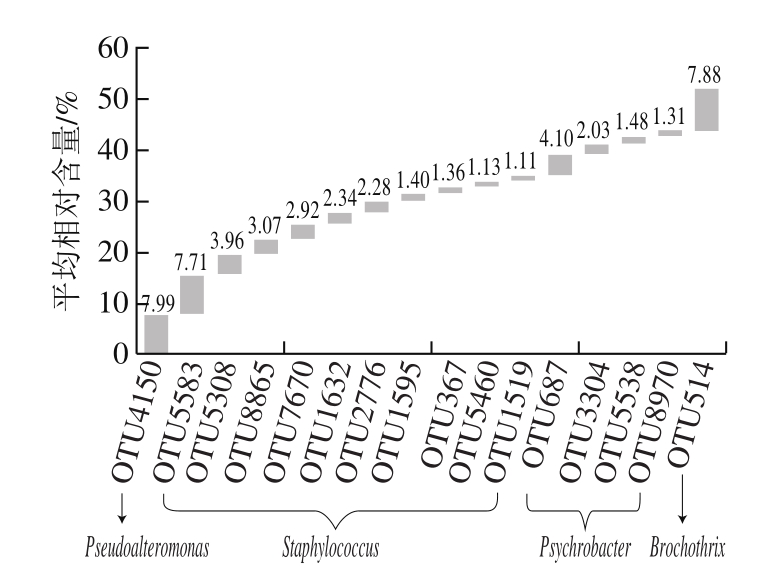
图5 腊肉样品中核心优势OTU的相对含量分析
Fig.5 Comparative analysis of the relative contents of the dominant core bacterial OTUs
将样品中共有且相对含量大于1.0%的OTU定义为核心优势OTU。由图5可知,腊肉中核心优势OTU有16个,其中OTU5583、OTU5308、OTU8865、OTU7670、OTU1632、OTU2776、OTU1595、OTU367、OTU5460和OTU1519隶属于葡萄球菌属(Staphylococcus),OTU687、OTU3304、OTU5538和OTU8970隶属于嗜冷杆菌属(Psychrobacter),OTU4150和OTU514分别隶属于和环丝菌属(Brochothrix)。由此可见,腊肉样品中含有大量核心优势细菌菌群,且主要为葡萄球菌属(Staphylococcus)、嗜冷杆菌属(Psychrobacter)、假交替单胞菌属(Pseudoalteromonas)和环丝菌属(Brochothrix)。
采用PCR-DGGE和MiSeq高通量测序技术相结合的方法对恩施地区腊肉细菌多样性进行解析。PCR-DGGE结果表明,腊肉中的细菌主要为葡萄球菌属(Staphylococcus)、嗜冷杆菌属(Psychrobacter)、乳酸杆菌属(Lactobacillus)和假交替单胞菌属(Pseudoalteromonas),且样品中均含有香料葡萄球菌(Staphylococcus condimenti)和食鹿角菜假交替单胞菌(Pseudoalteromonas carrageenovora);MiSeq高通量测序技术结果表明,腊肉中平均相对含量大于1.0%的细菌属为葡萄球菌属(Staphylococcus)、嗜冷杆菌属(Psychrobacter)、假交替单胞菌属(Pseudoalteromonas)、环丝菌属(Brochothrix)、科贝特氏菌属(Cobetia)和不动细菌属(Acinetobacter)。由此可见,2 种技术均表明恩施地区腊肉中的优势细菌为葡萄球菌属(Staphylococcus)、嗜冷杆菌属(Psychrobacter)和假交替单胞菌属(Pseudoalteromonas)。
参考文献:
[1] 张平, 杨勇, 曹春廷, 等. 食盐用量对四川腊肉加工及贮藏过程中肌肉蛋白质降解的影响[J]. 食品科学, 2014, 35(23): 67-72.DOI:10.7506/spkx1002-6630-201423014.
[2] 王电, 周国君, 王晓静, 等. 3 种贵州烟熏腊肉品质特征分析[J]. 肉类研究, 2015, 29(11): 1-6. DOI:10.15922/j.cnki.rlyj.2015.11.001.
[3] 刘洋, 王卫, 王新惠, 等. 微生物发酵剂对四川腊肉理化及微生物特性的影响[J]. 食品科技, 2014, 40(6): 124-129. DOI:10.13684/j.cnki.spkj.2014.06.028.
[4] 刘娜, 梁美莲, 谭媛元, 等. 天然涂膜液对切片腌腊肉品质及货架期的影响[J]. 肉类研究, 2017, 31(8): 12-17. DOI:10.7506/rlyj1001-8123-201708003.
[5] 刘晓蓉, 邓毛程, 连晓蔚. 腌肉中乳酸菌的分离选育[J]. 中国酿造,2009, 28(1): 46-48. DOI:10.3969/j.issn.0254-5071.2009.01.014.
[6] 刘书亮, 敖灵, 李燮昕, 等. 传统腌腊肉制品中乳酸菌的筛选与鉴定[J]. 食品与机械, 2007, 23(5): 19-21. DOI:10.13652/j.issn.1003-5788.2007.05.025.
[7] 陈竞适, 刘静, 任海姣, 等. 湘西陈年腊肉微生物群落分析及高产脂肪酶细菌的筛选[J]. 肉类研究, 2017, 31(3): 1-6. DOI:10.7506/rlyj1001-8123-201703001.
[8] 冯秀娟, 刘成国, 娄爱华, 等. 湖南腊肉中优势菌种的筛选及初步鉴定[J]. 中国酿造, 2012, 31(5): 127-130. DOI:10.3969/j.issn.0254-5071.2012.05.034.
[9] 王海燕, 马长伟, 李平兰, 等. 传统湖南腊肉中产香葡萄球菌的筛选及鉴定[J]. 食品与发酵工业, 2006, 32(5): 45-49. DOI:10.3321/j.issn:0253-990X.2006.05.012.
[10] 李福荣, 袁德峥, 宋淑红. 信阳传统发酵腊肉细菌的分离纯化及鉴定[J]. 信阳师范学院学报(自然科学版), 2009, 22(4): 590-592.DOI:10.3969/j.issn.1003-0972.2009.04.030.
[11] HOWITT S H, BLACKSHAW D, FONTAINE E, et al. Comparison of traditional microbiological culture and 16S polymerase chain reaction analyses for identification of preoperative airway colonization for patients undergoing lung resection[J]. Journal of Critical Care, 2018,46(8): 84-87. DOI:10.1016/j.jcrc.2018.04.013.
[12] GAROFALO C, BANCALARI E, MILANOVIĆ V, et al. Study of the bacterial diversity of foods: PCR-DGGE versus LH-PCR[J].International Journal of Food Microbiology, 2017, 242(2): 24-36.DOI:10.1016/j.ijfoodmicro.2016.11.008.
[13] ZHAO Fan, ZHOU Guanghong, YE Keping, et al. Microbial changes in vacuum-packed chilled pork during storage[J]. Meat Science, 2015,100(2): 145-149. DOI:10.1016/j.meatsci.2014.10.004.
[14] YE Keping, LIU Mei, LIU Jia, et al. Microbial diversity of different modified atmosphere packed pot-stewed duck wings products during 8 ℃ storage[J]. Letters in Applied Microbiology, 2017, 64(3):225-230. DOI:10.1111/lam.12704.
[15] YE Keping, JIANG Jian, WANG Yuefan, et al. Microbial analysis of MAP pot-stewed duck wings under different conditions during 15 ℃ storage[J]. Journal of Food Science and Technology, 2017,54(5): 1073-1079. DOI:10.1007/s13197.
[16] LIU Aiping, PENG Zhen, ZOU Likou, et al. The effects of lactic acid-based spray washing on bacterial profile and quality of chicken carcasses[J]. Food Control, 2016, 60(2): 615-620. DOI:10.1016/j.foodcont.2015.09.015.
[17] LI Peijun, LUO Huiting, KONG Baohua, et al. Formation of red myoglobin derivatives and inhibition of spoilage bacteria in raw meat batters by lactic acid bacteria and Staphylococcus xylosus[J]. LWTFood Science and Technology, 2016, 68(5): 251-257. DOI:10.1016/j.lwt.2015.12.035.
[18] DE SMIDT O. The use of PCR-DGGE to determine bacterial fingerprints for poultry and red meat abattoir effluent[J]. Letters in Applied Microbiology, 2016, 62(1): 1-8. DOI:10.1016/j.foodcont.2015.09.015.
[19] CAPORASO J G, LAUBER C L, WALTERS W A, et al. Ultra-highthroughput microbial community analysis on the Illumina HiSeq and MiSeq platforms[J]. The ISME Journal, 2012, 6(8): 1621-1624.DOI:10.1038/ismej.2012.8.
[20] LABRIE S J, EL HADDAD L, TREMBLAY D M, et al. First complete genome sequence of Staphylococcus xylosus, a meat starter culture and a host to propagate Staphylococcus aureus phages[J].Genome Announcements, 2014, 2(4): e00671-14. DOI:10.1128/genomeA.00671-14.
[21] MOURA Q, FERNANDES M R, CERDEIRA L, et al. Draft genome sequence of a multidrug-resistant CMY-2-producing Salmonella enterica subsp. entericaserovar Minnesota ST3088 isolated from chicken meat[J]. Journal of Global Antimicrobial Resistance, 2017,8(3): 67-69. DOI:10.1016/j.jgar.2016.10.011.
[22] LEE H S, KWON M, HEO S, et al. Characterization of the biodiversity of the spoilage microbiota in chicken meat using next generation sequencing and culture dependent approach[J]. Korean Journal for Food Science of Animal Resources, 2017, 37(4): 535-541.DOI:10.5851/kosfa.2017.37.4.535.
[23] PIOTROWSKA-CYPLIK A, MYSZKA K, CZARNY J, et al.Characterization of specific spoilage organisms (SSOs) in vacuumpacked ham by culture-plating techniques and MiSeq next-generation sequencing technologies[J]. Journal of the Science of Food and Agriculture, 2017, 97(2): 659-668. DOI:10.1002/jsfa.7785.
[24] ZHU Yingying, LIN Xisha, ZHAO Fan, et al. Meat, dairy and plant proteins alter bacterial composition of rat gut bacteria[J]. Scientific Reports, 2015, 5: 15220. DOI:10.1038/srep15220.
[25] KOO O K, KIM H J, BAKER C A, et al. Microbial diversity of ground beef products in South Korean retail market analyzed by PCR-DGGE and 454 pyrosequencing[J]. Food Biotechnology, 2016, 30(1): 63-77.DOI:10.1080/08905436.2015.1137216.
[26] 中国疾病预防控制中心营养与食品安全所. 食品卫生微生物检验肉与肉制品检验: GB/T 4789.17—2003[S]. 北京: 中国标准出版社, 2003.
[27] 丛敏, 李欣蔚, 武俊瑞, 等. PCR-DGGE分析东北传统发酵酸菜中乳酸菌多样性[J]. 食品科学, 2016, 37(7): 78-82. DOI:10.7506/spkx1002-6630-201607015.
[28] 郑艺, 张家超, 乔健敏, 等. 壮族人群肠道菌群多样性分析[J]. 中国食品学报, 2016, 16(1): 226-236. DOI:10.16429/j.1009-7848.2016.01.031.
[29] 沈馨, 尚雪娇, 董蕴, 等. 基于MiSeq高通量测序技术对3 个孝感凤窝酒曲细菌多样性的评价[J]. 中国微生态学杂志, 2018, 30(5): 525-530.DOI:10.13381/j.cnki.cjm.201805007.
[30] 郭壮, 蔡宏宇, 杨成聪, 等. 六名襄阳地区青年志愿者肠道菌群多样性的研究[J].中国微生态学杂志, 2017, 29(9): 998-1004.DOI:10.13381/j.cnki.cjm.201709002.
[31] CAPORASO J G, KUCZYNSKI J, STOMBAUGH J, et al. QIIME allows analysis of high-throughput community sequencing data[J].Nature Methods, 2010, 7(5): 335-336. DOI:10.1038/nmeth.f.303.
[32] DESANTIS T Z, HUGENHOLTZ P, LARSEN N, et al. Greengenes,a chimera-checked 16S rRNA gene database and workbench compatible with ARB[J]. Applied and Environmental Microbiology,2006, 72(7): 5069-5072. DOI:10.1128/AEM.03006-05.
[33] COLE J R, CHAI B, FARRIS R J, et al. The ribosomal database project (RDP-Ⅱ): introducing myRDP space and quality controlled public data[J]. Nucleic Acids Research, 2007, 35(1): 169-172.DOI:10.1093/nar/gkl889.
[34] 全拓, 邓大川, 李洪军, 等. 川味腊肉货架期间主要微生物的研究[J].西南大学学报(自然科学版), 2017, 39(2): 14-21. DOI:10.13718/j.cnki.xdzk.2017.02.003.
Evaluation of Bacterial Diversity in Chinese Bacon from Enshi by Denatured Gradient Gel Electrophoresis and MiSeq High-Throughput Sequencing
DONG Yun1, WANG Yurong1, WANG Yao1, LIAO Hua2, ZHAO Huijun1, GUO Zhuang1,3,*
(1.Northwest Hubei Research Institute of Traditional Fermented Food, College of Food Science and Technology, Hubei University of Arts and Science, Xiangyang 441053, China; 2.Enshi Agricultural Bureau, Enshi 445000, China;3.Enshi Public Inspection and Testing Center, Enshi 445000, China)
Abstract:The bacterial diversity in five Chinese bacon samples collected from Enshi, Hubei was analyzed by polymerase chain reaction-denaturing gradient gel electrophoresis (PCR-DGGE) and MiSeq high-throughput sequencing. The PCR-DGGE analysis indicated that the microbial flora in Chinese bacon was composed of Staphylococcus, Psychrobacter,Lactobacillus and Pseudoalteromonas. The results of MiSeq high-throughput sequencing indicated that Firmicutes and Proteobacteria were the dominate phyla, with a relative abundance of 54.05% and 44.28%, respectively. Meanwhile,Staphylococcus, Psychrobacter, Pseudoalteromonas, Brochothrix, Cobetia and Acinetobacter were the dominate genera,whose relative abundance was 40.18%, 24.02%, 9.37%, 8.53%, 4.71%, and 2.31%, respectively. A total of 252 operational taxonomic units (OTUs) were shared by all samples whose cumulative average was 74.22% of total qualified sequences.Thus, the dominant bacteria in Chinese bacon were Staphylococcus, Psychrobacter and Pseudoalteromonas.
Keywords:Chinese bacon; bacteria; denaturing gradient gel electrophoresis; MiSeq high-throughput sequencing
DONG Yun, WANG Yurong, WANG Yao, et al. Evaluation of bacterial diversity in Chinese bacon from Enshi by denatured gradient gel electrophoresis and Miseq high-throughput sequencing[J]. Meat Research, 2018, 32(10): 37-42. DOI:10.7506/rlyj1001-8123-201810007. http://www.rlyj.pub
DOI:10.7506/rlyj1001-8123-201810007. http://www.rlyj.pub
董蕴, 王玉荣, 王尧, 等. 基于变性梯度凝胶电泳和MiSeq高通量测序技术分析恩施地区腊肉的细菌多样性[J]. 肉类研究, 2018, 32(10): 37-42.
文章编号:1001-8123(2018)10-0037-06引文格式:
文献标志码:A
中图分类号:TS207.3
DOI:10.7506/rlyj1001-8123-201810007
*通信作者简介:郭壮(1984—),男,副教授,博士,研究方向为食品生物技术。E-mail:guozhuang1984@163.com
第一作者简介:董蕴(1997—),女,本科生,研究方向为食品生物技术。E-mail:1124222758@qq.com
基金项目:湖北省自然科学基金项目(2016CFB527);湖北文理学院食品新型工业化学科群建设项目(2018);湖北文理学院机电汽车学科群建设项目(2018);湖北文理学院食品科学技术学院大学生创新项目(2018)
收稿日期:2018-06-25